+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of PEIP-Bs_enolase complex | |||||||||
 マップデータ マップデータ | Cryo-EM structure of PEIP-Bs_enolase complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Enolase inhibitor / Glycolysis / Bacteriophage / ANTIMICROBIAL PROTEIN / LYASE-LYASE INHIBITOR complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報phosphopyruvate hydratase / phosphopyruvate hydratase complex / phosphopyruvate hydratase activity / sporulation resulting in formation of a cellular spore / glycolytic process / cell surface / magnesium ion binding / extracellular region 類似検索 - 分子機能 | |||||||||
| 生物種 |   Bacillus phage SP01 (ファージ) Bacillus phage SP01 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Li S / Zhang K | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2022 ジャーナル: Cell Rep / 年: 2022タイトル: Bacteriophage protein PEIP is a potent Bacillus subtilis enolase inhibitor. 著者: Kaining Zhang / Shanshan Li / Yawen Wang / Zhihao Wang / Nancy Mulvenna / Hang Yang / Peipei Zhang / Huan Chen / Yan Li / Hongliang Wang / Yongxiang Gao / Sivaramesh Wigneshweraraj / Steve ...著者: Kaining Zhang / Shanshan Li / Yawen Wang / Zhihao Wang / Nancy Mulvenna / Hang Yang / Peipei Zhang / Huan Chen / Yan Li / Hongliang Wang / Yongxiang Gao / Sivaramesh Wigneshweraraj / Steve Matthews / Kaiming Zhang / Bing Liu /   要旨: Enolase is a highly conserved enzyme that presents in all organisms capable of glycolysis or fermentation. Its immediate product phosphoenolpyruvate is essential for other important processes like ...Enolase is a highly conserved enzyme that presents in all organisms capable of glycolysis or fermentation. Its immediate product phosphoenolpyruvate is essential for other important processes like peptidoglycan synthesis and the phosphotransferase system in bacteria. Therefore, enolase inhibitors are of great interest. Here, we report that Gp60, a phage-encoded enolase inhibitor protein (PEIP) of bacteriophage SPO1 for Bacillus subtilis, is an enolase inhibitor. PEIP-expressing bacteria exhibit growth attenuation, thinner cell walls, and safranin color in Gram staining owing to impaired peptidoglycan synthesis. We solve the structure of PEIP-enolase tetramer and show that PEIP disassembles enolase by disrupting the basic dimer unit. The structure reveals that PEIP does not compete for substrate binding but induces a cascade of conformational changes that limit accessibility to the enolase catalytic site. This phage-inspired disassembly of enolase represents an alternative strategy for the development of anti-microbial drugs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33300.map.gz emd_33300.map.gz | 32.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33300-v30.xml emd-33300-v30.xml emd-33300.xml emd-33300.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33300.png emd_33300.png | 93.2 KB | ||
| Filedesc metadata |  emd-33300.cif.gz emd-33300.cif.gz | 5.4 KB | ||
| その他 |  emd_33300_half_map_1.map.gz emd_33300_half_map_1.map.gz emd_33300_half_map_2.map.gz emd_33300_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33300 http://ftp.pdbj.org/pub/emdb/structures/EMD-33300 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33300 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33300 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33300_validation.pdf.gz emd_33300_validation.pdf.gz | 769.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33300_full_validation.pdf.gz emd_33300_full_validation.pdf.gz | 768.7 KB | 表示 | |
| XML形式データ |  emd_33300_validation.xml.gz emd_33300_validation.xml.gz | 12.4 KB | 表示 | |
| CIF形式データ |  emd_33300_validation.cif.gz emd_33300_validation.cif.gz | 14.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33300 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33300 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33300 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33300 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7xmlMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33300.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33300.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of PEIP-Bs_enolase complex | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
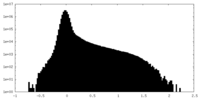
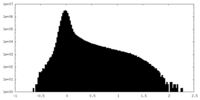
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map-2
| ファイル | emd_33300_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map-2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
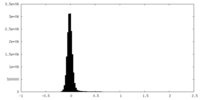
| 密度ヒストグラム |
-ハーフマップ: half map-1
| ファイル | emd_33300_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map-1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
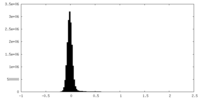
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PEIP-Bs_enolase complex
| 全体 | 名称: PEIP-Bs_enolase complex |
|---|---|
| 要素 |
|
-超分子 #1: PEIP-Bs_enolase complex
| 超分子 | 名称: PEIP-Bs_enolase complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 分子量 | 理論値: 100 KDa |
-超分子 #2: Bs_enolase
| 超分子 | 名称: Bs_enolase / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: PEIP
| 超分子 | 名称: PEIP / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Bacillus phage SP01 (ファージ) Bacillus phage SP01 (ファージ) |
-分子 #1: Enolase
| 分子 | 名称: Enolase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: phosphopyruvate hydratase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 46.626148 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPYIVDVYAR EVLDSRGNPT VEVEVYTETG AFGRALVPSG ASTGEYEAVE LRDGDKDRYL GKGVLTAVNN VNEIIAPELL GFDVTEQNA IDQLLIELDG TENKGKLGAN AILGVSMACA RAAADFLQIP LYQYLGGFNS KTLPVPMMNI VNGGEHADNN V DIQEFMIM ...文字列: MPYIVDVYAR EVLDSRGNPT VEVEVYTETG AFGRALVPSG ASTGEYEAVE LRDGDKDRYL GKGVLTAVNN VNEIIAPELL GFDVTEQNA IDQLLIELDG TENKGKLGAN AILGVSMACA RAAADFLQIP LYQYLGGFNS KTLPVPMMNI VNGGEHADNN V DIQEFMIM PVGAPNFREA LRMGAQIFHS LKSVLSAKGL NTAVGDEGGF APNLGSNEEA LQTIVEAIEK AGFKPGEEVK LA MDAASSE FYNKEDGKYH LSGEGVVKTS AEMVDWYEEL VSKYPIISIE DGLDENDWEG HKLLTERLGK KVQLVGDDLF VTN TKKLSE GIKNGVGNSI LIKVNQIGTL TETFDAIEMA KRAGYTAVIS HRSGETEDST IADIAVATNA GQIKTGAPSR TDRV AKYNQ LLRIEDQLAE TAQYHGINSF YNLNK UniProtKB: Enolase |
-分子 #2: Putative gene 60 protein
| 分子 | 名称: Putative gene 60 protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Bacillus phage SP01 (ファージ) Bacillus phage SP01 (ファージ) |
| 分子量 | 理論値: 8.558438 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLNQVEVLRE EYVEGYVVQM WRRNPSNAPV IEVFTEDNLE EGIIPEYVTA NDDTFDRIVD AVEFGYLEEL ELV UniProtKB: Putative gene 60 protein |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.3000000000000003 µm 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 553242 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)