+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human Cx36/GJD2 (N-terminal deletion mutant) gap junction channel in soybean lipids (D6 symmetry) | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Connexin 36 / Cx36 / Gap Junction Channel / GJD2 / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Electric Transmission Across Gap Junctions / connexin complex / Gap junction assembly / gap junction channel activity / neuronal action potential / visual perception / cell-cell signaling / chemical synaptic transmission / synapse / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Lee SN / Cho HJ / Jeong H / Ryu B / Lee HJ / Lee HH / Woo JS | |||||||||
| 資金援助 |  韓国, 2件 韓国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Cryo-EM structures of human Cx36/GJD2 neuronal gap junction channel. 著者: Seu-Na Lee / Hwa-Jin Cho / Hyeongseop Jeong / Bumhan Ryu / Hyuk-Joon Lee / Minsoo Kim / Jejoong Yoo / Jae-Sung Woo / Hyung Ho Lee /  要旨: Connexin 36 (Cx36) is responsible for signal transmission in electrical synapses by forming interneuronal gap junctions. Despite the critical role of Cx36 in normal brain function, the molecular ...Connexin 36 (Cx36) is responsible for signal transmission in electrical synapses by forming interneuronal gap junctions. Despite the critical role of Cx36 in normal brain function, the molecular architecture of the Cx36 gap junction channel (GJC) is unknown. Here, we determine cryo-electron microscopy structures of Cx36 GJC at 2.2-3.6 Å resolutions, revealing a dynamic equilibrium between its closed and open states. In the closed state, channel pores are obstructed by lipids, while N-terminal helices (NTHs) are excluded from the pore. In the open state with pore-lining NTHs, the pore is more acidic than those in Cx26 and Cx46/50 GJCs, explaining its strong cation selectivity. The conformational change during channel opening also includes the α-to-π-helix transition of the first transmembrane helix, which weakens the protomer-protomer interaction. Our structural analyses provide high resolution information on the conformational flexibility of Cx36 GJC and suggest a potential role of lipids in the channel gating. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33274.map.gz emd_33274.map.gz | 168 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33274-v30.xml emd-33274-v30.xml emd-33274.xml emd-33274.xml | 14.4 KB 14.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33274.png emd_33274.png | 129.5 KB | ||
| Filedesc metadata |  emd-33274.cif.gz emd-33274.cif.gz | 5.5 KB | ||
| その他 |  emd_33274_half_map_1.map.gz emd_33274_half_map_1.map.gz emd_33274_half_map_2.map.gz emd_33274_half_map_2.map.gz | 165.3 MB 165.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33274 http://ftp.pdbj.org/pub/emdb/structures/EMD-33274 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33274 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33274 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33274_validation.pdf.gz emd_33274_validation.pdf.gz | 994.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33274_full_validation.pdf.gz emd_33274_full_validation.pdf.gz | 994 KB | 表示 | |
| XML形式データ |  emd_33274_validation.xml.gz emd_33274_validation.xml.gz | 15.1 KB | 表示 | |
| CIF形式データ |  emd_33274_validation.cif.gz emd_33274_validation.cif.gz | 17.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33274 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33274 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33274 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33274 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7xl8MC  7xkiC  7xkkC  7xktC  7xnhC  7xnvC  8hkpC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33274.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33274.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8415 Å | ||||||||||||||||||||||||||||||||||||
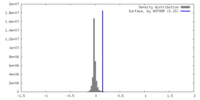
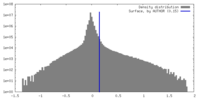
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map B
| ファイル | emd_33274_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
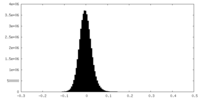
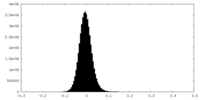
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_33274_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
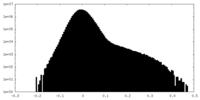
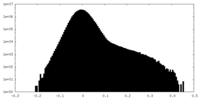
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cx36-delN8
| 全体 | 名称: Cx36-delN8 |
|---|---|
| 要素 |
|
-超分子 #1: Cx36-delN8
| 超分子 | 名称: Cx36-delN8 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Gap junction delta-2 protein
| 分子 | 名称: Gap junction delta-2 protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 36.584242 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MRLLEAAVQQ HSTMIGRILL TVVVIFRILI VAIVGETVYD DEQTMFVCNT LQPGCNQACY DRAFPISHIR YWVFQIIMVC TPSLCFITY SVHQSAKQRE RRYSTVFLAL DRDPPESIGG PGGTGGGGSG GGKREDKKLQ NAIVNGVLQN TENTSKETEP D CLEVKELT ...文字列: MRLLEAAVQQ HSTMIGRILL TVVVIFRILI VAIVGETVYD DEQTMFVCNT LQPGCNQACY DRAFPISHIR YWVFQIIMVC TPSLCFITY SVHQSAKQRE RRYSTVFLAL DRDPPESIGG PGGTGGGGSG GGKREDKKLQ NAIVNGVLQN TENTSKETEP D CLEVKELT PHPSGLRTAS KSKLRRQEGI SRFYIIQVVF RNALEIGFLV GQYFLYGFSV PGLYECNRYP CIKEVECYVS RP TEKTVFL VFMFAVSGIC VVLNLAELNH LGWRKIKLAV RGAQAKRKSI YEIRNKDLPR VSVPNFGRTQ SSDSAYVSRG DML EVLFQ UniProtKB: Gap junction delta-2 protein |
-分子 #2: 1,2-DIMYRISTOYL-RAC-GLYCERO-3-PHOSPHOCHOLINE
| 分子 | 名称: 1,2-DIMYRISTOYL-RAC-GLYCERO-3-PHOSPHOCHOLINE / タイプ: ligand / ID: 2 / コピー数: 84 / 式: MC3 |
|---|---|
| 分子量 | 理論値: 677.933 Da |
| Chemical component information | 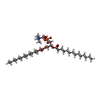 ChemComp-MC3: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 61.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.9 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 460806 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)