+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | neighboring ribosomes cluster1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 27.36 Å | |||||||||
 データ登録者 データ登録者 | Jiang W / Guo Q | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2022 ジャーナル: Nucleic Acids Res / 年: 2022タイトル: A transformation clustering algorithm and its application in polyribosomes structural profiling. 著者: Wenhong Jiang / Jonathan Wagner / Wenjing Du / Juergen Plitzko / Wolfgang Baumeister / Florian Beck / Qiang Guo /   要旨: Improvements in cryo-electron tomography sample preparation, electron-microscopy instrumentations, and image processing algorithms have advanced the structural analysis of macromolecules in situ. ...Improvements in cryo-electron tomography sample preparation, electron-microscopy instrumentations, and image processing algorithms have advanced the structural analysis of macromolecules in situ. Beyond such analyses of individual macromolecules, the study of their interactions with functionally related neighbors in crowded cellular habitats, i.e. 'molecular sociology', is of fundamental importance in biology. Here we present a NEighboring Molecule TOpology Clustering (NEMO-TOC) algorithm. We optimized this algorithm for the detection and profiling of polyribosomes, which play both constitutive and regulatory roles in gene expression. Our results suggest a model where polysomes are formed by connecting multiple nonstochastic blocks, in which translation is likely synchronized. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33074.map.gz emd_33074.map.gz | 17 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33074-v30.xml emd-33074-v30.xml emd-33074.xml emd-33074.xml | 11.7 KB 11.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33074.png emd_33074.png | 45.4 KB | ||
| マスクデータ |  emd_33074_msk_1.map emd_33074_msk_1.map | 22.2 MB |  マスクマップ マスクマップ | |
| その他 |  emd_33074_half_map_1.map.gz emd_33074_half_map_1.map.gz emd_33074_half_map_2.map.gz emd_33074_half_map_2.map.gz | 17 MB 17 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33074 http://ftp.pdbj.org/pub/emdb/structures/EMD-33074 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33074 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33074 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33074_validation.pdf.gz emd_33074_validation.pdf.gz | 608.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33074_full_validation.pdf.gz emd_33074_full_validation.pdf.gz | 607.6 KB | 表示 | |
| XML形式データ |  emd_33074_validation.xml.gz emd_33074_validation.xml.gz | 10.5 KB | 表示 | |
| CIF形式データ |  emd_33074_validation.cif.gz emd_33074_validation.cif.gz | 12.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33074 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33074 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33074 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33074 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33074.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33074.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 6.84 Å | ||||||||||||||||||||||||||||||||||||
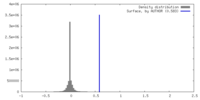
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_33074_msk_1.map emd_33074_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
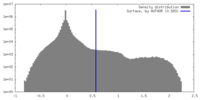
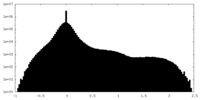
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33074_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
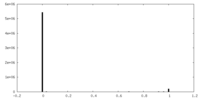
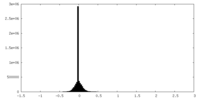
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33074_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
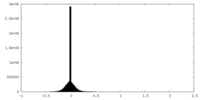
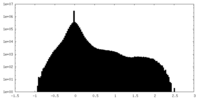
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : neighboring ribosomes cluter1
| 全体 | 名称: neighboring ribosomes cluter1 |
|---|---|
| 要素 |
|
-超分子 #1: neighboring ribosomes cluter1
| 超分子 | 名称: neighboring ribosomes cluter1 / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 1.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 6.0 µm / 最小 デフォーカス(公称値): 3.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 27.36 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用したサブトモグラム数: 3124 |
|---|---|
| 抽出 | トモグラム数: 18 / 使用した粒子像数: 11547 |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)