+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
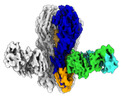
| タイトル | Cryo-EM structure of H1 hemagglutinin from A/Washington/05/2011 in complex with a neutralizing antibody 28-12 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Influenza A virus / H1N1 / antibody 12 fab / cryo-EM / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / host cell surface receptor binding / apical plasma membrane / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Influenza A virus (A型インフルエンザウイルス) / Influenza A virus (A型インフルエンザウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Cong Y / Liu CX | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Unique binding pattern for a lineage of human antibodies with broad reactivity against influenza A virus. 著者: Xiaoyu Sun / Caixuan Liu / Xiao Lu / Zhiyang Ling / Chunyan Yi / Zhen Zhang / Zi Li / Mingliang Jin / Wenshuai Wang / Shubing Tang / Fangfang Wang / Fang Wang / Sonam Wangmo / Shuangfeng Chen ...著者: Xiaoyu Sun / Caixuan Liu / Xiao Lu / Zhiyang Ling / Chunyan Yi / Zhen Zhang / Zi Li / Mingliang Jin / Wenshuai Wang / Shubing Tang / Fangfang Wang / Fang Wang / Sonam Wangmo / Shuangfeng Chen / Li Li / Liyan Ma / Yaguang Zhang / Zhuo Yang / Xiaoping Dong / Zhikang Qian / Jianping Ding / Dayan Wang / Yao Cong / Bing Sun /  要旨: Most structurally characterized broadly neutralizing antibodies (bnAbs) against influenza A viruses (IAVs) target the conserved conformational epitopes of hemagglutinin (HA). Here, we report a ...Most structurally characterized broadly neutralizing antibodies (bnAbs) against influenza A viruses (IAVs) target the conserved conformational epitopes of hemagglutinin (HA). Here, we report a lineage of naturally occurring human antibodies sharing the same germline gene, V3-48/V1-12. These antibodies broadly neutralize the major circulating strains of IAV in vitro and in vivo mainly by binding a contiguous epitope of H3N2 HA, but a conformational epitope of H1N1 HA, respectively. Our structural and functional studies of antibody 28-12 revealed that the continuous amino acids in helix A, particularly N49 of H3 HA, are critical to determine the binding feature with 28-12. In contrast, the conformational epitope feature is dependent on the discontinuous segments involving helix A, the fusion peptide, and several HA1 residues within H1N1 HA. We report that this antibody was initially selected by H3 (group 2) viruses and evolved via somatic hypermutation to enhance the reactivity to H3 and acquire cross-neutralization to H1 (group 1) virus. These findings enrich our understanding of different antigenic determinants of heterosubtypic influenza viruses for the recognition of bnAbs and provide a reference for the design of influenza vaccines and more effective antiviral drugs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33024.map.gz emd_33024.map.gz | 36.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33024-v30.xml emd-33024-v30.xml emd-33024.xml emd-33024.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33024.png emd_33024.png | 76.5 KB | ||
| Filedesc metadata |  emd-33024.cif.gz emd-33024.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33024 http://ftp.pdbj.org/pub/emdb/structures/EMD-33024 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33024 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33024 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33024_validation.pdf.gz emd_33024_validation.pdf.gz | 366.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33024_full_validation.pdf.gz emd_33024_full_validation.pdf.gz | 366 KB | 表示 | |
| XML形式データ |  emd_33024_validation.xml.gz emd_33024_validation.xml.gz | 5.8 KB | 表示 | |
| CIF形式データ |  emd_33024_validation.cif.gz emd_33024_validation.cif.gz | 6.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33024 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33024 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33024 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33024 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7x6oMC  7x6lC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33024.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33024.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.318 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : H1 hemagglutinin from A/Washington/05/2011 in complex with a neut...
| 全体 | 名称: H1 hemagglutinin from A/Washington/05/2011 in complex with a neutralizing antibody 28-12 |
|---|---|
| 要素 |
|
-超分子 #1: H1 hemagglutinin from A/Washington/05/2011 in complex with a neut...
| 超分子 | 名称: H1 hemagglutinin from A/Washington/05/2011 in complex with a neutralizing antibody 28-12 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
-分子 #1: Hemagglutinin
| 分子 | 名称: Hemagglutinin / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
| 分子量 | 理論値: 37.055742 KDa |
| 組換発現 | 生物種:  Insecta environmental sample (昆虫) Insecta environmental sample (昆虫) |
| 配列 | 文字列: TFATANSDTL CIGYHANNST DTVDTVLEKN VTVTHSVNLL EDKHNGKLCK LRGVAPLHLG KCNIAGWILG NPECESLSTA SSWSYIVET SSSDNGTCYP GDFIDYEELR EQLSSVSSFE RFEIFPKTSS WPNHDSNKGV TAACPHAGAK GFYKNLIWLV K KGNSYPKL ...文字列: TFATANSDTL CIGYHANNST DTVDTVLEKN VTVTHSVNLL EDKHNGKLCK LRGVAPLHLG KCNIAGWILG NPECESLSTA SSWSYIVET SSSDNGTCYP GDFIDYEELR EQLSSVSSFE RFEIFPKTSS WPNHDSNKGV TAACPHAGAK GFYKNLIWLV K KGNSYPKL SKSYINDKGK EVLVLWGIHH PSTTADQQSL YQNADTYVFV GTSRYSKKFK PEIAIRPKVR DQEGRMNYYW TL VEPGDKI TFEATGNLVV PRYAFAMERN AGSGIIISDT PVHDCNTTCQ TPKGAINTSL PFQNIHPITI GKCPKYVKST KLR LATGLR NVPSIQSR UniProtKB: Hemagglutinin |
-分子 #2: Hemagglutinin
| 分子 | 名称: Hemagglutinin / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス)株: A/Freiburg/728006/2011(H1N1) |
| 分子量 | 理論値: 20.160332 KDa |
| 組換発現 | 生物種:  Insecta environmental sample (昆虫) Insecta environmental sample (昆虫) |
| 配列 | 文字列: GLFGAIAGFI EGGWTGMVDG WYGYHHQNEQ GSGYAADLKS TQNAIDKITN KVNSVIEKMN TQFTAVGKEF NHLEKRIENL NKKVDDGFL DIWTYNAELL VLLENERTLD YHDSNVKNLY EKVRNQLKNN AKEIGNGCFE FYHKCDNTCM ESVKNGTYDY P KYSEEAKL NREEIDGV UniProtKB: Hemagglutinin |
-分子 #3: Heavy chain of antibody 12 fab
| 分子 | 名称: Heavy chain of antibody 12 fab / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.466277 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EVQLVESGGG LVQPGGSLRL SCAASGFTFS TYNMNWVRQA PGKGLEWLSY ISTSSNTIYY ADSVKGRFTI SRDNAKNSLF LQMNSLRDE DTAVYYCARD RGCSSTNCYV VGYYFYGMDV WGQGTTVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV K DYFPEPVT ...文字列: EVQLVESGGG LVQPGGSLRL SCAASGFTFS TYNMNWVRQA PGKGLEWLSY ISTSSNTIYY ADSVKGRFTI SRDNAKNSLF LQMNSLRDE DTAVYYCARD RGCSSTNCYV VGYYFYGMDV WGQGTTVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV K DYFPEPVT VSWNSGALTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKKV E |
-分子 #4: The light chain of antibody 12 fab
| 分子 | 名称: The light chain of antibody 12 fab / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.264861 KDa |
| 組換発現 | 生物種:  Insecta environmental sample (昆虫) Insecta environmental sample (昆虫) |
| 配列 | 文字列: DIQMTQSPSS VSASVGDRVT ITCRASQGIS SYLAWYQLKP GRAPKLLIYG ATRLQSGVPS RFSGSGSGTD FTLTISGLQP EDFATYHCQ QADSFPLTFG QGTRLEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: DIQMTQSPSS VSASVGDRVT ITCRASQGIS SYLAWYQLKP GRAPKLLIYG ATRLQSGVPS RFSGSGSGTD FTLTISGLQP EDFATYHCQ QADSFPLTFG QGTRLEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 38.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 124947 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)




















