+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3277 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structures of a CRISPR-Cas9 R-loop complex primed for DNA cleavage | |||||||||
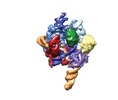
 マップデータ マップデータ | Reconstruction of Cas9 bound to sgRNA and 40-bp target DNA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR-Cas / Cas9 / genome editing | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / maintenance of CRISPR repeat elements / 3'-5' exonuclease activity / DNA endonuclease activity / endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / DNA binding / RNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptococcus pyogenes (化膿レンサ球菌) / lambda (unknown) Streptococcus pyogenes (化膿レンサ球菌) / lambda (unknown) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.0 Å | |||||||||
 データ登録者 データ登録者 | Jiang F / Taylor DW / Chen JS / Kornfeld JE / Zhou K / Thompson AJ / Nogales E / Doudna JA | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2016 ジャーナル: Science / 年: 2016タイトル: Structures of a CRISPR-Cas9 R-loop complex primed for DNA cleavage. 著者: Fuguo Jiang / David W Taylor / Janice S Chen / Jack E Kornfeld / Kaihong Zhou / Aubri J Thompson / Eva Nogales / Jennifer A Doudna /  要旨: Bacterial adaptive immunity and genome engineering involving the CRISPR (clustered regularly interspaced short palindromic repeats)-associated (Cas) protein Cas9 begin with RNA-guided DNA unwinding ...Bacterial adaptive immunity and genome engineering involving the CRISPR (clustered regularly interspaced short palindromic repeats)-associated (Cas) protein Cas9 begin with RNA-guided DNA unwinding to form an RNA-DNA hybrid and a displaced DNA strand inside the protein. The role of this R-loop structure in positioning each DNA strand for cleavage by the two Cas9 nuclease domains is unknown. We determine molecular structures of the catalytically active Streptococcus pyogenes Cas9 R-loop that show the displaced DNA strand located near the RuvC nuclease domain active site. These protein-DNA interactions, in turn, position the HNH nuclease domain adjacent to the target DNA strand cleavage site in a conformation essential for concerted DNA cutting. Cas9 bends the DNA helix by 30°, providing the structural distortion needed for R-loop formation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3277.map.gz emd_3277.map.gz | 84.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3277-v30.xml emd-3277-v30.xml emd-3277.xml emd-3277.xml | 11.8 KB 11.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3277.png emd_3277.png | 419.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3277 http://ftp.pdbj.org/pub/emdb/structures/EMD-3277 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3277 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3277 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3277_validation.pdf.gz emd_3277_validation.pdf.gz | 241.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3277_full_validation.pdf.gz emd_3277_full_validation.pdf.gz | 240.2 KB | 表示 | |
| XML形式データ |  emd_3277_validation.xml.gz emd_3277_validation.xml.gz | 6.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3277 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3277 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3277 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3277 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3277.map.gz / 形式: CCP4 / 大きさ: 89 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3277.map.gz / 形式: CCP4 / 大きさ: 89 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of Cas9 bound to sgRNA and 40-bp target DNA | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.01 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
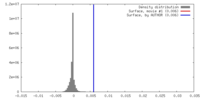
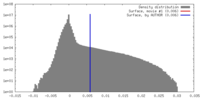
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cas9 bound to single guide-RNA and 40-bp target DNA
| 全体 | 名称: Cas9 bound to single guide-RNA and 40-bp target DNA |
|---|---|
| 要素 |
|
-超分子 #1000: Cas9 bound to single guide-RNA and 40-bp target DNA
| 超分子 | 名称: Cas9 bound to single guide-RNA and 40-bp target DNA / タイプ: sample / ID: 1000 / Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 218 KDa |
-分子 #1: CRISPR-associated endonuclease Cas9/Csn1
| 分子 | 名称: CRISPR-associated endonuclease Cas9/Csn1 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Cas9 / コピー数: 1 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) / 株: serotype M1 Streptococcus pyogenes (化膿レンサ球菌) / 株: serotype M1 |
| 分子量 | 理論値: 158 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: CRISPR-associated endonuclease Cas9/Csn1 GO: maintenance of CRISPR repeat elements, defense response to virus, GO: 0090305, DNA binding, RNA binding, endonuclease activity InterPro: CRISPR-associated endonuclease Cas9, CRISPR-associated endonuclease Cas9, bridge helix, CRISPR-associated endonuclease Cas9, PAM-interacting domain, CRISPR-associated endonuclease Cas9, REC ...InterPro: CRISPR-associated endonuclease Cas9, CRISPR-associated endonuclease Cas9, bridge helix, CRISPR-associated endonuclease Cas9, PAM-interacting domain, CRISPR-associated endonuclease Cas9, REC lobe, INTERPRO: IPR025978, HNH nuclease |
-分子 #2: single guide-RNA
| 分子 | 名称: single guide-RNA / タイプ: rna / ID: 2 / Name.synonym: sgRNA / 分類: OTHER / Structure: OTHER / Synthetic?: Yes |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) / 株: serotype M1 Streptococcus pyogenes (化膿レンサ球菌) / 株: serotype M1 |
| 分子量 | 理論値: 36 KDa |
| 配列 | 文字列: GGCGCAUAAA GAUGAGACGC GUUUUAGAGC UAUGCUGUUU UGAAAAAAAC AGCAUAGCAA GUUAAAAUAA GGCUAGUCCG UUAUCAACUU GAAAAAGUGG CACCGAGUCG GUGCUUCG |
-分子 #3: target 40-bp double stranded DNA
| 分子 | 名称: target 40-bp double stranded DNA / タイプ: dna / ID: 3 / Name.synonym: dsDNA 詳細: complementary strand nucleotide sequence: CGTGTTGATGCCGCGTATTTCTACTCTGCGACCGCTAATC 分類: DNA / Structure: DOUBLE HELIX / Synthetic?: Yes |
|---|---|
| 由来(天然) | 生物種: lambda (unknown) / 株: lambda 1 / 別称: lambda phage |
| 分子量 | 理論値: 24 KDa |
| 配列 | 文字列: GCACAACTAC GGCGCATAAA GATGAGACGC TGGCGATTAG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.25 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 30mM Tris 8.0, 150mM NaCl, 20mM EDTA, 5mM DTT and 0.1% glycerol |
| グリッド | 詳細: 4/2 C-flat grids with a thin-layer of carbon over the holes |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 100 K / 装置: FEI VITROBOT MARK IV 手法: Grids were rapidly plunged into liquid ethane using an FEI Vitrobot MarkIV maintained at 4 degrees C after being blotted for 4-4.5 seconds with a blotting force of 15-20. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective astigmatism was corrected at 210,000 times magnification |
| 詳細 | Data acquired using Leginon. We collected a 6 s exposure fractionated into 20, 300 ms frames with a dose of 8 electrons per square Angstrom per second. |
| 日付 | 2015年6月29日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 5600 / 平均電子線量: 48 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: CTFFind3 |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 6.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Relion / 使用した粒子像数: 50000 |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)