+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the barley Yellow stripe 1 transporter | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | iron-nicotianamine transmembrane transporter activity / Metal-nicotianamine transporter YSL-like / Oligopeptide transporter, OPT superfamily / OPT oligopeptide transporter protein / oligopeptide transmembrane transporter activity / seed development / response to iron ion / plasma membrane / Iron-phytosiderophore transporter 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
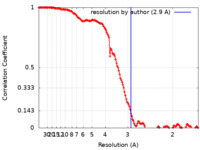
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Yamagata A | |||||||||
| 資金援助 |  日本, 1件 日本, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Uptake mechanism of iron-phytosiderophore from the soil based on the structure of yellow stripe transporter. 著者: Atsushi Yamagata / Yoshiko Murata / Kosuke Namba / Tohru Terada / Shuya Fukai / Mikako Shirouzu /  要旨: Calcareous soils cover one-third of all land and cause severe growth defects in plants due to the poor water solubility of iron at high pH. Poaceae species use a unique chelation strategy, whereby ...Calcareous soils cover one-third of all land and cause severe growth defects in plants due to the poor water solubility of iron at high pH. Poaceae species use a unique chelation strategy, whereby plants secrete a high-affinity metal chelator, known as phytosiderophores (mugineic acids), and reabsorb the iron-phytosiderophore complex by the yellow stripe 1/yellow stripe 1-like (YS1/YSL) transporter for efficient uptake of iron from the soil. Here, we present three cryo-electron microscopy structures of barley YS1 (HvYS1) in the apo state, in complex with an iron-phytosiderophore complex, Fe(III)-deoxymugineic acid (Fe(III)-DMA), and in complex with the iron-bound synthetic DMA analog (Fe(III)-PDMA). The structures reveal a homodimeric assembly mediated through an anti-parallel β-sheet interaction with cholesterol hemisuccinate. Each protomer adopts an outward open conformation, and Fe(III)-DMA is bound near the extracellular space in the central cavity. Fe(III)-PDMA occupies the same binding site as Fe(III)-DMA, demonstrating that PDMA can function as a potent fertilizer in an essentially identical manner to DMA. Our results provide a structural framework for iron-phytosiderophore recognition and transport by YS1/YSL transporters, which will enable the rational design of new, high-potency fertilizers. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32765.map.gz emd_32765.map.gz | 229.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32765-v30.xml emd-32765-v30.xml emd-32765.xml emd-32765.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32765_fsc.xml emd_32765_fsc.xml | 13.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32765.png emd_32765.png | 98.5 KB | ||
| マスクデータ |  emd_32765_msk_1.map emd_32765_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_32765_half_map_1.map.gz emd_32765_half_map_1.map.gz emd_32765_half_map_2.map.gz emd_32765_half_map_2.map.gz | 225.6 MB 225.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32765 http://ftp.pdbj.org/pub/emdb/structures/EMD-32765 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32765 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32765 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32765_validation.pdf.gz emd_32765_validation.pdf.gz | 661.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32765_full_validation.pdf.gz emd_32765_full_validation.pdf.gz | 660.9 KB | 表示 | |
| XML形式データ |  emd_32765_validation.xml.gz emd_32765_validation.xml.gz | 21.9 KB | 表示 | |
| CIF形式データ |  emd_32765_validation.cif.gz emd_32765_validation.cif.gz | 27.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32765 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32765 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32765 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32765 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32765.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32765.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.8285 Å | ||||||||||||||||||||
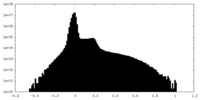
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_32765_msk_1.map emd_32765_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
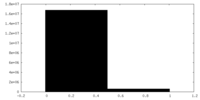
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_32765_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
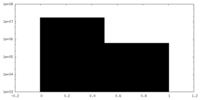
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_32765_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
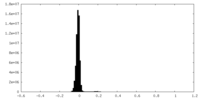
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Yellow stripe 1 in the apo state
| 全体 | 名称: Yellow stripe 1 in the apo state |
|---|---|
| 要素 |
|
-超分子 #1: Yellow stripe 1 in the apo state
| 超分子 | 名称: Yellow stripe 1 in the apo state / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Iron-phytosiderophore transporter
| 分子 | 名称: Iron-phytosiderophore transporter / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 76.070984 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDIVAPDRTR IAPEIDRDEA LEGDRESDPA LASTREWQLE DMPRWQDELT VRGLVAALLI GFIYTVIVMK IALTTGLVPT LNVSAALLS FLALRGWTRL LERFGVVSRP FTRQENTIVQ TCGVACYTIA FAGGFGSTLL GLNKKTYELA GDSPGNVPGS W KEPGIGWM ...文字列: MDIVAPDRTR IAPEIDRDEA LEGDRESDPA LASTREWQLE DMPRWQDELT VRGLVAALLI GFIYTVIVMK IALTTGLVPT LNVSAALLS FLALRGWTRL LERFGVVSRP FTRQENTIVQ TCGVACYTIA FAGGFGSTLL GLNKKTYELA GDSPGNVPGS W KEPGIGWM TGFLLACSFG GLLTLIPLRQ VLVVDYKLVY PSGTATAILI NGFHTDQGDK NSRKQIRGFL KYFGGSFLWS FF QWFYTGG DACGFVQFPT FGLKAWKQTF YFDFSMTYVG AGMICPHIVN ISTLLGAIIS WGIMWPLISK NKGDWYPAKV PES SMKSLY GYKAFICIAL IMGDGMYHFI KIVGITAMSM YRQFSHKQVN NKAKNADDTV SLEELHRQEI FKRGHIPSWM AYAG YALFS VLAVVTIPVM FKQVKWYYVV IAYVVAPMLG FANSYGTGLT DINMGYNYGK IALFVFAGWA GKENGVIAGL VAGTL VKQL VLISADLMQD FKTSYLTQTS PKSMMIAQVV GTAMGCIVSP LTFMLFYKAF DIGNPDGTWK APYALIYRNM AILGVE GFS VLPKYCIVIS GGFFAFAAIL SITRDVMPHK YAKYVPLPMA MAVPFLVGGS FAIDMCLGSL IVFAWTKINK KEAGFMV PA VASALICGDG IWTFPASILA LAKIKPPICM KFLPAATSAA HHHHHHHH |
-分子 #2: CHOLESTEROL HEMISUCCINATE
| 分子 | 名称: CHOLESTEROL HEMISUCCINATE / タイプ: ligand / ID: 2 / コピー数: 4 / 式: Y01 |
|---|---|
| 分子量 | 理論値: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X