+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Pseudomonas aeruginosa RNAP sigmaS holoenzyme complexes | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Pseudomonas aeruginosa / RNA polymerase / sigmaS / RNAP beta lobe / closed beta lobe / holoenzyme / transcription | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of response to salt stress / negative regulation of secondary metabolite biosynthetic process / positive regulation of single-species biofilm formation on inanimate substrate / regulation of cellular response to oxidative stress / positive regulation of chemotaxis / regulation of cell motility / sigma factor activity / regulation of cellular response to heat / DNA-directed RNA polymerase complex / DNA-templated transcription initiation ...regulation of response to salt stress / negative regulation of secondary metabolite biosynthetic process / positive regulation of single-species biofilm formation on inanimate substrate / regulation of cellular response to oxidative stress / positive regulation of chemotaxis / regulation of cell motility / sigma factor activity / regulation of cellular response to heat / DNA-directed RNA polymerase complex / DNA-templated transcription initiation / ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity / DNA-directed RNA polymerase / protein dimerization activity / DNA-templated transcription / regulation of DNA-templated transcription / magnesium ion binding / DNA binding / zinc ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) | |||||||||
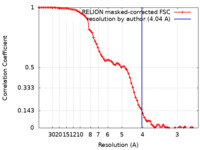
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.04 Å | |||||||||
 データ登録者 データ登録者 | He DW / You LL / Zhang Y | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Pseudomonas aeruginosa SutA wedges RNAP lobe domain open to facilitate promoter DNA unwinding. 著者: Dingwei He / Linlin You / Xiaoxian Wu / Jing Shi / Aijia Wen / Zhi Yan / Wenhui Mu / Chengli Fang / Yu Feng / Yu Zhang /  要旨: Pseudomonas aeruginosa (Pae) SutA adapts bacteria to hypoxia and nutrition-limited environment during chronic infection by increasing transcription activity of an RNA polymerase (RNAP) holoenzyme ...Pseudomonas aeruginosa (Pae) SutA adapts bacteria to hypoxia and nutrition-limited environment during chronic infection by increasing transcription activity of an RNA polymerase (RNAP) holoenzyme comprising the stress-responsive σ factor σ (RNAP-σ). SutA shows no homology to previously characterized RNAP-binding proteins. The structure and mode of action of SutA remain unclear. Here we determined cryo-EM structures of Pae RNAP-σ holoenzyme, Pae RNAP-σ holoenzyme complexed with SutA, and Pae RNAP-σ transcription initiation complex comprising SutA. The structures show SutA pinches RNAP-β protrusion and facilitates promoter unwinding by wedging RNAP-β lobe open. Our results demonstrate that SutA clears an energetic barrier to facilitate promoter unwinding of RNAP-σ holoenzyme. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31948.map.gz emd_31948.map.gz | 4.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31948-v30.xml emd-31948-v30.xml emd-31948.xml emd-31948.xml | 23 KB 23 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_31948_fsc.xml emd_31948_fsc.xml | 7.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_31948.png emd_31948.png | 173.7 KB | ||
| Filedesc metadata |  emd-31948.cif.gz emd-31948.cif.gz | 8.2 KB | ||
| その他 |  emd_31948_additional_1.map.gz emd_31948_additional_1.map.gz | 31.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31948 http://ftp.pdbj.org/pub/emdb/structures/EMD-31948 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31948 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31948 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31948_validation.pdf.gz emd_31948_validation.pdf.gz | 420.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31948_full_validation.pdf.gz emd_31948_full_validation.pdf.gz | 419.7 KB | 表示 | |
| XML形式データ |  emd_31948_validation.xml.gz emd_31948_validation.xml.gz | 10 KB | 表示 | |
| CIF形式データ |  emd_31948_validation.cif.gz emd_31948_validation.cif.gz | 13 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31948 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31948 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31948 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31948 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31948.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31948.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.307 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_31948_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Pseudomonas aeruginosa RNAP sigmaS holoenzyme complexes
| 全体 | 名称: Pseudomonas aeruginosa RNAP sigmaS holoenzyme complexes |
|---|---|
| 要素 |
|
-超分子 #1: Pseudomonas aeruginosa RNAP sigmaS holoenzyme complexes
| 超分子 | 名称: Pseudomonas aeruginosa RNAP sigmaS holoenzyme complexes タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) |
| 分子量 | 理論値: 432 KDa |
-分子 #1: DNA-directed RNA polymerase subunit alpha
| 分子 | 名称: DNA-directed RNA polymerase subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 |
| 分子量 | 理論値: 38.264258 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGHHHHHHHH HHMQSSVNEF LTPRHIDVQV VSQTRAKITL EPLERGFGHT LGNALRRILL SSMPGCAVVE AEIDGVLHEY SAIEGVQED VIEILLNLKG LAIKLHGRDE VTLTLAKKGS GVVTAADIQL DHDVEIINGD HVIANLADNG ALNMKLKVAR G RGYEPADA ...文字列: MGHHHHHHHH HHMQSSVNEF LTPRHIDVQV VSQTRAKITL EPLERGFGHT LGNALRRILL SSMPGCAVVE AEIDGVLHEY SAIEGVQED VIEILLNLKG LAIKLHGRDE VTLTLAKKGS GVVTAADIQL DHDVEIINGD HVIANLADNG ALNMKLKVAR G RGYEPADA RQSDEDESRS IGRLQLDASF SPVRRVSYVV ENARVEQRTN LDKLVLDLET NGTLDPEEAI RRAATILQQQ LA AFVDLKG DSEPVVEEQE DEIDPILLRP VDDLELTVRS ANCLKAENIY YIGDLIQRTE VELLKTPNLG KKSLTEIKDV LAS RGLSLG MRLDNWPPAS LKKDDKATA UniProtKB: DNA-directed RNA polymerase subunit alpha |
-分子 #2: DNA-directed RNA polymerase subunit beta
| 分子 | 名称: DNA-directed RNA polymerase subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 |
| 分子量 | 理論値: 151.225297 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGMAYSYTEK KRIRKDFSKL PDVMDVPYLL AIQLDSYREF LQAGATKEQF RDVGLHAAFK SVFPIISYSG NAALEYVGYR LGEPAFDVK ECVLRGVTFA VPLRVKVRLI IFDRESSNKA IKDIKEQEVY MGEIPLMTEN GTFIINGTER VIVSQLHRSP G VFFDHDRG ...文字列: MGMAYSYTEK KRIRKDFSKL PDVMDVPYLL AIQLDSYREF LQAGATKEQF RDVGLHAAFK SVFPIISYSG NAALEYVGYR LGEPAFDVK ECVLRGVTFA VPLRVKVRLI IFDRESSNKA IKDIKEQEVY MGEIPLMTEN GTFIINGTER VIVSQLHRSP G VFFDHDRG KTHSSGKLLY SARIIPYRGS WLDFEFDPKD CVFVRIDRRR KLPASVLLRA LGYSTEEILN AFYATNVFHI KG ETLNLEL VPQRLRGEVA SIDIKDGSGK VIVEQGRRIT ARHINQLEKA GVSQLEVPFD YLIGRTIAKA IVHPATGEII AEC NTELTL DLLAKVAKAQ VVRIETLYTN DIDCGPFISD TLKIDNTSNQ LEALVEIYRM MRPGEPPTKE AAETLFGNLF FSAE RYDLS AVGRMKFNRR IGRTEIEGPG VLSKEDIIDV LKTLVDIRNG KGIVDDIDHL GNRRVRCVGE MAENQFRVGL VRVER AVKE RLSMAESEGL MPQDLINAKP VAAAIKEFFG SSQLSQFMDQ NNPLSEITHK RRVSALGPGG LTRERAGFEV RDVHPT HYG RVCPIETPEG PNIGLINSLA TYARTNKYGF LESPYRVVKD SLVTDEIVFL SAIEEADHVI AQASATLNEK GQLVDEL VA VRHLNEFTVK APEDVTLMDV SPKQVVSVAA SLIPFLEHDD ANRALMGSNM QRQAVPTLRA DKPLVGTGME RNVARDSG V CVVARRGGVI DSVDASRVVV RVADDEVETG EAGVDIYNLT KYTRSNQNTC INQRPLVSKG DVVARGDILA DGPSTDMGE LALGQNMRVA FMPWNGFNFE DSICLSERVV QEDRFTTIHI QELTCVARDT KLGPEEITAD IPNVGEAALN KLDEAGIVYV GAEVQAGDI LVGKVTPKGE TQLTPEEKLL RAIFGEKASD VKDTSLRVPT GTKGTVIDVQ VFTRDGVERD SRALSIEKMQ L DQIRKDLN EEFRIVEGAT FERLRAALVG AKAEGGPALK KGTEITDDYL DGLERGQWFK LRMADDALNE QLEKAQAYIS DR RQLLDDK FEDKKRKLQQ GDDLAPGVLK IVKVYLAIKR RIQPGDKMAG RHGNKGVVSV IMPVEDMPHD ANGTPVDIVL NPL GVPSRM NVGQILETHL GLAAKGLGEK INRMLEEQRK VAELRKFLHE IYNEIGGREE NLDELGDNEI LALAKNLRGG VPMA TPVFD GAKEREIKAM LKLADLPESG QMRLFDGRTG NQFERPTTVG YMYMLKLNHL VDDKMHARST GSYSLVTQQP LGGKA QFGG QRFGEMEVWA LEAYGAAYTL QEMLTVKSDD VNGRTKMYKN IVDGDHRMEA GMPESFNVLI KEIRSLGIDI ELETE UniProtKB: DNA-directed RNA polymerase subunit beta |
-分子 #3: DNA-directed RNA polymerase subunit beta'
| 分子 | 名称: DNA-directed RNA polymerase subunit beta' / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 |
| 分子量 | 理論値: 156.038516 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLKDLLNLLK NQGQIEEFDA IRIGLASPEM IRSWSFGEVK KPETINYRTF KPERDGLFCA KIFGPVKDYE CLCGKYKRLK HRGVICEKC GVEVALAKVR RERMGHIELA SPVAHIWFLK SLPSRIGLLL DMTLRDIERV LYFESYVVID PGMTTLEKGQ L LNDEQYFE ...文字列: MLKDLLNLLK NQGQIEEFDA IRIGLASPEM IRSWSFGEVK KPETINYRTF KPERDGLFCA KIFGPVKDYE CLCGKYKRLK HRGVICEKC GVEVALAKVR RERMGHIELA SPVAHIWFLK SLPSRIGLLL DMTLRDIERV LYFESYVVID PGMTTLEKGQ L LNDEQYFE ALEEFGDDFD ARMGAEAVHE LLNAIDLEHE IGRLREEIPQ TNSETKIKKL SKRLKLMEAF QGSGNKPEWM VL TVLPVLP PDLRPLVPLD GGRFATSDLN DLYRRVINRN NRLKRLLDLA APDIIVRNEK RMLQEAVDAL LDNGRRGRAI TGS NKRPLK SLADMIKGKQ GRFRQNLLGK RVDYSGRSVI TVGPTLRLHQ CGLPKKMALE LFKPFIFGKL EGRGMATTIK AAKK MVERE LPEVWDVLAE VIREHPVLLN RAPTLHRLGI QAFEPVLIEG KAIQLHPLVC AAYNADFDGD QMAVHVPLTL EAQLE ARAL MMSTNNILSP ANGEPIIVPS QDVVMGLYYM TREAINAKGE GMAFADLQEV DRAYRSGQAS LHARVKVRIN EKIKGE DGQ LTANTRIVDT TVGRALLFQV VPAGLPFDVV NQSMKKKAIS KLINHCYRVV GLKDTVIFAD QLMYTGFAYS TISGVSI GV NDFVIPDEKA RIINAATDEV KEIESQYASG LVTQGEKYNK VIDLWSKAND EVSKAMMANL SKEKVVDREG KEVDQESF N SMYMMADSGA RGSAAQIRQL AGMRGLMAKP DGSIIETPIT ANFREGLNVL QYFISTHGAR KGLADTALKT ANSGYLTRR LVDVAQDLVV TEIDCGTEHG LLMSPHIEGG DVVEPLGERV LGRVIARDVF KPGSDEVIVP AGTLIDEKWV DFLEVMSVDE VVVRSPITC ETRHGICAMC YGRDLARGHR VNIGEAVGVI AAQSIGEPGT QLTMRTFHIG GAASRTSAAD NVQVKNGGTI R LHNLKHVV RADGALVAVS RSGELAVADD FGRERERYKL PYGAVISVKE GDKVDPGAIV AKWDPHTHPI VTEVDGTVAF VG MEEGITV KRQTDELTGL TNIEVMDPKD RPAAGKDIRP AVKLIDAAGK DLLLPGTDVP AQYFLPANAL VNLTDGAKVS IGD VVARIP QETSKTRDIT GGLPRVADLF EARRPKEPSI LAEISGTISF GKETKGKRRL VITPNDGSDP YEELIPKWRH LNVF EGEQV NRGEVISDGP SNPHDILRLL GVSSLAKYIV NEIQDVYRLQ GVKINDKHIE TILRQMLRKV EVSESGDSSF IKGDQ VELT QVLEENEQLG TEDKFPAKYE RVLLGITKAS LSTESFISAA SFQETTRVLT EAAVTGKRDF LRGLKENVVV GRLIPA GTG LAYHSERKRQ RDLGKPQRVS ASEAEAALTE ALNSSGNGSG SWSHPQFEK UniProtKB: DNA-directed RNA polymerase subunit beta' |
-分子 #4: DNA-directed RNA polymerase subunit omega
| 分子 | 名称: DNA-directed RNA polymerase subunit omega / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 |
| 分子量 | 理論値: 9.783876 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MARVTVEDCL DNVDNRFELV MLATKRARQL ATGGKEPKVA WENDKPTVVA LREIASGLVD ENVVQQEDIV EDEPLFAAFD DEANTEAL UniProtKB: DNA-directed RNA polymerase subunit omega |
-分子 #5: RNA polymerase sigma factor RpoS
| 分子 | 名称: RNA polymerase sigma factor RpoS / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 |
| 分子量 | 理論値: 38.606742 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GAMGMALKKE GPEFDHDDEV LLLEPGIMLD ESSADEQPSP RATPKATTSF SSKQHKHIDY TRALDATQLY LNEIGFSPLL TPEEEVHFA RLAQKGDPAG RKRMIESNLR LVVKIARRYV NRGLSLLDLI EEGNLGLIRA VEKFDPERGF RFSTYATWWI R QTIERAIM ...文字列: GAMGMALKKE GPEFDHDDEV LLLEPGIMLD ESSADEQPSP RATPKATTSF SSKQHKHIDY TRALDATQLY LNEIGFSPLL TPEEEVHFA RLAQKGDPAG RKRMIESNLR LVVKIARRYV NRGLSLLDLI EEGNLGLIRA VEKFDPERGF RFSTYATWWI R QTIERAIM NQTRTIRLPI HVVKELNVYL RAARELTHKL DHEPSPEEIA NLLEKPVAEV KRMLGLNERV TSVDVSLGPD SD KTLLDTL TDDRPTDPCE LLQDDDLSES IDQWLTELTD KQREVVIRRF GLRGHESSTL EEVGQEIGLT RERVRQIQVE ALK RLREIL EKNGLSSDAL FQ UniProtKB: RNA polymerase sigma factor RpoS |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #7: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 7 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 4 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV / 詳細: blot 8 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 58.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 29000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)