+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3183 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Localized reconstruction of Sec13/31 vertex from dodecahedral COPII cage | |||||||||
 マップデータ マップデータ | Localized reconstrucion of COPII sec13/31 vertex calculated from data published by Stagg et al. (2006) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | COPII / vertex / sec13 / sec31 | |||||||||
| 機能・相同性 | COPII vesicle coat / intracellular protein transport / WD40 repeat 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
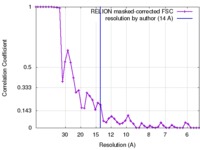
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 14.0 Å | |||||||||
 データ登録者 データ登録者 | Ilca S / Kotecha A / Sun X / Poranen MP / Stuart DI / Huiskonen JT | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2006 ジャーナル: Nature / 年: 2006タイトル: Structure of the Sec13/31 COPII coat cage. 著者: Scott M Stagg / Cemal Gürkan / Douglas M Fowler / Paul LaPointe / Ted R Foss / Clinton S Potter / Bridget Carragher / William E Balch /  要旨: Endomembranes of eukaryotic cells are dynamic structures that are in continuous communication through the activity of specialized cellular machineries, such as the coat protein complex II (COPII), ...Endomembranes of eukaryotic cells are dynamic structures that are in continuous communication through the activity of specialized cellular machineries, such as the coat protein complex II (COPII), which mediates cargo export from the endoplasmic reticulum (ER). COPII consists of the Sar1 GTPase, Sec23 and Sec24 (Sec23/24), where Sec23 is a Sar1-specific GTPase-activating protein and Sec24 functions in cargo selection, and Sec13 and Sec31 (Sec13/31), which has a structural role. Whereas recent results have shown that Sec23/24 and Sec13/31 can self-assemble to form COPII cage-like particles, we now show that Sec13/31 can self-assemble to form minimal cages in the absence of Sec23/24. We present a three-dimensional reconstruction of these Sec13/31 cages at 30 A resolution using cryo-electron microscopy and single particle analysis. These results reveal a novel cuboctahedron geometry with the potential to form a flexible lattice and to generate a diverse range of containers. Our data are consistent with a model for COPII coat complex assembly in which Sec23/24 has a non-structural role as a multivalent ligand localizing the self-assembly of Sec13/31 to form a cage lattice driving ER cargo export. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3183.map.gz emd_3183.map.gz | 5.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3183-v30.xml emd-3183-v30.xml emd-3183.xml emd-3183.xml | 12.9 KB 12.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3183_fsc.xml emd_3183_fsc.xml | 4.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  EMD-3183.jpg EMD-3183.jpg emd_3183.jpg emd_3183.jpg | 61.8 KB 61.8 KB | ||
| マスクデータ |  emd_3183_msk_1.map emd_3183_msk_1.map | 6.6 MB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3183 http://ftp.pdbj.org/pub/emdb/structures/EMD-3183 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3183 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3183 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3183_validation.pdf.gz emd_3183_validation.pdf.gz | 258.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3183_full_validation.pdf.gz emd_3183_full_validation.pdf.gz | 257.6 KB | 表示 | |
| XML形式データ |  emd_3183_validation.xml.gz emd_3183_validation.xml.gz | 7.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3183 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3183 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3183 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3183 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3183.map.gz / 形式: CCP4 / 大きさ: 6.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3183.map.gz / 形式: CCP4 / 大きさ: 6.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Localized reconstrucion of COPII sec13/31 vertex calculated from data published by Stagg et al. (2006) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.78 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
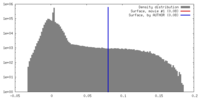
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-セグメンテーションマップ: Mask used for FSC
| 注釈 | Mask used for FSC | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_3183_msk_1.map emd_3183_msk_1.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
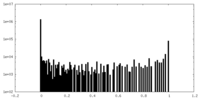
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Oligomeric assembly of Sec13-31
| 全体 | 名称: Oligomeric assembly of Sec13-31 |
|---|---|
| 要素 |
|
-超分子 #1000: Oligomeric assembly of Sec13-31
| 超分子 | 名称: Oligomeric assembly of Sec13-31 / タイプ: sample / ID: 1000 集合状態: 24-mer of Sec13-31 heterotetramers forming a cuboctahedron Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 8.14 MDa |
-分子 #1: Sec31
| 分子 | 名称: Sec31 / タイプ: protein_or_peptide / ID: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 実験値: 139 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) / 組換プラスミド: pFastBac Trichoplusia ni (イラクサキンウワバ) / 組換プラスミド: pFastBac |
| 配列 | GO: COPII vesicle coat / InterPro: WD40 repeat |
-分子 #2: Sec13
| 分子 | 名称: Sec13 / タイプ: protein_or_peptide / ID: 2 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 実験値: 33 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) / 組換プラスミド: pFastBac Trichoplusia ni (イラクサキンウワバ) / 組換プラスミド: pFastBac |
| 配列 | GO: intracellular protein transport / InterPro: WD40 repeat |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.90 mg/mL |
|---|---|
| 緩衝液 | pH: 6.5 / 詳細: 50mM MES pH 6.5, 225mM KOAc, 1mM MgCl2 |
| グリッド | 詳細: Quantifoil 2/2, 400 mesh copper grid |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: OTHER 詳細: Vitrification instrument: FEI Vitrobot. Grid plasma cleaned for 30s with Fischione 1020 plasma cleaner using 75-25 Argon-Oxygen mix. 手法: Temperature of chamber was 4 degrees C. 0 seconds drain time. Single blot. 0 mm offset. 4 ul sample applied to grid. Blot for 3.0 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 日付 | 2006年1月1日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: OTHER / 実像数: 810 / 平均電子線量: 20 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 50360 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 6.6 µm / 最小 デフォーカス(公称値): 3.4 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)