+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative-staining map of T4 DNA ligase-AMP-DNA Ternary Complex IV | |||||||||
 マップデータ マップデータ | T4 DNA ligase binding nick double-stranded DNA complex conformation Bent III | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ligase / Complex / DNA Repair / DNA replication / DNA BINDING PROTEIN | |||||||||
| 生物種 |  bacteriophage T (ウイルス) bacteriophage T (ウイルス) | |||||||||
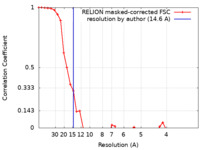
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 14.6 Å | |||||||||
 データ登録者 データ登録者 | Zhang L / Li N | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Repair of DNA nick by T4 DNA ligase in three parallel pathways 著者: Zhang L / Li N | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31749.map.gz emd_31749.map.gz | 3.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31749-v30.xml emd-31749-v30.xml emd-31749.xml emd-31749.xml | 11.5 KB 11.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_31749_fsc.xml emd_31749_fsc.xml | 3.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_31749.png emd_31749.png | 24.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31749 http://ftp.pdbj.org/pub/emdb/structures/EMD-31749 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31749 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31749 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31749_validation.pdf.gz emd_31749_validation.pdf.gz | 360.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31749_full_validation.pdf.gz emd_31749_full_validation.pdf.gz | 359.9 KB | 表示 | |
| XML形式データ |  emd_31749_validation.xml.gz emd_31749_validation.xml.gz | 7.4 KB | 表示 | |
| CIF形式データ |  emd_31749_validation.cif.gz emd_31749_validation.cif.gz | 9.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31749 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31749 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31749 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31749 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31749.map.gz / 形式: CCP4 / 大きさ: 3.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31749.map.gz / 形式: CCP4 / 大きさ: 3.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | T4 DNA ligase binding nick double-stranded DNA complex conformation Bent III | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.607 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : T4 DNA ligase binding nick double-stranded DNA complex conformati...
| 全体 | 名称: T4 DNA ligase binding nick double-stranded DNA complex conformation Bent III |
|---|---|
| 要素 |
|
-超分子 #1: T4 DNA ligase binding nick double-stranded DNA complex conformati...
| 超分子 | 名称: T4 DNA ligase binding nick double-stranded DNA complex conformation Bent III タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  bacteriophage T (ウイルス) bacteriophage T (ウイルス) |
| 分子量 | 理論値: 55 KDa |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.011 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Acetate 詳細: A 4.0 uL drop of the sample was placed on a glow-discharged EM specimen grid (CF200-Cu, Electron Microscopy Sciences, Hatfield, PA, USA) with a thin carbon film for 1 min and the excess ...詳細: A 4.0 uL drop of the sample was placed on a glow-discharged EM specimen grid (CF200-Cu, Electron Microscopy Sciences, Hatfield, PA, USA) with a thin carbon film for 1 min and the excess solution was blotted with filter paper. Then, the grid was washed with a clean drop (~35 uL/drop) of water and stained two times with two drops (~25 uL/drop) of 2% (w/v) uranyl acetate (UA) for 10 s, 30 s respectively before air-drying. | ||||||||||||||||||
| グリッド | モデル: C-flat / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 3 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.038 kPa | ||||||||||||||||||
| 詳細 | T4 DNA ligase and nicked double-stranded DNA substrate were mixed at a molar ratio of 1:1 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS TALOS F200C |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI CETA (4k x 4k) / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 実像数: 3765 / 平均露光時間: 1.0 sec. / 平均電子線量: 160.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)