+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-31197 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure and Activity of SLAC1 Channels for Stomatal Signaling in Leaves | ||||||||||||||||||
 マップデータ マップデータ | Map used for building. Post-processed from half maps using phenix.auto_sharpen. | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報response to humidity / stomatal closure / regulation of stomatal opening / voltage-gated monoatomic anion channel activity / regulation of stomatal closure / response to ozone / intracellular monoatomic ion homeostasis / organic anion transport / response to carbon dioxide / response to abscisic acid ...response to humidity / stomatal closure / regulation of stomatal opening / voltage-gated monoatomic anion channel activity / regulation of stomatal closure / response to ozone / intracellular monoatomic ion homeostasis / organic anion transport / response to carbon dioxide / response to abscisic acid / multicellular organismal-level water homeostasis / response to light stimulus / protein phosphatase binding / membrane => GO:0016020 / protein kinase binding / plasma membrane 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Brachypodium distachyon (セイヨウヤマカモジ) Brachypodium distachyon (セイヨウヤマカモジ) | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 2.97 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Deng Y / Kashtoh H / Wang Q / Zhen GX / Li QY / Tang L / Gao HL / Zhang CR / Qin L / Su M ...Deng Y / Kashtoh H / Wang Q / Zhen GX / Li QY / Tang L / Gao HL / Zhang CR / Qin L / Su M / Li F / Huang XH / Wang YC / Xie Q / Clarke OB / Hendrickson WA / Chen YH | ||||||||||||||||||
| 資金援助 |  中国, 中国,  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2021 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021タイトル: Structure and activity of SLAC1 channels for stomatal signaling in leaves. 著者: Ya-Nan Deng / Hamdy Kashtoh / Quan Wang / Guang-Xiao Zhen / Qi-Yu Li / Ling-Hui Tang / Hai-Long Gao / Chun-Rui Zhang / Li Qin / Min Su / Fei Li / Xia-He Huang / Ying-Chun Wang / Qi Xie / ...著者: Ya-Nan Deng / Hamdy Kashtoh / Quan Wang / Guang-Xiao Zhen / Qi-Yu Li / Ling-Hui Tang / Hai-Long Gao / Chun-Rui Zhang / Li Qin / Min Su / Fei Li / Xia-He Huang / Ying-Chun Wang / Qi Xie / Oliver B Clarke / Wayne A Hendrickson / Yu-Hang Chen /   要旨: Stomata in leaves regulate gas exchange between the plant and its atmosphere. Various environmental stimuli elicit abscisic acid (ABA); ABA leads to phosphoactivation of slow anion channel 1 (SLAC1); ...Stomata in leaves regulate gas exchange between the plant and its atmosphere. Various environmental stimuli elicit abscisic acid (ABA); ABA leads to phosphoactivation of slow anion channel 1 (SLAC1); SLAC1 activity reduces turgor pressure in aperture-defining guard cells; and stomatal closure ensues. We used electrophysiology for functional characterizations of SLAC1 (SLAC1) and cryoelectron microscopy (cryo-EM) for structural analysis of SLAC1 (SLAC1), at 2.97-Å resolution. We identified 14 phosphorylation sites in SLAC1 and showed nearly 330-fold channel-activity enhancement with 4 to 6 of these phosphorylated. Seven SLAC1-conserved arginines are poised in SLAC1 for regulatory interaction with the N-terminal extension. This SLAC1 structure has its pores closed, in a basal state, spring loaded by phenylalanyl residues in high-energy conformations. SLAC1 phosphorylation fine-tunes an equilibrium between basal and activated SLAC1 trimers, thereby controlling the degree of stomatal opening. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31197.map.gz emd_31197.map.gz | 27.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31197-v30.xml emd-31197-v30.xml emd-31197.xml emd-31197.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_31197_fsc.xml emd_31197_fsc.xml | 7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_31197.png emd_31197.png | 56.4 KB | ||
| マスクデータ |  emd_31197_msk_1.map emd_31197_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| その他 |  emd_31197_half_map_1.map.gz emd_31197_half_map_1.map.gz emd_31197_half_map_2.map.gz emd_31197_half_map_2.map.gz | 28.4 MB 28.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31197 http://ftp.pdbj.org/pub/emdb/structures/EMD-31197 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31197 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31197 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31197.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31197.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map used for building. Post-processed from half maps using phenix.auto_sharpen. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0475 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_31197_msk_1.map emd_31197_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
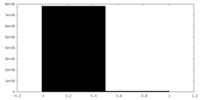
| 投影像・断面図 |
| ||||||||||||
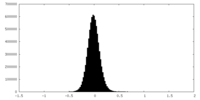
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_31197_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
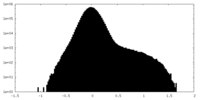
| 投影像・断面図 |
| ||||||||||||
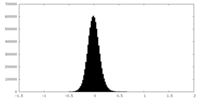
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_31197_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of Brachypodium distachyon SLAC1
| 全体 | 名称: Cryo-EM structure of Brachypodium distachyon SLAC1 |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of Brachypodium distachyon SLAC1
| 超分子 | 名称: Cryo-EM structure of Brachypodium distachyon SLAC1 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Brachypodium distachyon (セイヨウヤマカモジ) Brachypodium distachyon (セイヨウヤマカモジ) |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Acetate |
| グリッド | モデル: UltrAuFoil R0.6/1 / 材質: GOLD / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 71.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X