+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30668 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo_EM structure of delta N-NPC1L1-EZE | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報vitamin transport / cholesterol import / intestinal cholesterol absorption / lipid transporter activity / response to muscle activity / heterocyclic compound binding / cholesterol binding / cholesterol homeostasis / brush border membrane / response to xenobiotic stimulus / apical plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.58 Å | |||||||||
 データ登録者 データ登録者 | Hu M / Sun S | |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: Structural insights into the mechanism of human NPC1L1-mediated cholesterol uptake. 著者: Miaoqing Hu / Fan Yang / Yawen Huang / Xin You / Desheng Liu / Shan Sun / Sen-Fang Sui /  要旨: Niemann-Pick C1-like 1 (NPC1L1) protein plays a central role in the intestinal cholesterol absorption and is the target of a drug, ezetimibe, which inhibits NPC1L1 to reduce cholesterol absorption. ...Niemann-Pick C1-like 1 (NPC1L1) protein plays a central role in the intestinal cholesterol absorption and is the target of a drug, ezetimibe, which inhibits NPC1L1 to reduce cholesterol absorption. Here, we present cryo-electron microscopy structures of human NPC1L1 in apo state, cholesterol-enriched state, and ezetimibe-bound state to reveal molecular details of NPC1L1-mediated cholesterol uptake and ezetimibe inhibition. Comparison of these structures reveals that the sterol-sensing domain (SSD) could respond to the cholesterol level alteration by binding different number of cholesterol molecules. Upon increasing cholesterol level, SSD binds more cholesterol molecules, which, in turn, triggers the formation of a stable structural cluster in SSD, while binding of ezetimibe causes the deformation of the SSD and destroys the structural cluster, leading to the inhibition of NPC1L1 function. These results provide insights into mechanisms of NPC1L1 function and ezetimibe action and are of great significance for the development of new cholesterol absorption inhibitors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30668.map.gz emd_30668.map.gz | 36.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30668-v30.xml emd-30668-v30.xml emd-30668.xml emd-30668.xml | 10.7 KB 10.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_30668.png emd_30668.png | 110.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30668 http://ftp.pdbj.org/pub/emdb/structures/EMD-30668 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30668 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30668 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_30668_validation.pdf.gz emd_30668_validation.pdf.gz | 378.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_30668_full_validation.pdf.gz emd_30668_full_validation.pdf.gz | 377.8 KB | 表示 | |
| XML形式データ |  emd_30668_validation.xml.gz emd_30668_validation.xml.gz | 6.5 KB | 表示 | |
| CIF形式データ |  emd_30668_validation.cif.gz emd_30668_validation.cif.gz | 7.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30668 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30668 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30668 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30668 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30668.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30668.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.091 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : delta N-hNPC1L1-EZE
| 全体 | 名称: delta N-hNPC1L1-EZE |
|---|---|
| 要素 |
|
-超分子 #1: delta N-hNPC1L1-EZE
| 超分子 | 名称: delta N-hNPC1L1-EZE / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293 Homo sapiens (ヒト) / 組換細胞: HEK293 |
-分子 #1: NPC1-like intracellular cholesterol transporter 1
| 分子 | 名称: NPC1-like intracellular cholesterol transporter 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 139.612125 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAEAGLRGWL LWALLLRLAQ SEPYTTIHQP GYCAFYDECG KNPELSGSLM TLSNVSCLSN TPARKITGDH LILLQKICPR LYTGPNTQA CCSAKQLVSL EASLSITKAL LTRCPACSDN FVNLHCHNTC SPNQSLFINV TRVAQLGAGQ LPAVVAYEAF Y QHSFAEQS ...文字列: MAEAGLRGWL LWALLLRLAQ SEPYTTIHQP GYCAFYDECG KNPELSGSLM TLSNVSCLSN TPARKITGDH LILLQKICPR LYTGPNTQA CCSAKQLVSL EASLSITKAL LTRCPACSDN FVNLHCHNTC SPNQSLFINV TRVAQLGAGQ LPAVVAYEAF Y QHSFAEQS YDSCSRVRVP AAATLAVGTM CGVYGSALCN AQRWLNFQGD TGNGLAPLDI TFHLLEPGQA VGSGIQPLNE GV ARCNESQ GDDVATCSCQ DCAASCPAIA RPQALDSTFY LGQMPGSLVL IIILCSVFAV VTILLVGFRV APARDKSKMV DPK KGTSLS DKLSFSTHTL LGQFFQGWGT WVASWPLTIL VLSVIPVVAL AAGLVFTELT TDPVELWSAP NSQARSEKAF HDQH FGPFF RTNQVILTAP NRSSYRYDSL LLGPKNFSGI LDLDLLLELL ELQERLRHLQ VWSPEAQRNI SLQDICYAPL NPDNT SLYD CCINSLLQYF QNNRTLLLLT ANQTLMGQTS QVDWKDHFLY CANAPLTFKD GTALALSCMA DYGAPVFPFL AIGGYK GKD YSEAEALIMT FSLNNYPAGD PRLAQAKLWE EAFLEEMRAF QRRMAGMFQV TFMAERSLED EINRTTAEDL PIFATSY IV IFLYISLALG SYSSWSRVMV DSKATLGLGG VAVVLGAVMA AMGFFSYLGI RSSLVILQVV PFLVLSVGAD NIFIFVLE Y QRLPRRPGEP REVHIGRALG RVAPSMLLCS LSEAICFFLG ALTPMPAVRT FALTSGLAVI LDFLLQMSAF VALLSLDSK RQEASRLDVC CCVKPQELPP PGQGEGLLLG FFQKAYAPFL LHWITRGVVL LLFLALFGVS LYSMCHISVG LDQELALPKD SYLLDYFLF LNRYFEVGAP VYFVTTLGYN FSSEAGMNAI CSSAGCNNFS FTQKIQYATE FPEQSYLAIP ASSWVDDFID W LTPSSCCR LYISGPNKDK FCPSTVNSLN CLKNCMSITM GSVRPSVEQF HKYLPWFLND RPNIKCPKGG LAAYSTSVNL TS DGQVLAS RFMAYHKPLK NSQDYTEALR AARELAANIT ADLRKVPGTD PAFEVFPYTI TNVFYEQYLT ILPEGLFMLS LCL VPTFAV SCLLLGLDLR SGLLNLLSIV MILVDTVGFM ALWGISYNAV SLINLVSAVG MSVEFVSHIT RSFAISTKPT WLER AKEAT ISMGSAVFAG VAMTNLPGIL VLGLAKAQLI QIFFFRLNLL ITLLGLLHGL VFLPVILSYV GPDVNPAL |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 5 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #6: (3~{R},4~{S})-1-(4-fluorophenyl)-3-[(3~{S})-3-(4-fluorophenyl)-3-...
| 分子 | 名称: (3~{R},4~{S})-1-(4-fluorophenyl)-3-[(3~{S})-3-(4-fluorophenyl)-3-oxidanyl-propyl]-4-(4-hydroxyphenyl)azetidin-2-one タイプ: ligand / ID: 6 / コピー数: 1 / 式: H56 |
|---|---|
| 分子量 | 理論値: 409.425 Da |
| Chemical component information | 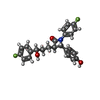 ChemComp-H56: |
-分子 #7: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 7 / コピー数: 4 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.58 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 2204129 |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー





















