| 登録情報 | データベース: PDB / ID: 2zin
|
|---|
| タイトル | Crystal structure of the catalytic domain of pyrrolysyl-tRNA synthetase in complex with BocLys and an ATP analogue |
|---|
 要素 要素 | Pyrrolysyl-tRNA synthetase |
|---|
 キーワード キーワード | LIGASE / aminoacyl-tRNA synthetase / pyrrolysyl-tRNA synthetase / pyrrolysine / tRNA / ATP analogue / non-natural amino acid / unnatural amino acid / ATP-binding / Cytoplasm / Nucleotide-binding / Protein biosynthesis / Structural Genomics / NPPSFA / National Project on Protein Structural and Functional Analyses / RIKEN Structural Genomics/Proteomics Initiative / RSGI |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
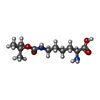
pyrrolysine-tRNAPyl ligase / pyrrolysyl-tRNA synthetase activity / tRNA aminoacylation for protein translation / tRNA binding / ATP binding / cytoplasm類似検索 - 分子機能 Pyrrolysyl-tRNA ligase / Pyrrolysyl-tRNA ligase, N-terminal / Helix hairpin bin / Pyrrolysyl-tRNA ligase, C-terminal / Phenylalanyl-tRNA synthetase / tRNA synthetases class II core domain (F) / Bira Bifunctional Protein; Domain 2 / BirA Bifunctional Protein; domain 2 / Aminoacyl-tRNA synthetase, class II / Aminoacyl-transfer RNA synthetases class-II family profile. ...Pyrrolysyl-tRNA ligase / Pyrrolysyl-tRNA ligase, N-terminal / Helix hairpin bin / Pyrrolysyl-tRNA ligase, C-terminal / Phenylalanyl-tRNA synthetase / tRNA synthetases class II core domain (F) / Bira Bifunctional Protein; Domain 2 / BirA Bifunctional Protein; domain 2 / Aminoacyl-tRNA synthetase, class II / Aminoacyl-transfer RNA synthetases class-II family profile. / Class II Aminoacyl-tRNA synthetase/Biotinyl protein ligase (BPL) and lipoyl protein ligase (LPL) / Helix Hairpins / 2-Layer Sandwich / Orthogonal Bundle / Mainly Alpha / Alpha Beta類似検索 - ドメイン・相同性 PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / N~6~-(tert-butoxycarbonyl)-L-lysine / Pyrrolysine--tRNA ligase類似検索 - 構成要素 |
|---|
| 生物種 |  Methanosarcina mazei (古細菌) Methanosarcina mazei (古細菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.79 Å 分子置換 / 解像度: 1.79 Å |
|---|
 データ登録者 データ登録者 | Yanagisawa, T. / Ishii, R. / Yokoyama, S. / RIKEN Structural Genomics/Proteomics Initiative (RSGI) |
|---|
 引用 引用 |  ジャーナル: Chem.Biol. / 年: 2008 ジャーナル: Chem.Biol. / 年: 2008
タイトル: Multistep Engineering of Pyrrolysyl-tRNA Synthetase to Genetically Encode N(varepsilon)-(o-Azidobenzyloxycarbonyl) lysine for Site-Specific Protein Modification
著者: Yanagisawa, T. / Ishii, R. / Fukunaga, R. / Kobayashi, T. / Sakamoto, K. / Yokoyama, S. |
|---|
| 履歴 | | 登録 | 2008年2月19日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2008年12月2日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2023年11月1日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Methanosarcina mazei (古細菌)
Methanosarcina mazei (古細菌) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.79 Å
分子置換 / 解像度: 1.79 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Chem.Biol. / 年: 2008
ジャーナル: Chem.Biol. / 年: 2008 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2zin.cif.gz
2zin.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2zin.ent.gz
pdb2zin.ent.gz PDB形式
PDB形式 2zin.json.gz
2zin.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/zi/2zin
https://data.pdbj.org/pub/pdb/validation_reports/zi/2zin ftp://data.pdbj.org/pub/pdb/validation_reports/zi/2zin
ftp://data.pdbj.org/pub/pdb/validation_reports/zi/2zin リンク
リンク 集合体
集合体

 要素
要素 Methanosarcina mazei (古細菌) / 株: JCM9314 / 遺伝子: pylS / プラスミド: pET28c / 発現宿主:
Methanosarcina mazei (古細菌) / 株: JCM9314 / 遺伝子: pylS / プラスミド: pET28c / 発現宿主: 








 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SPring-8
SPring-8  / ビームライン: BL41XU / 波長: 1 Å
/ ビームライン: BL41XU / 波長: 1 Å 解析
解析 分子置換
分子置換
 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj















