登録情報 データベース : PDB / ID : 2mlmタイトル Solution structure of sortase A from S. aureus in complex with benzo[d]isothiazol-3-one based inhibitor Sortase family protein キーワード / 機能・相同性 / / / / / 生物種 Staphylococcus aureus (黄色ブドウ球菌)手法 / Model details closest to the average, model1 データ登録者 Jaudzems, K. / Zhulenkovs, D. / Leonchiks, A. ジャーナル : Bioorg.Med.Chem. / 年 : 2014タイトル : Discovery and structure-activity relationship studies of irreversible benzisothiazolinone-based inhibitors against Staphylococcus aureus sortase A transpeptidase.著者 : Zhulenkovs, D. / Rudevica, Z. / Jaudzems, K. / Turks, M. / Leonchiks, A. 履歴 登録 2014年3月3日 登録サイト / 処理サイト 改定 1.0 2014年11月19日 Provider / タイプ 改定 1.1 2014年12月17日 Group 改定 1.2 2018年6月27日 Group / Derived calculations / Experimental preparationカテゴリ / struct_conn / struct_conn_typeItem 改定 1.3 2024年10月30日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Structure summary カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / pdbx_nmr_software / struct_conn / struct_ref_seq_dif / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _struct_conn.pdbx_leaving_atom_flag / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
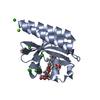
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 引用
引用 ジャーナル: Bioorg.Med.Chem. / 年: 2014
ジャーナル: Bioorg.Med.Chem. / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2mlm.cif.gz
2mlm.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2mlm.ent.gz
pdb2mlm.ent.gz PDB形式
PDB形式 2mlm.json.gz
2mlm.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ml/2mlm
https://data.pdbj.org/pub/pdb/validation_reports/ml/2mlm ftp://data.pdbj.org/pub/pdb/validation_reports/ml/2mlm
ftp://data.pdbj.org/pub/pdb/validation_reports/ml/2mlm リンク
リンク 集合体
集合体
 要素
要素

 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj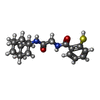

 HSQC
HSQC