+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | local refinement of the primase/DNA/primer region in mutant T4 primosome | |||||||||
 マップデータ マップデータ | focus refinement of the primase/DNA-RNA region | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  phage (ファージ) / phage (ファージ) /  complex / complex /  helicase (ヘリカーゼ) / helicase (ヘリカーゼ) /  REPLICATION (DNA複製) REPLICATION (DNA複製) | |||||||||
| 生物種 |   Escherichia phage T4 (ファージ) Escherichia phage T4 (ファージ) | |||||||||
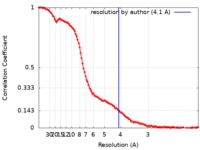
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.1 Å クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Feng X / Li H | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structural basis of the T4 bacteriophage primosome assembly and primer synthesis. 著者: Xiang Feng / Michelle M Spiering / Ruda de Luna Almeida Santos / Stephen J Benkovic / Huilin Li /  要旨: The T4 bacteriophage gp41 helicase and gp61 primase assemble into a primosome to couple DNA unwinding with RNA primer synthesis for DNA replication. How the primosome is assembled and how the primer ...The T4 bacteriophage gp41 helicase and gp61 primase assemble into a primosome to couple DNA unwinding with RNA primer synthesis for DNA replication. How the primosome is assembled and how the primer length is defined are unclear. Here we report a series of cryo-EM structures of T4 primosome assembly intermediates. We show that gp41 alone is an open spiral, and ssDNA binding triggers a large-scale scissor-like conformational change that drives the ring closure and activates the helicase. Helicase activation exposes a cryptic hydrophobic surface to recruit the gp61 primase. The primase binds the helicase in a bipartite mode in which the N-terminal Zn-binding domain and the C-terminal RNA polymerase domain each contain a helicase-interacting motif that bind to separate gp41 N-terminal hairpin dimers, leading to the assembly of one primase on the helicase hexamer. Our study reveals the T4 primosome assembly process and sheds light on the RNA primer synthesis mechanism. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29744.map.gz emd_29744.map.gz | 63.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29744-v30.xml emd-29744-v30.xml emd-29744.xml emd-29744.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29744_fsc.xml emd_29744_fsc.xml | 14.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29744.png emd_29744.png | 44.1 KB | ||
| その他 |  emd_29744_half_map_1.map.gz emd_29744_half_map_1.map.gz emd_29744_half_map_2.map.gz emd_29744_half_map_2.map.gz | 115.8 MB 115.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29744 http://ftp.pdbj.org/pub/emdb/structures/EMD-29744 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29744 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29744 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29744.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29744.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | focus refinement of the primase/DNA-RNA region | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.029 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
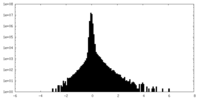
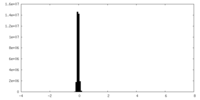
| ファイル | emd_29744_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
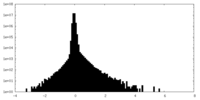
| ファイル | emd_29744_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of mutated T4 bacteriophage helicase gp41 with ssDNA/RNA ...
| 全体 | 名称: Complex of mutated T4 bacteriophage helicase gp41 with ssDNA/RNA hybrid |
|---|---|
| 要素 |
|
-超分子 #1: Complex of mutated T4 bacteriophage helicase gp41 with ssDNA/RNA ...
| 超分子 | 名称: Complex of mutated T4 bacteriophage helicase gp41 with ssDNA/RNA hybrid タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Escherichia phage T4 (ファージ) Escherichia phage T4 (ファージ) |
-分子 #1: Bacteriophage T4 DNA primase
| 分子 | 名称: Bacteriophage T4 DNA primase / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO EC番号:  転移酵素; リンを含む基を移すもの; 核酸を移すもの 転移酵素; リンを含む基を移すもの; 核酸を移すもの |
|---|---|
| 由来(天然) | 生物種:   Escherichia phage T4 (ファージ) Escherichia phage T4 (ファージ) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MSSIPWIDNE FAYRALAHLP KFTQVNNSST FKLRFRCPVC GDSKTDQNKA RGWYYGDNNE GNIHCYNCN YHAPIGIYLK EFEPDLYREY IFEIRKEKGK SRPIEKPKEL PKQPEKKIIK S LPSCVRLD KLAEDHPIIK YVKARCIPKD KWKYLWFTTE WPKLVNSIAP ...文字列: MSSIPWIDNE FAYRALAHLP KFTQVNNSST FKLRFRCPVC GDSKTDQNKA RGWYYGDNNE GNIHCYNCN YHAPIGIYLK EFEPDLYREY IFEIRKEKGK SRPIEKPKEL PKQPEKKIIK S LPSCVRLD KLAEDHPIIK YVKARCIPKD KWKYLWFTTE WPKLVNSIAP GTYKKEISEP RL VIPIYNA NGKAESFQGR ALKKDAPQKY ITIEAYPEAT KIYGVERVKD GDVYVLEGPI DSL FIENGI AITGGQLDLE VVPFKDRRVW VLDNEPRHPD TIKRMTKLVD AGERVMFWDK SPWK SKDVN DMIRKEGATP EQIMEYMKNN IAQGLMAKMR LSKYAKI |
-分子 #2: DNA (70-mer)
| 分子 | 名称: DNA (70-mer) / タイプ: dna / ID: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Escherichia phage T4 (ファージ) Escherichia phage T4 (ファージ) |
| 配列 | 文字列: (DG)(DA)(DA)(DT)(DG)(DA)(DG)(DG)(DA)(DG) (DT)(DA)(DG)(DT)(DA)(DG)(DT)(DG)(DA)(DA) (DT)(DG)(DT)(DA)(DG)(DT)(DG)(DA)(DG) (DG)(DT)(DA)(DA)(DT)(DA)(DT)(DC)(DG)(DG) (DC) (DT)(DG)(DG)(DT)(DC) ...文字列: (DG)(DA)(DA)(DT)(DG)(DA)(DG)(DG)(DA)(DG) (DT)(DA)(DG)(DT)(DA)(DG)(DT)(DG)(DA)(DA) (DT)(DG)(DT)(DA)(DG)(DT)(DG)(DA)(DG) (DG)(DT)(DA)(DA)(DT)(DA)(DT)(DC)(DG)(DG) (DC) (DT)(DG)(DG)(DT)(DC)(DT)(DG)(DG) (DT)(DC)(DT)(DG)(DT)(DG)(DC)(DC)(DA)(DA) (DG)(DT) (DT)(DG)(DC)(DT)(DG)(DC)(DA) (DA)(DA)(DA) |
-分子 #3: RNA (5'-R(*(GTP)P*CP*CP*GP*A)-3')
| 分子 | 名称: RNA (5'-R(*(GTP)P*CP*CP*GP*A)-3') / タイプ: rna / ID: 3 |
|---|---|
| 由来(天然) | 生物種:   Escherichia phage T4 (ファージ) Escherichia phage T4 (ファージ) |
| 配列 | 文字列: (GTP)CCGA |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 構成要素:
詳細: 20 mM HEPES pH 7.8, 100 mM NaCl, 10 mM MgCl2 and 2 mM DTT | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 66.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER |
|---|
 ムービー
ムービー コントローラー
コントローラー
























 Z
Z Y
Y X
X