[日本語] English
 万見
万見- EMDB-29396: Antibody vFP53.02 in complex with HIV-1 envelope trimer BG505 DS-SOSIP -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
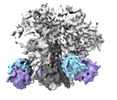
| タイトル | Antibody vFP53.02 in complex with HIV-1 envelope trimer BG505 DS-SOSIP | |||||||||
 マップデータ マップデータ | CryoSPARC map with C3 symmetry | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | fusion peptide envelope trimer murine antibody vFP1 class /  ANTIVIRAL PROTEIN / ANTIVIRAL PROTEIN /  VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell /  viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / エンベロープ (ウイルス) ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell /  viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / identical protein binding / エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.5 Å クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Wang S / Kwong PD | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2023 ジャーナル: J Virol / 年: 2023タイトル: Diverse Murine Vaccinations Reveal Distinct Antibody Classes to Target Fusion Peptide and Variation in Peptide Length to Improve HIV Neutralization. 著者: Mallika Sastry / Anita Changela / Jason Gorman / Kai Xu / Gwo-Yu Chuang / Chen-Hsiang Shen / Cheng Cheng / Hui Geng / Sijy O'Dell / Li Ou / Reda Rawi / Mateo Reveiz / Guillaume B E Stewart- ...著者: Mallika Sastry / Anita Changela / Jason Gorman / Kai Xu / Gwo-Yu Chuang / Chen-Hsiang Shen / Cheng Cheng / Hui Geng / Sijy O'Dell / Li Ou / Reda Rawi / Mateo Reveiz / Guillaume B E Stewart-Jones / Shuishu Wang / Baoshan Zhang / Tongqing Zhou / Andrea Biju / Michael Chambers / Xuejun Chen / Angela R Corrigan / Bob C Lin / Mark K Louder / Krisha McKee / Alexandra F Nazzari / Adam S Olia / Danealle K Parchment / Edward K Sarfo / Tyler Stephens / Jonathan Stuckey / Yaroslav Tsybovsky / Raffaello Verardi / Yiran Wang / Cheng-Yan Zheng / Yuling Chen / Nicole A Doria-Rose / Adrian B McDermott / John R Mascola / Peter D Kwong /  要旨: While neutralizing antibodies that target the HIV-1 fusion peptide have been elicited in mice by vaccination, antibodies reported thus far have been from only a single antibody class that could ...While neutralizing antibodies that target the HIV-1 fusion peptide have been elicited in mice by vaccination, antibodies reported thus far have been from only a single antibody class that could neutralize ~30% of HIV-1 strains. To explore the ability of the murine immune system to generate cross-clade neutralizing antibodies and to investigate how higher breadth and potency might be achieved, we tested 17 prime-boost regimens that utilized diverse fusion peptide-carrier conjugates and HIV-1 envelope trimers with different fusion peptides. We observed priming in mice with fusion peptide-carrier conjugates of variable peptide length to elicit higher neutralizing responses, a result we confirmed in guinea pigs. From vaccinated mice, we isolated 21 antibodies, belonging to 4 distinct classes of fusion peptide-directed antibodies capable of cross-clade neutralization. Top antibodies from each class collectively neutralized over 50% of a 208-strain panel. Structural analyses - both X-ray and cryo-EM - revealed each antibody class to recognize a distinct conformation of fusion peptide and to have a binding pocket capable of accommodating diverse fusion peptides. Murine vaccinations can thus elicit diverse neutralizing antibodies, and altering peptide length during prime can improve the elicitation of cross-clade responses targeting the fusion peptide site of HIV-1 vulnerability. The HIV-1 fusion peptide has been identified as a site for elicitation of broadly neutralizing antibodies, with prior studies demonstrating that priming with fusion peptide-based immunogens and boosting with soluble envelope (Env) trimers can elicit cross-clade HIV-1-neutralizing responses. To improve the neutralizing breadth and potency of fusion peptide-directed responses, we evaluated vaccine regimens that incorporated diverse fusion peptide-conjugates and Env trimers with variation in fusion peptide length and sequence. We found that variation in peptide length during prime elicits enhanced neutralizing responses in mice and guinea pigs. We identified vaccine-elicited murine monoclonal antibodies from distinct classes capable of cross-clade neutralization and of diverse fusion peptide recognition. Our findings lend insight into improved immunogens and regimens for HIV-1 vaccine development. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29396.map.gz emd_29396.map.gz | 171.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29396-v30.xml emd-29396-v30.xml emd-29396.xml emd-29396.xml | 22.6 KB 22.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29396_fsc.xml emd_29396_fsc.xml | 14.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29396.png emd_29396.png | 124.6 KB | ||
| その他 |  emd_29396_half_map_1.map.gz emd_29396_half_map_1.map.gz emd_29396_half_map_2.map.gz emd_29396_half_map_2.map.gz | 318.2 MB 318.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29396 http://ftp.pdbj.org/pub/emdb/structures/EMD-29396 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29396 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29396 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29396.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29396.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoSPARC map with C3 symmetry | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: cryoSPARC half map B
| ファイル | emd_29396_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
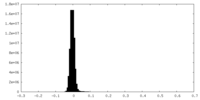
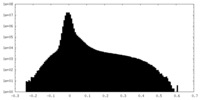
| 注釈 | cryoSPARC half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: cryoSPARC half map A
| ファイル | emd_29396_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
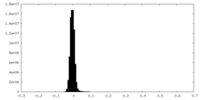
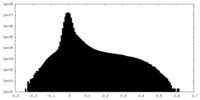
| 注釈 | cryoSPARC half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of antibody Fab vFP53.02 with HIV-1 Env trimer BG505 DS-SOSIP
| 全体 | 名称: Complex of antibody Fab vFP53.02 with HIV-1 Env trimer BG505 DS-SOSIP |
|---|---|
| 要素 |
|
-超分子 #1: Complex of antibody Fab vFP53.02 with HIV-1 Env trimer BG505 DS-SOSIP
| 超分子 | 名称: Complex of antibody Fab vFP53.02 with HIV-1 Env trimer BG505 DS-SOSIP タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|
-超分子 #2: HIV-1 Env trimer BG505 DS-SOSIP
| 超分子 | 名称: HIV-1 Env trimer BG505 DS-SOSIP / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-超分子 #3: Antibody Fab
| 超分子 | 名称: Antibody Fab / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
-分子 #1: Envelope glycoprotein gp41
| 分子 | 名称: Envelope glycoprotein gp41 / タイプ: protein_or_peptide / ID: 1 / 詳細: from HIV-1 BG505 DS-SOSIP / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 17.162525 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: AVGIGAVFLG FLGAAGSTMG AASMTLTVQA RNLLSGIVQQ QSNLLRAIEA QQHLLKLTVW GIKQLQARVL AVERYLRDQQ LLGIWGCSG KLICCTNVPW NSSWSNRNLS EIWDNMTWLQ WDKEISNYTQ IIYGLLEESQ NQQEKNEQDL LALD |
-分子 #2: Envelope glycoprotein gp120
| 分子 | 名称: Envelope glycoprotein gp120 / タイプ: protein_or_peptide / ID: 2 / 詳細: from HIV-1 BG505 DS-SOSIP / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 52.986969 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: AENLWVTVYY GVPVWKDAET TLFCASDAKA YETEKHNVWA THACVPTDPN PQEIHLENVT EEFNMWKNNM VEQMHTDIIS LWDQSLKPC VKLTPLCVTL QCTNVTNNIT DDMRGELKNC SFNMTTELRD KKQKVYSLFY RLDVVQINEN QGNRSNNSNK E YRLINCNT ...文字列: AENLWVTVYY GVPVWKDAET TLFCASDAKA YETEKHNVWA THACVPTDPN PQEIHLENVT EEFNMWKNNM VEQMHTDIIS LWDQSLKPC VKLTPLCVTL QCTNVTNNIT DDMRGELKNC SFNMTTELRD KKQKVYSLFY RLDVVQINEN QGNRSNNSNK E YRLINCNT SACTQACPKV SFEPIPIHYC APAGFAILKC KDKKFNGTGP CPSVSTVQCT HGIKPVVSTQ LLLNGSLAEE EV MIRSENI TNNAKNILVQ FNTPVQINCT RPNNNTRKSI RIGPGQAFYA TGDIIGDIRQ AHCNVSKATW NETLGKVVKQ LRK HFGNNT IIRFANSSGG DLEVTTHSFN CGGEFFYCNT SGLFNSTWIS NTSVQGSNST GSNDSITLPC RIKQIINMWQ RIGQ CMYAP PIQGVIRCVS NITGLILTRD GGSTNSTTET FRPGGGDMRD NWRSELYKYK VVKIEPLGVA PTRCKRRV |
-分子 #3: vFP53.02 Fab heavy chain
| 分子 | 名称: vFP53.02 Fab heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 24.566695 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QAQLQQSGTE LVRPGASVTL SCKASGYKFN DYEIHWVKQT PVHGLEWIGA IVPETGFTAY SQKFRGKALL TADKSSSTVY MDLRSLTSA DSAVFYCSRL QLFGYFDVWG TGTTVIVSSA STKGPSVFPL APSSKSTSGG TAALGCLVKD YFPEPVTVSW N SGALTSGV ...文字列: QAQLQQSGTE LVRPGASVTL SCKASGYKFN DYEIHWVKQT PVHGLEWIGA IVPETGFTAY SQKFRGKALL TADKSSSTVY MDLRSLTSA DSAVFYCSRL QLFGYFDVWG TGTTVIVSSA STKGPSVFPL APSSKSTSGG TAALGCLVKD YFPEPVTVSW N SGALTSGV HTFPAVLQSS GLYSLSSVVT VPSSSLGTQT YICNVNHKPS NTKVDKKVEP KSCDKGLEVL FQ |
-分子 #4: vFP53.02 Fab light chain
| 分子 | 名称: vFP53.02 Fab light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 24.112863 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DVLMTQNPLS LPVSLGDQAS ISCRSSQSVV YSDGNAYLEW YLQKPGQSPK LLIYKASNRF SGVPDRFSAS GSGTDFTLRI SRVETEDLG LYYCFQGTHI PYTFGGGTKL EMKRTVAAPS VFIFPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL Q SGNSQESV ...文字列: DVLMTQNPLS LPVSLGDQAS ISCRSSQSVV YSDGNAYLEW YLQKPGQSPK LLIYKASNRF SGVPDRFSAS GSGTDFTLRI SRVETEDLG LYYCFQGTHI PYTFGGGTKL EMKRTVAAPS VFIFPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL Q SGNSQESV TEQDSKDSTY SLSSTLTLSK ADYEKHKVYA CEVTHQGLSS PVTKSFNRGE C |
-分子 #9: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 9 / コピー数: 21 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: GOLD / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 11520 pixel / デジタル化 - サイズ - 縦: 8184 pixel / 撮影したグリッド数: 1 / 実像数: 11874 / 平均電子線量: 54.4 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 557806 |
FSC曲線 (解像度の算出) | 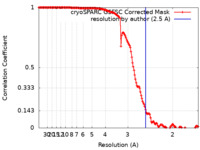 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-8fr6: |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X