+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Glutamine synthetase from Pseudomonas aeruginosa, filament double-unit in compressed conformation | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | glutamine biosynthetic process nitrogen utilization / Structural Genomics / Seattle Structural Genomics Center for Infectious Disease / SSGCID / LIGASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nitrogen utilization / glutamine synthetase / glutamine biosynthetic process / glutamine synthetase activity / ATP binding / membrane / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Phan IQ / Staker B / Shek R / Moser TH / Evans JE / van Voorhis WC / Myler PJ / Seattle Structural Genomics Center for Infectious Disease (SSGCID) | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Glutamine synthetase from Pseudomonas aeruginosa, filament double-unit in compressed conformation 著者: Phan IQ / Staker B / Shek R / Moser TH / Evans JE / van Voorhis WC / Myler PJ | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28965.map.gz emd_28965.map.gz | 32.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28965-v30.xml emd-28965-v30.xml emd-28965.xml emd-28965.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28965.png emd_28965.png | 82.2 KB | ||
| Filedesc metadata |  emd-28965.cif.gz emd-28965.cif.gz | 6.1 KB | ||
| その他 |  emd_28965_half_map_1.map.gz emd_28965_half_map_1.map.gz emd_28965_half_map_2.map.gz emd_28965_half_map_2.map.gz | 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28965 http://ftp.pdbj.org/pub/emdb/structures/EMD-28965 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28965 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28965 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28965_validation.pdf.gz emd_28965_validation.pdf.gz | 912.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28965_full_validation.pdf.gz emd_28965_full_validation.pdf.gz | 912.1 KB | 表示 | |
| XML形式データ |  emd_28965_validation.xml.gz emd_28965_validation.xml.gz | 12.3 KB | 表示 | |
| CIF形式データ |  emd_28965_validation.cif.gz emd_28965_validation.cif.gz | 14.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28965 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28965 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28965 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28965 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8fbpMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28965.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28965.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
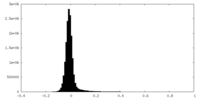
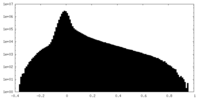
-ハーフマップ: #2
| ファイル | emd_28965_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
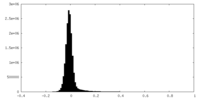
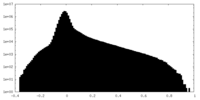
-ハーフマップ: #1
| ファイル | emd_28965_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : P. aeruginosa GS filament.
| 全体 | 名称: P. aeruginosa GS filament. |
|---|---|
| 要素 |
|
-超分子 #1: P. aeruginosa GS filament.
| 超分子 | 名称: P. aeruginosa GS filament. / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Filament formation during sample preparation. |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Glutamine synthetase
| 分子 | 名称: Glutamine synthetase / タイプ: protein_or_peptide / ID: 1 / コピー数: 28 / 光学異性体: LEVO / EC番号: glutamine synthetase |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) |
| 分子量 | 理論値: 53.039711 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAHHHHHHMS YKSHQLIKDH DVKWVDLRFT DTKGKQQHVT MPARDALDDE FFEAGKMFDG SSIAGWKGIE ASDMILMPDD STAVLDPFT EEPTLILVCD IIEPSTMQGY ERDPRNIAKR AEEYLKSTGI GDTVFVGPEP EFFIFDEVKF KSDISGSMFK I FSEQASWN ...文字列: MAHHHHHHMS YKSHQLIKDH DVKWVDLRFT DTKGKQQHVT MPARDALDDE FFEAGKMFDG SSIAGWKGIE ASDMILMPDD STAVLDPFT EEPTLILVCD IIEPSTMQGY ERDPRNIAKR AEEYLKSTGI GDTVFVGPEP EFFIFDEVKF KSDISGSMFK I FSEQASWN TDADIESGNK GHRPGVKGGY FPVPPVDHDH EIRTAMCNAL EEMGLVVEVH HHEVATAGQN EIGVKFNTLV AK ADEVQTL KYCVHNVADA YGKTVTFMPK PLYGDNGSGM HVHMSISKDG KNTFAGEGYA GLSETALYFI GGIIKHGKAL NGF TNPSTN SYKRLVPGFE APVMLAYSAR NRSASIRIPY VSSPKARRIE ARFPDPAANP YLAFAALLMA GLDGIQNKIH PGDA ADKNL YDLPPEEAKE IPQVCGSLKE ALEELDKGRA FLTKGGVFTD EFIDAYIELK SEEEIKVRTF VHPLEYDLYY SV UniProtKB: Glutamine synthetase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7 構成要素:
詳細: Buffer components of purified protein, final vitrification diluted 50/50 with water. | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.3 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL In silico モデル: Single chain modeled with TrRoseTTA was fit into map and the 28-mer model was built in Phenix using NCS operators. The model was refined iteratively using ISOLDE and Phenix Refine + Coot. |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C7 (7回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 2.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: ISOLDE (ver. 1.4) 詳細: CryoSPARC GSFSC: no mask 3.3A, loose 3.0A, tight 2.8A, corrected 2.8A. 使用した粒子像数: 346920 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.3.2) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.3.2) |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 62 当てはまり具合の基準: Cross-correlation coefficient |
|---|---|
| 得られたモデル |  PDB-8fbp: |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X