+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 70S map for: "Atomistic simulations of the E. coli ribosome provide selection criteria for translationally active substrates" | |||||||||
 マップデータ マップデータ | Post-processed, masked 70S map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ribosome / tRNA / e. coli | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.2 Å | |||||||||
 データ登録者 データ登録者 | Watson ZL / Cate JHD | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Chem / 年: 2023 ジャーナル: Nat Chem / 年: 2023タイトル: Atomistic simulations of the Escherichia coli ribosome provide selection criteria for translationally active substrates. 著者: Zoe L Watson / Isaac J Knudson / Fred R Ward / Scott J Miller / Jamie H D Cate / Alanna Schepartz / Ara M Abramyan /  要旨: As genetic code expansion advances beyond L-α-amino acids to backbone modifications and new polymerization chemistries, delineating what substrates the ribosome can accommodate remains a challenge. ...As genetic code expansion advances beyond L-α-amino acids to backbone modifications and new polymerization chemistries, delineating what substrates the ribosome can accommodate remains a challenge. The Escherichia coli ribosome tolerates non-L-α-amino acids in vitro, but few structural insights that explain how are available, and the boundary conditions for efficient bond formation are so far unknown. Here we determine a high-resolution cryogenic electron microscopy structure of the E. coli ribosome containing α-amino acid monomers and use metadynamics simulations to define energy surface minima and understand incorporation efficiencies. Reactive monomers across diverse structural classes favour a conformational space where the aminoacyl-tRNA nucleophile is <4 Å from the peptidyl-tRNA carbonyl with a Bürgi-Dunitz angle of 76-115°. Monomers with free energy minima that fall outside this conformational space do not react efficiently. This insight should accelerate the in vivo and in vitro ribosomal synthesis of sequence-defined, non-peptide heterooligomers. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28255.map.gz emd_28255.map.gz | 52.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28255-v30.xml emd-28255-v30.xml emd-28255.xml emd-28255.xml | 22.2 KB 22.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28255_fsc.xml emd_28255_fsc.xml | 16 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28255.png emd_28255.png | 184.4 KB | ||
| マスクデータ |  emd_28255_msk_1.map emd_28255_msk_1.map | 361.7 MB |  マスクマップ マスクマップ | |
| その他 |  emd_28255_additional_1.map.gz emd_28255_additional_1.map.gz emd_28255_half_map_1.map.gz emd_28255_half_map_1.map.gz emd_28255_half_map_2.map.gz emd_28255_half_map_2.map.gz | 287.7 MB 289.2 MB 289.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28255 http://ftp.pdbj.org/pub/emdb/structures/EMD-28255 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28255 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28255 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28255_validation.pdf.gz emd_28255_validation.pdf.gz | 971.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28255_full_validation.pdf.gz emd_28255_full_validation.pdf.gz | 970.9 KB | 表示 | |
| XML形式データ |  emd_28255_validation.xml.gz emd_28255_validation.xml.gz | 23.4 KB | 表示 | |
| CIF形式データ |  emd_28255_validation.cif.gz emd_28255_validation.cif.gz | 31.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28255 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28255 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28255 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28255 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28255.map.gz / 形式: CCP4 / 大きさ: 361.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28255.map.gz / 形式: CCP4 / 大きさ: 361.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed, masked 70S map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8296 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_28255_msk_1.map emd_28255_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
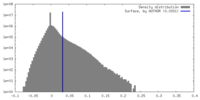
| 密度ヒストグラム |
-追加マップ: 70S map from 3D auto-refine without post-processing
| ファイル | emd_28255_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 70S map from 3D auto-refine without post-processing | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
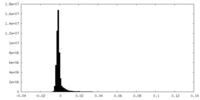
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half-map 2
| ファイル | emd_28255_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
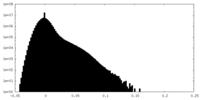
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half-map 1
| ファイル | emd_28255_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 70S ribosome complex with mRNA, A- and P-site Met-NH-tRNAs
| 全体 | 名称: 70S ribosome complex with mRNA, A- and P-site Met-NH-tRNAs |
|---|---|
| 要素 |
|
-超分子 #1: 70S ribosome complex with mRNA, A- and P-site Met-NH-tRNAs
| 超分子 | 名称: 70S ribosome complex with mRNA, A- and P-site Met-NH-tRNAs タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#54 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)