| 登録情報 | データベース: EMDB / ID: EMD-27644
|
|---|
| タイトル | Local refinement of SARS-CoV-2 vaccine induced antibody Ab026500 bound to SARS-CoV-2 HexaPro RBD Spike ectodomain |
|---|
 マップデータ マップデータ | Sharp map from homogeneous C1 refinement in cryosparc |
|---|
 試料 試料 | - 複合体: Ab026500 Fab bound to SARS-CoV-2 HexaPro Spike S1 local refinement
- タンパク質・ペプチド: Spike protein S1
- タンパク質・ペプチド: DH1338 Fab Heavy Chain
- タンパク質・ペプチド: DH1338 Fab Light Chain
|
|---|
 キーワード キーワード | SARS-CoV-2 / VIRAL PROTEIN / Antibody / Fab / RBD / VIRAL PROTEIN-Immune System complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell / Attachment and Entry / membrane fusion / symbiont-mediated suppression of host innate immune response / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / receptor ligand activity / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane類似検索 - 分子機能 Spike (S) protein S1 subunit, receptor-binding domain, SARS-CoV-2 / Spike (S) protein S1 subunit, N-terminal domain, SARS-CoV-like / Betacoronavirus spike (S) glycoprotein S1 subunit N-terminal (NTD) domain profile. / Spike glycoprotein, N-terminal domain superfamily / Betacoronavirus spike (S) glycoprotein S1 subunit C-terminal (CTD) domain profile. / Spike glycoprotein, betacoronavirus / Spike (S) protein S1 subunit, receptor-binding domain, betacoronavirus / Spike S1 subunit, receptor binding domain superfamily, betacoronavirus / Betacoronavirus spike glycoprotein S1, receptor binding / Spike glycoprotein S1, N-terminal domain, betacoronavirus-like ...Spike (S) protein S1 subunit, receptor-binding domain, SARS-CoV-2 / Spike (S) protein S1 subunit, N-terminal domain, SARS-CoV-like / Betacoronavirus spike (S) glycoprotein S1 subunit N-terminal (NTD) domain profile. / Spike glycoprotein, N-terminal domain superfamily / Betacoronavirus spike (S) glycoprotein S1 subunit C-terminal (CTD) domain profile. / Spike glycoprotein, betacoronavirus / Spike (S) protein S1 subunit, receptor-binding domain, betacoronavirus / Spike S1 subunit, receptor binding domain superfamily, betacoronavirus / Betacoronavirus spike glycoprotein S1, receptor binding / Spike glycoprotein S1, N-terminal domain, betacoronavirus-like / Betacoronavirus-like spike glycoprotein S1, N-terminal / Spike glycoprotein S2, coronavirus, heptad repeat 1 / Spike glycoprotein S2, coronavirus, heptad repeat 2 / Coronavirus spike (S) glycoprotein S2 subunit heptad repeat 2 (HR2) region profile. / Coronavirus spike (S) glycoprotein S2 subunit heptad repeat 1 (HR1) region profile. / Spike glycoprotein S2 superfamily, coronavirus / Spike glycoprotein S2, coronavirus / Coronavirus spike glycoprotein S2 / Coronavirus spike glycoprotein S1, C-terminal / Coronavirus spike glycoprotein S1, C-terminal類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Severe acute respiratory syndrome coronavirus 2 (ウイルス) / Severe acute respiratory syndrome coronavirus 2 (ウイルス) /   Macaca mulatta (アカゲザル) Macaca mulatta (アカゲザル) |
|---|
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.38 Å |
|---|
 データ登録者 データ登録者 | Stalls V / May AJ / Acharya P |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | Organization | Grant number | 国 |
|---|
| National Institutes of Health/National Institute Of Allergy and Infectious Diseases (NIH/NIAID) | R01 AI165947 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Local refinement of SARS-CoV-2 vaccine induced antibody Ab026500 bound to SARS-CoV-2 HexaPro RBD Spike ectodomain
著者: Stalls V / Acharya P / May AJ / Malewana D |
|---|
| 履歴 | | 登録 | 2022年7月18日 | - |
|---|
| ヘッダ(付随情報) 公開 | 2023年8月2日 | - |
|---|
| マップ公開 | 2023年8月2日 | - |
|---|
| 更新 | 2023年8月2日 | - |
|---|
| 現状 | 2023年8月2日 | 処理サイト: RCSB / 状態: 公開 |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報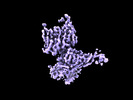
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報

 データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_27644.map.gz
emd_27644.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-27644-v30.xml
emd-27644-v30.xml emd-27644.xml
emd-27644.xml EMDBヘッダ
EMDBヘッダ emd_27644.png
emd_27644.png emd_27644_half_map_1.map.gz
emd_27644_half_map_1.map.gz emd_27644_half_map_2.map.gz
emd_27644_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-27644
http://ftp.pdbj.org/pub/emdb/structures/EMD-27644 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27644
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27644
 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
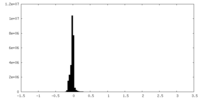
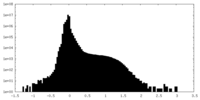
マップ ダウンロード / ファイル: emd_27644.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_27644.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素

 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X