+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Influenza A virus A/Ohio/09/2015 hemagglutinin bound to CR6261 Fab | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |    Influenza A virus (A型インフルエンザウイルス) / Influenza A virus (A型インフルエンザウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
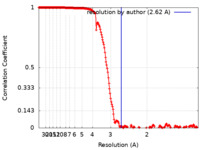
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.62 Å クライオ電子顕微鏡法 / 解像度: 2.62 Å | |||||||||
 データ登録者 データ登録者 | Seattle Structural Genomics Center for Infectious Disease (SSGCID) | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: CryoEM structure of Influenza A virus A/Ohio/09/2015 hemagglutinin bound to CR6261 Fab 著者: Yang M / Edwards TE / Horanyi PS / Lorimer DD | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27440.map.gz emd_27440.map.gz | 339.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27440-v30.xml emd-27440-v30.xml emd-27440.xml emd-27440.xml | 19.5 KB 19.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27440_fsc.xml emd_27440_fsc.xml | 19.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27440.png emd_27440.png | 85.7 KB | ||
| その他 |  emd_27440_half_map_1.map.gz emd_27440_half_map_1.map.gz emd_27440_half_map_2.map.gz emd_27440_half_map_2.map.gz | 614.7 MB 614.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27440 http://ftp.pdbj.org/pub/emdb/structures/EMD-27440 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27440 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27440 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27440.map.gz / 形式: CCP4 / 大きさ: 662.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27440.map.gz / 形式: CCP4 / 大きさ: 662.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.675 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_27440_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
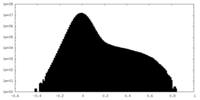
| 投影像・断面図 |
| ||||||||||||
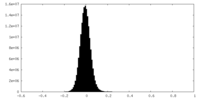
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_27440_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
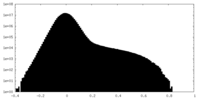
| 投影像・断面図 |
| ||||||||||||
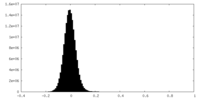
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Influenza A virus A/Ohio/09/2015 hemagglutinin bound to CR6261 Fab
| 全体 | 名称: Influenza A virus A/Ohio/09/2015 hemagglutinin bound to CR6261 Fab |
|---|---|
| 要素 |
|
-超分子 #1: Influenza A virus A/Ohio/09/2015 hemagglutinin bound to CR6261 Fab
| 超分子 | 名称: Influenza A virus A/Ohio/09/2015 hemagglutinin bound to CR6261 Fab タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 理論値: 450 KDa |
-分子 #1: hemagglutinin
| 分子 | 名称: hemagglutinin / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス)株: A/Ohio/09/2016(H1N1) |
| 分子量 | 理論値: 64.428137 KDa |
| 組換発現 | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MVLVNQSHQG FNKEHTSKMV SAIVLYVLLA AAAHSAFAGS DKICIGYHAN NSTDTVDTVL EKNVTVTHSV NLLENKHNGK LCKLRGVAP LHLGKCNIAG WLLGNPECES LATASSWSYI VETSSSNNGT CYPGDFINYE ELREQLSSVS SFEKFEIFPK T SSWPNHET ...文字列: MVLVNQSHQG FNKEHTSKMV SAIVLYVLLA AAAHSAFAGS DKICIGYHAN NSTDTVDTVL EKNVTVTHSV NLLENKHNGK LCKLRGVAP LHLGKCNIAG WLLGNPECES LATASSWSYI VETSSSNNGT CYPGDFINYE ELREQLSSVS SFEKFEIFPK T SSWPNHET NKGVTAACPH AGTNSFYKNL IWLVKKENSY PKINISYTNN RGKEVLVLWA IHHPPTSTDQ QSLYQNANSY VF VGSSRYS RKFEPEIATR PKVRGQAGRM NYYWTLVEPG DKITFEATGN LVVPRYAFAL KRNSGSGIII SETPVHDCDT TCQ TPNGAI NTSLPFQNIH PVTIGECPKY VKSTKLRMAT GLRNIPSIQS RGLFGAIAGF IEGGWTGMID GWYGYHHQNE QGSG YAADL KSTQNAIDGI TNKVNSVIEK MNTQFTAVGK EFSHLERRIE NLNKKVDDGF IDIWTYNAEL LVLLENERTL DYHDS NVKT LYEKVRSQLK NNAKEIGNGC FEFYHKCDDT CMESVKNGTY DYPKYSEEFL VPRGSPGSGY IPEAPRDGQA YVRKDG EWV LLSTFLGHHH HHH |
-分子 #2: CR6261 Fab heavy chain
| 分子 | 名称: CR6261 Fab heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 27.066621 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEWSWVFLFF LSVTTGVHSE VQLVESGAEV KKPGSSVKVS CKASGGPFRS YAISWVRQAP GQGPEWMGGI IPIFGTTKYA PKFQGRVTI TADDFAGTVY MELSSLRSED TAMYYCAKHM GYQVRETMDV WGKGTTVTVS SASTKGPSVF PLAPSSKSTS G GTAALGCL ...文字列: MEWSWVFLFF LSVTTGVHSE VQLVESGAEV KKPGSSVKVS CKASGGPFRS YAISWVRQAP GQGPEWMGGI IPIFGTTKYA PKFQGRVTI TADDFAGTVY MELSSLRSED TAMYYCAKHM GYQVRETMDV WGKGTTVTVS SASTKGPSVF PLAPSSKSTS G GTAALGCL VKDYFPEPVT VSWNSGALTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKRV EP KSCDKHH HHHH |
-分子 #3: CR6261 Fab light chain
| 分子 | 名称: CR6261 Fab light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.514395 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEWSWVFLFF LSVTTGVHSQ SVLTQPPSVS AAPGQKVTIS CSGSSSNIGN DYVSWYQQLP GTAPKLLIYD NNKRPSGIPD RFSGSKSGT SATLGITGLQ TGDEANYYCA TWDRRPTAYV VFGGGTKLTV LGAAAGQPKA APSVTLFPPS SEELQANKAT L VCLISDFY ...文字列: MEWSWVFLFF LSVTTGVHSQ SVLTQPPSVS AAPGQKVTIS CSGSSSNIGN DYVSWYQQLP GTAPKLLIYD NNKRPSGIPD RFSGSKSGT SATLGITGLQ TGDEANYYCA TWDRRPTAYV VFGGGTKLTV LGAAAGQPKA APSVTLFPPS SEELQANKAT L VCLISDFY PGAVTVAWKA DSSPVKAGVE TTTPSKQSNN KYAASSYLSL TPEQWKSHRS YSCQVTHEGS TVEKTVAPTE CS |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 9 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 18 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: EMS Lacey Carbon / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: LACEY | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 12095 / 平均露光時間: 2.3 sec. / 平均電子線量: 80.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X