+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Group A streptococcus Enolase K252A, K255A, K434A, K435A mutant | |||||||||
 マップデータ マップデータ | GAS enolase with K252A, K255A, K434A, and K435 mutations made full map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | metalloenzyme / hPg-receptor / LYASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報phosphopyruvate hydratase / phosphopyruvate hydratase complex / phosphopyruvate hydratase activity / peptidoglycan-based cell wall / glycolytic process / cell surface / magnesium ion binding / extracellular region 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptococcus pyogenes (化膿レンサ球菌) / Streptococcus pyogenes (化膿レンサ球菌) /  Streptococcus sp. 'group A' (バクテリア) Streptococcus sp. 'group A' (バクテリア) | |||||||||
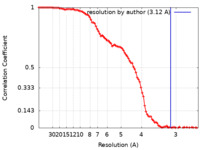
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.12 Å | |||||||||
 データ登録者 データ登録者 | Tjia-Fleck SC / Readnour BM / Castellino FJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2023 ジャーナル: Biochemistry / 年: 2023タイトル: High-Resolution Single-Particle Cryo-EM Hydrated Structure of Enolase Offers Insights into Its Function as a Plasminogen Receptor. 著者: Sheiny Tjia-Fleck / Bradley M Readnour / Yetunde A Ayinuola / Francis J Castellino /  要旨: Cellular plasminogen (Pg) receptors (PgRs) are utilized to recruit Pg; stimulate its activation to the serine protease, plasmin (Pm); and sterically protect the surface Pm from inactivation by host ...Cellular plasminogen (Pg) receptors (PgRs) are utilized to recruit Pg; stimulate its activation to the serine protease, plasmin (Pm); and sterically protect the surface Pm from inactivation by host inhibitors. One such PgR is the moonlighting enzyme, enolase, some of which leaves the cytoplasm and resides at the cell surface to potentially function as a PgR. Since microbes employ conscription of host Pg by PgRs as one virulence mechanism, we explored the structural basis of the ability of enolase (Sen) to function in this manner. Employing single-particle cryo-electron microscopy (cryo-EM), recombinant Sen from was modeled at 2.6 Å as a stable symmetrical doughnut-shaped homooctamer with point group 422 (D4) symmetry, with a monomeric subunit molecular weight of ∼49 kDa. Binding sites for hPg were reported in other studies to include an internal K and the COOH-terminal K residues of Sen. However, in native Sen, the latter are buried within the minor interfaces of the octamer and do not function as a Pg-binding epitope. Whereas Sen and hPg do not interact in solution, when Sen is bound to a surface, hPg interacts with Sen independently of K. PgRs devoid of COOH-terminal lysine utilize lysine isosteres comprising a basic residue, "", and an anionic residue at " + 3" around one turn of an α-helix. We highlight a number of surface-exposed potential hPg-binding lysine isosteres and further conclude that while the octameric structure of Sen is critical for hPg binding, disruption of this octamer without dissociation exposes hPg-binding epitopes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27407.map.gz emd_27407.map.gz | 59.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27407-v30.xml emd-27407-v30.xml emd-27407.xml emd-27407.xml | 16.3 KB 16.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27407_fsc.xml emd_27407_fsc.xml | 11.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27407.png emd_27407.png | 150 KB | ||
| マスクデータ |  emd_27407_msk_1.map emd_27407_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_27407_half_map_1.map.gz emd_27407_half_map_1.map.gz emd_27407_half_map_2.map.gz emd_27407_half_map_2.map.gz | 59.1 MB 59.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27407 http://ftp.pdbj.org/pub/emdb/structures/EMD-27407 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27407 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27407 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27407_validation.pdf.gz emd_27407_validation.pdf.gz | 979.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27407_full_validation.pdf.gz emd_27407_full_validation.pdf.gz | 979.5 KB | 表示 | |
| XML形式データ |  emd_27407_validation.xml.gz emd_27407_validation.xml.gz | 15.8 KB | 表示 | |
| CIF形式データ |  emd_27407_validation.cif.gz emd_27407_validation.cif.gz | 20.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27407 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27407 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27407 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27407 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8dg4MC  7uguC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27407.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27407.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GAS enolase with K252A, K255A, K434A, and K435 mutations made full map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.29 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_27407_msk_1.map emd_27407_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: GAS enolase with K252A, K255A, K434A, and K435...
| ファイル | emd_27407_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GAS enolase with K252A, K255A, K434A, and K435 mutations made first half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: GAS enolase with K252A, K255A, K434A, and K435...
| ファイル | emd_27407_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GAS enolase with K252A, K255A, K434A, and K435 mutations made second half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Octameric Structure of Enolase from Streptococcus Pyogenes
| 全体 | 名称: Octameric Structure of Enolase from Streptococcus Pyogenes |
|---|---|
| 要素 |
|
-超分子 #1: Octameric Structure of Enolase from Streptococcus Pyogenes
| 超分子 | 名称: Octameric Structure of Enolase from Streptococcus Pyogenes タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) / 株: AP53 Streptococcus pyogenes (化膿レンサ球菌) / 株: AP53 |
| 分子量 | 理論値: 400 KDa |
-分子 #1: Enolase
| 分子 | 名称: Enolase / タイプ: protein_or_peptide / ID: 1 / 光学異性体: DEXTRO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus sp. 'group A' (バクテリア) / 株: AP53 Streptococcus sp. 'group A' (バクテリア) / 株: AP53 |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HMSIITDVYA REVLDSRGNP TLEVEVYTES GAFGRGMVPS GASTGEHEAV ELRDGDKSRY LGLGTQKAVD NVNNIIAEAI IGYDVRDQQA IDRAMIALDG TPNKGKLGAN AILGVSIAVA RAAADYLEVP LYTYLGGFNT KVLPTPMMNI INGGSHSDAP IAFQEFMIMP ...文字列: HMSIITDVYA REVLDSRGNP TLEVEVYTES GAFGRGMVPS GASTGEHEAV ELRDGDKSRY LGLGTQKAVD NVNNIIAEAI IGYDVRDQQA IDRAMIALDG TPNKGKLGAN AILGVSIAVA RAAADYLEVP LYTYLGGFNT KVLPTPMMNI INGGSHSDAP IAFQEFMIMP VGAPTFKEGL RWGAEVFHAL KKILKERGLV TAVGDEGGFA PKFEGTEDGV ETILKAIEAA GYEAGENGIM IGFDCASSEF YDAERAVYDY TKFEGEGAAV RTSAEQVDYL EELVNKYPII TIEDGMDEND WDGWKVLTER LGKRVQLVGD DFFVTNTEYL ARGIKENAAN SILIKVNQIG TLTETFEAIE MAKEAGYTAV VSHRSGETED STIADIAVAT NAGQIKTGSL SRTDRIAKYN QLLRIEDQLG EVAQYKGIKS FYNLAA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 構成要素 - 濃度: 0.05 mM / 構成要素 - 式: NaH2PO4 / 構成要素 - 名称: Sodium Phosphate |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | 位相板: VOLTA PHASE PLATE |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 撮影したグリッド数: 1 / 実像数: 2756 / 平均電子線量: 61.37 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.2 µm / 最小 デフォーカス(公称値): 1.1 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)