+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E.coli RNaseP Holoenzyme with Mg2+ | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ribozyme / protein-RNA complex / divalent ion / RNA / HYDROLASE-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報3'-tRNA processing endoribonuclease activity / ribonuclease P complex / ribonuclease P / ribonuclease P activity / tRNA 5'-leader removal / tRNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Huang W / Taylor DJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural and mechanistic basis for recognition of alternative tRNA precursor substrates by bacterial ribonuclease P. 著者: Jiaqiang Zhu / Wei Huang / Jing Zhao / Loc Huynh / Derek J Taylor / Michael E Harris /  要旨: Binding of precursor tRNAs (ptRNAs) by bacterial ribonuclease P (RNase P) involves an encounter complex (ES) that isomerizes to a catalytic conformation (ES*). However, the structures of ...Binding of precursor tRNAs (ptRNAs) by bacterial ribonuclease P (RNase P) involves an encounter complex (ES) that isomerizes to a catalytic conformation (ES*). However, the structures of intermediates and the conformational changes that occur during binding are poorly understood. Here, we show that pairing between the 5' leader and 3'RCCA extending the acceptor stem of ptRNA inhibits ES* formation. Cryo-electron microscopy single particle analysis reveals a dynamic enzyme that becomes ordered upon formation of ES* in which extended acceptor stem pairing is unwound. Comparisons of structures with alternative ptRNAs reveals that once unwinding is completed RNase P primarily uses stacking interactions and shape complementarity to accommodate alternative sequences at its cleavage site. Our study reveals active site interactions and conformational changes that drive molecular recognition by RNase P and lays the foundation for understanding how binding interactions are linked to helix unwinding and catalysis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
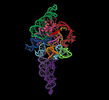
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26637.map.gz emd_26637.map.gz | 4.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26637-v30.xml emd-26637-v30.xml emd-26637.xml emd-26637.xml | 11.3 KB 11.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26637.png emd_26637.png | 67.5 KB | ||
| Filedesc metadata |  emd-26637.cif.gz emd-26637.cif.gz | 5.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26637 http://ftp.pdbj.org/pub/emdb/structures/EMD-26637 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26637 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26637 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26637_validation.pdf.gz emd_26637_validation.pdf.gz | 424.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26637_full_validation.pdf.gz emd_26637_full_validation.pdf.gz | 424.3 KB | 表示 | |
| XML形式データ |  emd_26637_validation.xml.gz emd_26637_validation.xml.gz | 4.4 KB | 表示 | |
| CIF形式データ |  emd_26637_validation.cif.gz emd_26637_validation.cif.gz | 4.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26637 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26637 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26637 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26637 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26637.map.gz / 形式: CCP4 / 大きさ: 4.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26637.map.gz / 形式: CCP4 / 大きさ: 4.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.4878 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : E.coli RNase P holoenzyme with pre-tRNA substrate AU
| 全体 | 名称: E.coli RNase P holoenzyme with pre-tRNA substrate AU |
|---|---|
| 要素 |
|
-超分子 #1: E.coli RNase P holoenzyme with pre-tRNA substrate AU
| 超分子 | 名称: E.coli RNase P holoenzyme with pre-tRNA substrate AU タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: E.coli RNase P RNA
| 分子 | 名称: E.coli RNase P RNA / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 121.191891 KDa |
| 配列 | 文字列: GAAGCUGACC AGACAGUCGC CGCUUCGUCG UCGUCCUCUU CGGGGGAGAC GGGCGGAGGG GAGGAAAGUC CGGGCUCCAU AGGGCAGGG UGCCAGGUAA CGCCUGGGGG GGAAACCCAC GACCAGUGCA ACAGAGAGCA AACCGCCGAU GGCCCGCGCA A GCGGGAUC ...文字列: GAAGCUGACC AGACAGUCGC CGCUUCGUCG UCGUCCUCUU CGGGGGAGAC GGGCGGAGGG GAGGAAAGUC CGGGCUCCAU AGGGCAGGG UGCCAGGUAA CGCCUGGGGG GGAAACCCAC GACCAGUGCA ACAGAGAGCA AACCGCCGAU GGCCCGCGCA A GCGGGAUC AGGUAAGGGU GAAAGGGUGC GGUAAGAGCG CACCGCGCGG CUGGUAACAG UCCGUGGCAC GGUAAACUCC AC CCGGAGC AAGGCCAAAU AGGGGUUCAU AAGGUACGGC CCGUACUGAA CCCGGGUAGG CUGCUUGAGC CAGUGAGCGA UUG CUGGCC UAGAUGAAUG ACUGUCCACG ACAGAACCCG GCUUAUCGGU CAGUUUC GENBANK: GENBANK: CP053595.1 |
-分子 #2: Precursor tRNA substrate U(-1) and A(-2)
| 分子 | 名称: Precursor tRNA substrate U(-1) and A(-2) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 26.281574 KDa |
| 配列 | 文字列: UCUAUGGCUA CGUAGCUCAG UUGGUUAGAG CACAUCACUC AUAAUGAUGG GGUCACAGGU UCGAAUCCCG UCGUAGCCAC CA |
-分子 #3: Ribonuclease P protein component
| 分子 | 名称: Ribonuclease P protein component / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: ribonuclease P |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.10237 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KLAFPRELRL LTPSQFTFVF QQPQRAGTPQ ITILGRLNSL GHPRIGLTVA KKNVRRAHER NRIKRLTRES FRLRQHELPA MDFVVVAKK GVADLDNRAL SEALEKLWRR HCR UniProtKB: Ribonuclease P protein component |
-分子 #4: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 4 / コピー数: 10 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.1 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: ab initio map generated by cryosparc |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 704493 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー











 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)




















