+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
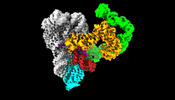
| タイトル | CryoEM Structure of an Group II Intron Retroelement (apo-complex) | |||||||||
 マップデータ マップデータ | ApoRNP Full Consensus Map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNA / intron / group II / maturase / splicing / retrotransposition / RNA BINDING PROTEIN-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  [Eubacterium] rectale (バクテリア) [Eubacterium] rectale (バクテリア) | |||||||||
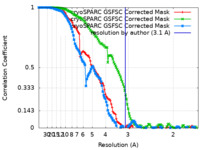
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Chung K / Xu L | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2022 ジャーナル: Science / 年: 2022タイトル: Structures of a mobile intron retroelement poised to attack its structured DNA target. 著者: Kevin Chung / Ling Xu / Pengxin Chai / Junhui Peng / Swapnil C Devarkar / Anna Marie Pyle /  要旨: Group II introns are ribozymes that catalyze their self-excision and function as retroelements that invade DNA. As retrotransposons, group II introns form ribonucleoprotein (RNP) complexes that roam ...Group II introns are ribozymes that catalyze their self-excision and function as retroelements that invade DNA. As retrotransposons, group II introns form ribonucleoprotein (RNP) complexes that roam the genome, integrating by reversal of forward splicing. Here we show that retrotransposition is achieved by a tertiary complex between a structurally elaborate ribozyme, its protein mobility factor, and a structured DNA substrate. We solved cryo-electron microscopy structures of an intact group IIC intron-maturase retroelement that was poised for integration into a DNA stem-loop motif. By visualizing the RNP before and after DNA targeting, we show that it is primed for attack and fits perfectly with its DNA target. This study reveals design principles of a prototypical retroelement and reinforces the hypothesis that group II introns are ancient elements of genetic diversification. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26549.map.gz emd_26549.map.gz | 203.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26549-v30.xml emd-26549-v30.xml emd-26549.xml emd-26549.xml | 32.2 KB 32.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
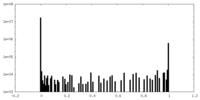
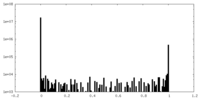
| FSC (解像度算出) |  emd_26549_fsc.xml emd_26549_fsc.xml emd_26549_fsc_2.xml emd_26549_fsc_2.xml emd_26549_fsc_3.xml emd_26549_fsc_3.xml | 12.7 KB 12.7 KB 12.8 KB | 表示 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26549.png emd_26549.png | 56.5 KB | ||
| マスクデータ |  emd_26549_msk_1.map emd_26549_msk_1.map emd_26549_msk_2.map emd_26549_msk_2.map | 216 MB 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-26549.cif.gz emd-26549.cif.gz | 6.8 KB | ||
| その他 |  emd_26549_additional_1.map.gz emd_26549_additional_1.map.gz emd_26549_additional_2.map.gz emd_26549_additional_2.map.gz emd_26549_additional_3.map.gz emd_26549_additional_3.map.gz emd_26549_additional_4.map.gz emd_26549_additional_4.map.gz emd_26549_additional_5.map.gz emd_26549_additional_5.map.gz emd_26549_additional_6.map.gz emd_26549_additional_6.map.gz emd_26549_additional_7.map.gz emd_26549_additional_7.map.gz emd_26549_half_map_1.map.gz emd_26549_half_map_1.map.gz emd_26549_half_map_2.map.gz emd_26549_half_map_2.map.gz | 200.3 MB 10.9 MB 150.2 MB 200.2 MB 200.2 MB 200.2 MB 200.2 MB 200.5 MB 200.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26549 http://ftp.pdbj.org/pub/emdb/structures/EMD-26549 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26549 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26549 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26549_validation.pdf.gz emd_26549_validation.pdf.gz | 1.3 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26549_full_validation.pdf.gz emd_26549_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  emd_26549_validation.xml.gz emd_26549_validation.xml.gz | 21.7 KB | 表示 | |
| CIF形式データ |  emd_26549_validation.cif.gz emd_26549_validation.cif.gz | 28 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26549 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26549 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26549 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26549 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7uimMC  7uinC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26549.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26549.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ApoRNP Full Consensus Map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.832 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+マスク #1
+マスク #2
+追加マップ: ApoRNP Focused Refined (Left) Map
+追加マップ: ApoRNP Focused Refined (Right) Map
+追加マップ: ApoRNP Composite Map
+追加マップ: ApoRNP Focused Refined (Left) Half Map A
+追加マップ: ApoRNP Focused Refined (Left) Half Map B
+追加マップ: ApoRNP Focused Refined (Right) Half Map A
+追加マップ: ApoRNP Focused Refined (Right) Half Map B
+ハーフマップ: ApoRNP Full Consensus Half Map A
+ハーフマップ: ApoRNP Full Consensus Half Map B
- 試料の構成要素
試料の構成要素
-全体 : Complex of RNA and protein
| 全体 | 名称: Complex of RNA and protein |
|---|---|
| 要素 |
|
-超分子 #1: Complex of RNA and protein
| 超分子 | 名称: Complex of RNA and protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  [Eubacterium] rectale (バクテリア) [Eubacterium] rectale (バクテリア) |
| 分子量 | 理論値: 250 KDa |
-分子 #1: E.r IIC Intron
| 分子 | 名称: E.r IIC Intron / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  [Eubacterium] rectale (バクテリア) [Eubacterium] rectale (バクテリア) |
| 分子量 | 理論値: 206.779859 KDa |
| 配列 | 文字列: GUUUGCGCGC CAUGGGCGCG CUCUAACGGG UGUAAGUCCC GAACAUGCCC AGGUAGUGGG AAAUGUAUAG CCGAACAGCA AGGGUGUCU ACUGUGAGGU GGAAUCUGAA GGAAGCUGUA AGCGAAUCUC UGGUCCGACG GACAGAAAUC GCAUAUAAGG C UAGGCUUC ...文字列: GUUUGCGCGC CAUGGGCGCG CUCUAACGGG UGUAAGUCCC GAACAUGCCC AGGUAGUGGG AAAUGUAUAG CCGAACAGCA AGGGUGUCU ACUGUGAGGU GGAAUCUGAA GGAAGCUGUA AGCGAAUCUC UGGUCCGACG GACAGAAAUC GCAUAUAAGG C UAGGCUUC GAGUGAUAAG CUGGCAAAGA ACAGUGAAGU CUAAUAACUA CCACGUUUGU AGAAGCAGAG UAAAUGCGGC GG AUAUAUG GAGAGAAAGA GCGUGCACCU UAAGCGUGGA GGUCUCACAG AGGUUUCAUU AGCCUAGUAA CAACGAACUG UGA GAAGUC AGCCGAGCCC AUAGUAGUGA AGAAGUCUCU GUAAUGGGGA UGGAGCGAAG GGGCGAACAA UCAUUCAGUU UGAG AAUGU CUCGUAUUGC AGAAAUGACA ACAUCUGCCG UAACCAAUCG GGUAAAAGGU GGUCAAAUCA AGCGAGACGG AAAGG AAAG AACGCAUGGA CACAAGUAAU CUAAUUUCGG UUAGAUUACU ACAUCGAAAA GUGUGUUACU UGUUAAGUUG AUUGAA CCG CCGUAUACGG AACCGUACGU ACGGUGGUGU GAGAGGUCGG AAUUUCUCAA UUAAGAGAAA UUCUUCCUAC UCGAU |
-分子 #2: Group II intron reverse transcriptase/maturase
| 分子 | 名称: Group II intron reverse transcriptase/maturase / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA-directed DNA polymerase |
|---|---|
| 由来(天然) | 生物種:  [Eubacterium] rectale (バクテリア) [Eubacterium] rectale (バクテリア) |
| 分子量 | 理論値: 49.083914 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDTSNLMEQI LSSDNLNRAY LQVVRNKGAE GVDGMKYTEL KEHLAKNGET IKGQLRTRKY KPQPARRVEI PKPDGGVRNL GVPTVTDRF IQQAIAQVLT PIYEEQFHDH SYGFRPNRCA QQAILTALNI MNDGNDWIVD IDLEKFFDTV NHDKLMTLIG R TIKDGDVI ...文字列: MDTSNLMEQI LSSDNLNRAY LQVVRNKGAE GVDGMKYTEL KEHLAKNGET IKGQLRTRKY KPQPARRVEI PKPDGGVRNL GVPTVTDRF IQQAIAQVLT PIYEEQFHDH SYGFRPNRCA QQAILTALNI MNDGNDWIVD IDLEKFFDTV NHDKLMTLIG R TIKDGDVI SIVRKYLVSG IMIDDEYEDS IVGTPQGGNL SPLLANIMLN ELDKEMEKRG LNFVRYADDC IIMVGSEMSA NR VMRNISR FIEEKLGLKV NMTKSKVDRP SGLKYLGFGF YFDPRAHQFK AKPHAKSVAK FKKRMKELTC RSWGVSNSYK VEK LNQLIR GWINYFKIGS MKTLCKELDS RIRYRLRMCI WKQWKTPQNQ EKNLVKLGID RNTARRVAYT GKRIAYVCNK GAVN VAISN KRLASFGLIS MLDYYIEKCV TC UniProtKB: Group II intron reverse transcriptase/maturase |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 13 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: AMMONIUM ION
| 分子 | 名称: AMMONIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: NH4 |
|---|---|
| 分子量 | 理論値: 18.038 Da |
| Chemical component information |  ChemComp-NH4: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 5 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 297 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)