+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single-molecule 3D density map of HIV cellular entry by liquid-phase electron tomography (particle #1) | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | HIV cellular entry / CELL INVASION | ||||||||||||
| 生物種 |   Human immunodeficiency virus (ヒト免疫不全ウイルス) / Human immunodeficiency virus (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
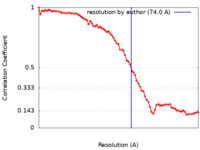
| 手法 | 電子線トモグラフィー法 / 解像度: 74.0 Å | ||||||||||||
 データ登録者 データ登録者 | Kong L / Ren G | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Facile hermetic TEM grid preparation for molecular imaging of hydrated biological samples at room temperature. 著者: Lingli Kong / Jianfang Liu / Meng Zhang / Zhuoyang Lu / Han Xue / Amy Ren / Jiankang Liu / Jinping Li / Wai Li Ling / Gang Ren /    要旨: Although structures of vitrified supramolecular complexes have been determined at near-atomic resolution, elucidating in situ molecular structure in living cells remains a challenge. Here, we report ...Although structures of vitrified supramolecular complexes have been determined at near-atomic resolution, elucidating in situ molecular structure in living cells remains a challenge. Here, we report a straightforward liquid cell technique, originally developed for real-time visualization of dynamics at a liquid-gas interface using transmission electron microscopy, to image wet biological samples. Due to the scattering effects from the liquid phase, the micrographs display an amplitude contrast comparable to that observed in negatively stained samples. We succeed in resolving subunits within the protein complex GroEL imaged in a buffer solution at room temperature. Additionally, we capture various stages of virus cell entry, a process for which only sparse structural data exists due to their transient nature. To scrutinize the morphological details further, we used individual particle electron tomography for 3D reconstruction of each virus. These findings showcase this approach potential as an efficient, cost-effective complement to other microscopy technique in addressing biological questions at the molecular level. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25890.map.gz emd_25890.map.gz | 23.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25890-v30.xml emd-25890-v30.xml emd-25890.xml emd-25890.xml | 12.6 KB 12.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25890_fsc.xml emd_25890_fsc.xml | 7.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25890.png emd_25890.png | 98.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25890 http://ftp.pdbj.org/pub/emdb/structures/EMD-25890 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25890 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25890 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25890_validation.pdf.gz emd_25890_validation.pdf.gz | 345.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25890_full_validation.pdf.gz emd_25890_full_validation.pdf.gz | 344.9 KB | 表示 | |
| XML形式データ |  emd_25890_validation.xml.gz emd_25890_validation.xml.gz | 9.7 KB | 表示 | |
| CIF形式データ |  emd_25890_validation.cif.gz emd_25890_validation.cif.gz | 12.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25890 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25890 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25890 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25890 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25890.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25890.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 21.44 Å | ||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Single molecule 3D map of HIV at its intermediate stage of cellul...
| 全体 | 名称: Single molecule 3D map of HIV at its intermediate stage of cellular entry (Particle #1, half embedded in cell), determined by liquid-phase electron tomography |
|---|---|
| 要素 |
|
-超分子 #1: Single molecule 3D map of HIV at its intermediate stage of cellul...
| 超分子 | 名称: Single molecule 3D map of HIV at its intermediate stage of cellular entry (Particle #1, half embedded in cell), determined by liquid-phase electron tomography タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 詳細: In the penetrating stage of viral entry, in which a virus half embeds into the cell membrane. |
|---|
-超分子 #2: HIV engineered with VSV G glycoprotein
| 超分子 | 名称: HIV engineered with VSV G glycoprotein / タイプ: organelle_or_cellular_component / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus (ヒト免疫不全ウイルス) Human immunodeficiency virus (ヒト免疫不全ウイルス) |
-超分子 #3: cell membrane
| 超分子 | 名称: cell membrane / タイプ: organelle_or_cellular_component / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
 解析 解析 | 電子線トモグラフィー法 |
|---|---|
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 構成要素:
詳細: HeLa cells were grown in Gibco minimum essential media (MEM, containing 10 percent FBS) at 37 degree with 5 percent CO2, and virus-infected cells were prepared by incubating HeLa cells with ...詳細: HeLa cells were grown in Gibco minimum essential media (MEM, containing 10 percent FBS) at 37 degree with 5 percent CO2, and virus-infected cells were prepared by incubating HeLa cells with virus at a ratio of 50 to 1 for 12 hours. | ||||||
|---|---|---|---|---|---|---|---|
| 糖包埋 | 材質: sanwiched by two formvar films 詳細: sample solution (native liquid state, label-free, without any staining) was deposited and then sandwiched between two layers of 300 mesh TEM Formvar-coated copper grids at room temperature. | ||||||
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: FORMVAR / 支持フィルム - トポロジー: CONTINUOUS | ||||||
| 詳細 | HeLa cells were grown in Gibco minimum essential media and virus-infected cells were prepared by incubating HeLa cells with virus at a ratio of 50:1 for 12 hours. | ||||||
| Cryo protectant | Liquid-phase | ||||||
| 切片作成 | その他: NO SECTIONING |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | ZEISS LIBRA120PLUS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 97 / 平均電子線量: 1.0 e/Å2 詳細: The total illumination electron doses were ~30 electron per angstrom square. |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 最大 デフォーカス(補正後): 11.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.4 mm / 最大 デフォーカス(公称値): 11.0 µm / 最小 デフォーカス(公称値): 3.0 µm |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)