+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24676 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | AMC016 SOSIP.v4.2 in complex with PGV04 Fab | ||||||||||||
 マップデータ マップデータ | AMC016 SOSIP.v4.2 in complex with PGV04 Fab | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
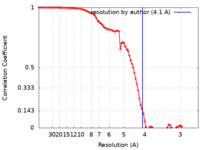
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | ||||||||||||
 データ登録者 データ登録者 | Bader DLV / Cottrell CA / Ward AB | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2022 ジャーナル: J Virol / 年: 2022タイトル: The Glycan Hole Area of HIV-1 Envelope Trimers Contributes Prominently to the Induction of Autologous Neutralization. 著者: Anna Schorcht / Christopher A Cottrell / Pavel Pugach / Rajesh P Ringe / Alvin X Han / Joel D Allen / Tom L G M van den Kerkhof / Gemma E Seabright / Edith E Schermer / Thomas J Ketas / ...著者: Anna Schorcht / Christopher A Cottrell / Pavel Pugach / Rajesh P Ringe / Alvin X Han / Joel D Allen / Tom L G M van den Kerkhof / Gemma E Seabright / Edith E Schermer / Thomas J Ketas / Judith A Burger / Jelle van Schooten / Celia C LaBranche / Gabriel Ozorowski / Natalia de Val / Daniel L V Bader / Hanneke Schuitemaker / Colin A Russell / David C Montefiori / Marit J van Gils / Max Crispin / P J Klasse / Andrew B Ward / John P Moore / Rogier W Sanders /    要旨: The human immunodeficiency virus type 1 (HIV-1) trimeric envelope glycoprotein (Env) is heavily glycosylated, creating a dense glycan shield that protects the underlying peptidic surface from ...The human immunodeficiency virus type 1 (HIV-1) trimeric envelope glycoprotein (Env) is heavily glycosylated, creating a dense glycan shield that protects the underlying peptidic surface from antibody recognition. The absence of conserved glycans, due to missing potential N-linked glycosylation sites (PNGS), can result in strain-specific, autologous neutralizing antibody (NAb) responses. Here, we sought to gain a deeper understanding of the autologous neutralization by introducing holes in the otherwise dense glycan shields of the AMC011 and AMC016 SOSIP trimers. Specifically, when we knocked out the N130 and N289 glycans, which are absent from the well-characterized B41 SOSIP trimer, we observed stronger autologous NAb responses. We also analyzed the highly variable NAb responses induced in rabbits by diverse SOSIP trimers from subtypes A, B, and C. Statistical analysis, using linear regression, revealed that the cumulative area exposed on a trimer by glycan holes correlates with the magnitude of the autologous NAb response. Forty years after the first description of HIV-1, the search for a protective vaccine is still ongoing. The sole target for antibodies that can neutralize the virus are the trimeric envelope glycoproteins (Envs) located on the viral surface. The glycoprotein surface is covered with glycans that shield off the underlying protein components from recognition by the immune system. However, the Env trimers of some viral strains have holes in the glycan shield. Immunized animals developed antibodies against such glycan holes. These antibodies are generally strain specific. Here, we sought to gain a deeper understanding of what drives these specific immune responses. First, we show that strain-specific neutralizing antibody responses can be increased by creating artificial holes in the glycan shield. Second, when studying a diverse set of Env trimers with different characteristics, we found that the surface area of the glycan holes contributes prominently to the induction of strain-specific neutralizing antibodies. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24676.map.gz emd_24676.map.gz | 53.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24676-v30.xml emd-24676-v30.xml emd-24676.xml emd-24676.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24676_fsc.xml emd_24676_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24676.png emd_24676.png | 62.1 KB | ||
| その他 |  emd_24676_half_map_1.map.gz emd_24676_half_map_1.map.gz emd_24676_half_map_2.map.gz emd_24676_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24676 http://ftp.pdbj.org/pub/emdb/structures/EMD-24676 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24676 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24676 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24676_validation.pdf.gz emd_24676_validation.pdf.gz | 697.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24676_full_validation.pdf.gz emd_24676_full_validation.pdf.gz | 697.4 KB | 表示 | |
| XML形式データ |  emd_24676_validation.xml.gz emd_24676_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_24676_validation.cif.gz emd_24676_validation.cif.gz | 20.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24676 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24676 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24676 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24676 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24676.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24676.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | AMC016 SOSIP.v4.2 in complex with PGV04 Fab | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: AMC016 SOSIP.v4.2 in complex with PGV04 Fab
| ファイル | emd_24676_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | AMC016 SOSIP.v4.2 in complex with PGV04 Fab | ||||||||||||
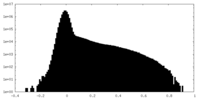
| 投影像・断面図 |
| ||||||||||||
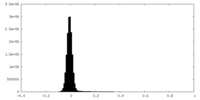
| 密度ヒストグラム |
-ハーフマップ: AMC016 SOSIP.v4.2 in complex with PGV04 Fab
| ファイル | emd_24676_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | AMC016 SOSIP.v4.2 in complex with PGV04 Fab | ||||||||||||
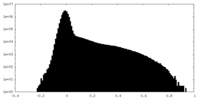
| 投影像・断面図 |
| ||||||||||||
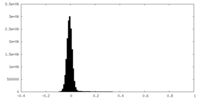
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : AMC016 SOSIP.v4.2 in complex with PGV04 Fab
| 全体 | 名称: AMC016 SOSIP.v4.2 in complex with PGV04 Fab |
|---|---|
| 要素 |
|
-超分子 #1: AMC016 SOSIP.v4.2 in complex with PGV04 Fab
| 超分子 | 名称: AMC016 SOSIP.v4.2 in complex with PGV04 Fab / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-分子 #1: AMC016 gp120
| 分子 | 名称: AMC016 gp120 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 54.890215 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AEEELWVTVY YGVPVWKEAT TTLFCASDAK AYDTEVRNVW ATHACVPTDP SPQEVVLANV TENFNMWKNN MVEQMHEDII SLWDQSLKP CVKLTPLCVT LNCTDLGNAT DAINRNTTDA PNSTLRTMEE KGEIKNCSFN ITTSVRDKMQ KEYATFYKLD I VPIDNDNN ...文字列: AEEELWVTVY YGVPVWKEAT TTLFCASDAK AYDTEVRNVW ATHACVPTDP SPQEVVLANV TENFNMWKNN MVEQMHEDII SLWDQSLKP CVKLTPLCVT LNCTDLGNAT DAINRNTTDA PNSTLRTMEE KGEIKNCSFN ITTSVRDKMQ KEYATFYKLD I VPIDNDNN SYRLINCNTS VITQACPKVS FEPIPIHYCA PAGFAILKCN NKTFNGTGPC TNVSTVQCTH GIRPVVSTQL LL NGSLAEE EIVIRSENFT DNAKTIIVQL NESVEINCTR PNNNTRKSIH IGPGRWFYTT GQIIGNIRQA HCNISRAKWN NTL HKIVKK LREQFRNKTI VFKQSSGGDP EIVMHSFNCG GEFFYCNSTQ LFNSTWYGNE SSDNPGVEGN ITLPCRIKQI INLW QEVGK AMYAPPIGGQ IRCSSNITGL LLTRDGGNNN ITTEIFRPGG GDMRDNWRSE LYKYKVVKIE PLGVAPTKCK RRVVQ RRRR RR |
-分子 #2: AMC016 gp41
| 分子 | 名称: AMC016 gp41 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 17.311746 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVGTIGAMFL GFLGAAGSTM GAASMTLTVQ ARQLLSGIVQ QQSNLLRAPE AQQHLLKLTV WGIKQLQARV LAVERYLKDQ QLLGIWGCS GKLICCTAVP WNASWSNKSL DNIWNNMTWM EWEKEISNYT NLIYNLIEES QNQQEKNEQE LLELD |
-分子 #3: PGV04 Fab heavy chain
| 分子 | 名称: PGV04 Fab heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.759861 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLVQSGSG VKKPGASVRV SCWTSEDIFE RTELIHWVRQ APGQGLEWIG WVKTVTGAVN FGSPDFRQRV SLTRDRDLFT AHMDIRGLT QGDTATYFCA RQKFYTGGQG WYFDLWGRGT LIVVSSASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP E PVTVSWNS ...文字列: QVQLVQSGSG VKKPGASVRV SCWTSEDIFE RTELIHWVRQ APGQGLEWIG WVKTVTGAVN FGSPDFRQRV SLTRDRDLFT AHMDIRGLT QGDTATYFCA RQKFYTGGQG WYFDLWGRGT LIVVSSASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP E PVTVSWNS GALTSGVHTF PAVLQSSGLY SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKKVEPKSC D |
-分子 #4: PGV04 kappa chain
| 分子 | 名称: PGV04 kappa chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.073822 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EIVLTQSPGT LSLSPGETAS LSCTAASYGH MTWYQKKPGQ PPKLLIFATS KRASGIPDRF SGSQFGKQYT LTITRMEPED FARYYCQQL EFFGQGTRLE IRRTVAAPSV FIFPPSDEQL KSGTASVVCL LNNFYPREAK VQWKVDNALQ SGNSQESVTE Q DSKDSTYS ...文字列: EIVLTQSPGT LSLSPGETAS LSCTAASYGH MTWYQKKPGQ PPKLLIFATS KRASGIPDRF SGSQFGKQYT LTITRMEPED FARYYCQQL EFFGQGTRLE IRRTVAAPSV FIFPPSDEQL KSGTASVVCL LNNFYPREAK VQWKVDNALQ SGNSQESVTE Q DSKDSTYS LSSTLTLSKA DYEKHKVYAC EVTHQGLSSP VTKSFNRGEC |
-分子 #9: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 9 / コピー数: 45 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: C-flat-2/2 |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 3801 / 平均露光時間: 7.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7rso: |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X