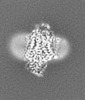
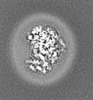
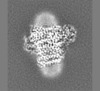
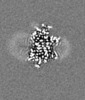
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24265 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
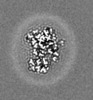
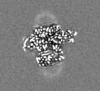
| タイトル | E. coli cytochrome bo3 in MSP nanodisc | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ubiquinone / Heme-copper Oxidoreductase / Electron transport / Bioenergetics / Proton pump / Membrane protein | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytochrome bo3 ubiquinol oxidase activity => GO:0009486 / ubiquinol oxidase (H+-transporting) / cytochrome bo3 ubiquinol oxidase activity / aerobic electron transport chain / oxidoreductase activity, acting on diphenols and related substances as donors, oxygen as acceptor / cytochrome-c oxidase activity / : / electron transport coupled proton transport / ATP synthesis coupled electron transport / aerobic respiration ...cytochrome bo3 ubiquinol oxidase activity => GO:0009486 / ubiquinol oxidase (H+-transporting) / cytochrome bo3 ubiquinol oxidase activity / aerobic electron transport chain / oxidoreductase activity, acting on diphenols and related substances as donors, oxygen as acceptor / cytochrome-c oxidase activity / : / electron transport coupled proton transport / ATP synthesis coupled electron transport / aerobic respiration / membrane => GO:0016020 / copper ion binding / heme binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.19 Å | |||||||||
 データ登録者 データ登録者 | Vallese F / Clarke OB | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2021 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021タイトル: Cryo-EM structures of cytochrome reveal bound phospholipids and ubiquinone-8 in a dynamic substrate binding site. 著者: Jiao Li / Long Han / Francesca Vallese / Ziqiao Ding / Sylvia K Choi / Sangjin Hong / Yanmei Luo / Bin Liu / Chun Kit Chan / Emad Tajkhorshid / Jiapeng Zhu / Oliver Clarke / Kai Zhang / Robert Gennis /   要旨: Two independent structures of the proton-pumping, respiratory cytochrome ubiquinol oxidase (cyt ) have been determined by cryogenic electron microscopy (cryo-EM) in styrene-maleic acid (SMA) ...Two independent structures of the proton-pumping, respiratory cytochrome ubiquinol oxidase (cyt ) have been determined by cryogenic electron microscopy (cryo-EM) in styrene-maleic acid (SMA) copolymer nanodiscs and in membrane scaffold protein (MSP) nanodiscs to 2.55- and 2.19-Å resolution, respectively. The structures include the metal redox centers (heme , heme , and Cu), the redox-active cross-linked histidine-tyrosine cofactor, and the internal water molecules in the proton-conducting D channel. Each structure also contains one equivalent of ubiquinone-8 (UQ8) in the substrate binding site as well as several phospholipid molecules. The isoprene side chain of UQ8 is clamped within a hydrophobic groove in subunit I by transmembrane helix TM0, which is only present in quinol oxidases and not in the closely related cytochrome oxidases. Both structures show carbonyl O1 of the UQ8 headgroup hydrogen bonded to D75 and R71 In both structures, residue H98 occupies two conformations. In conformation 1, H98 forms a hydrogen bond with carbonyl O4 of the UQ8 headgroup, but in conformation 2, the imidazole side chain of H98 has flipped to form a hydrogen bond with E14 at the N-terminal end of TM0. We propose that H98 dynamics facilitate proton transfer from ubiquinol to the periplasmic aqueous phase during oxidation of the substrate. Computational studies show that TM0 creates a channel, allowing access of water to the ubiquinol headgroup and to H98. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24265.map.gz emd_24265.map.gz | 242 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24265-v30.xml emd-24265-v30.xml emd-24265.xml emd-24265.xml | 24.8 KB 24.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24265.png emd_24265.png | 412.6 KB | ||
| Filedesc metadata |  emd-24265.cif.gz emd-24265.cif.gz | 7.1 KB | ||
| その他 |  emd_24265_additional_1.map.gz emd_24265_additional_1.map.gz emd_24265_half_map_1.map.gz emd_24265_half_map_1.map.gz emd_24265_half_map_2.map.gz emd_24265_half_map_2.map.gz | 241.2 MB 475.8 MB 475.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24265 http://ftp.pdbj.org/pub/emdb/structures/EMD-24265 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24265 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24265 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24265_validation.pdf.gz emd_24265_validation.pdf.gz | 918.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24265_full_validation.pdf.gz emd_24265_full_validation.pdf.gz | 917.7 KB | 表示 | |
| XML形式データ |  emd_24265_validation.xml.gz emd_24265_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_24265_validation.cif.gz emd_24265_validation.cif.gz | 18.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24265 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24265 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24265 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24265 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7n9zMC  7cubC  7cuqC  7cuwC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10737 (タイトル: Cryo-EM structures of E. coli cytochrome bo3 in MSP Nanodiscs EMPIAR-10737 (タイトル: Cryo-EM structures of E. coli cytochrome bo3 in MSP NanodiscsData size: 851.4 Data #1: Unaligned multi-frame micrographs of cyt bo3 in MSP Nanodiscs [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24265.map.gz / 形式: CCP4 / 大きさ: 260.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24265.map.gz / 形式: CCP4 / 大きさ: 260.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.41385 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: #1
| ファイル | emd_24265_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
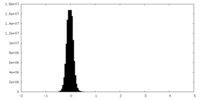
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_24265_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
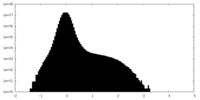
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_24265_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
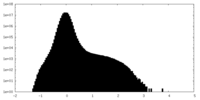
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Cytochrome bo 3 ubiquinol oxidase
+超分子 #1: Cytochrome bo 3 ubiquinol oxidase
+分子 #1: Cytochrome o ubiquinol oxidase, subunit I
+分子 #2: Ubiquinol oxidase subunit 2
+分子 #3: Cytochrome o ubiquinol oxidase
+分子 #4: Cytochrome o ubiquinol oxidase, subunit IV
+分子 #5: Ubiquinone-8
+分子 #6: 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE
+分子 #7: 1,2-Distearoyl-sn-glycerophosphoethanolamine
+分子 #8: HEME O
+分子 #9: PROTOPORPHYRIN IX CONTAINING FE
+分子 #10: CARDIOLIPIN
+分子 #11: COPPER (II) ION
+分子 #12: ZINC ION
+分子 #13: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.6 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 / 構成要素:
| ||||||
| グリッド | モデル: UltrAuFoil R0.6/1 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 58.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.19 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 94681 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー

























 Z
Z Y
Y X
X