+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23751 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Outward facing conformation of the MetNI methionine ABC transporter in complex with lipo-MetQ | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報L-methionine transmembrane transporter activity / D-methionine transmembrane transport / ATPase-coupled transmembrane transporter activity / ATP hydrolysis activity / ATP binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Neisseria meningitidis serogroup B (髄膜炎菌) / Neisseria meningitidis serogroup B (髄膜炎菌) /  Neisseria meningitidis serogroup B (strain MC58) (髄膜炎菌) Neisseria meningitidis serogroup B (strain MC58) (髄膜炎菌) | |||||||||
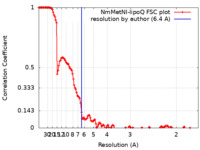
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.4 Å | |||||||||
 データ登録者 データ登録者 | Sharaf NG / Rees DC | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: Characterization of the ABC methionine transporter from reveals that lipidated MetQ is required for interaction. 著者: Naima G Sharaf / Mona Shahgholi / Esther Kim / Jeffrey Y Lai / David G VanderVelde / Allen T Lee / Douglas C Rees /  要旨: NmMetQ is a substrate-binding protein (SBP) from that has been identified as a surface-exposed candidate antigen for meningococcal vaccines. However, this location for NmMetQ challenges the ...NmMetQ is a substrate-binding protein (SBP) from that has been identified as a surface-exposed candidate antigen for meningococcal vaccines. However, this location for NmMetQ challenges the prevailing view that SBPs in Gram-negative bacteria are localized to the periplasmic space to promote interaction with their cognate ABC transporter embedded in the bacterial inner membrane. To elucidate the roles of NmMetQ, we characterized NmMetQ with and without its cognate ABC transporter (NmMetNI). Here, we show that NmMetQ is a lipoprotein (lipo-NmMetQ) that binds multiple methionine analogs and stimulates the ATPase activity of NmMetNI. Using single-particle electron cryo-microscopy, we determined the structures of NmMetNI in the presence and absence of lipo-NmMetQ. Based on our data, we propose that NmMetQ tethers to membranes via a lipid anchor and has dual function and localization, playing a role in NmMetNI-mediated transport at the inner membrane and moonlighting on the bacterial surface. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23751.map.gz emd_23751.map.gz | 229.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23751-v30.xml emd-23751-v30.xml emd-23751.xml emd-23751.xml | 12.4 KB 12.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23751_fsc.xml emd_23751_fsc.xml | 14.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23751.png emd_23751.png | 29.6 KB | ||
| Filedesc metadata |  emd-23751.cif.gz emd-23751.cif.gz | 5.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23751 http://ftp.pdbj.org/pub/emdb/structures/EMD-23751 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23751 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23751 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23751.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23751.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.856 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : ABC transporter, permease protein, Lipoprotein, ABC transporter, ...
| 全体 | 名称: ABC transporter, permease protein, Lipoprotein, ABC transporter, ATP-binding protein |
|---|---|
| 要素 |
|
-超分子 #1: ABC transporter, permease protein, Lipoprotein, ABC transporter, ...
| 超分子 | 名称: ABC transporter, permease protein, Lipoprotein, ABC transporter, ATP-binding protein タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Neisseria meningitidis serogroup B (髄膜炎菌) Neisseria meningitidis serogroup B (髄膜炎菌)株: strain MC58 |
-分子 #1: ABC transporter, permease protein
| 分子 | 名称: ABC transporter, permease protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Neisseria meningitidis serogroup B (strain MC58) (髄膜炎菌) Neisseria meningitidis serogroup B (strain MC58) (髄膜炎菌)株: MC58 |
| 分子量 | 理論値: 24.363064 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MADLTFQQAV STIVGMKDEI FRALGETFVM VGLSTTFAVI FGTLLGVLLF VTSSRQLHYN KLVNFLLDNL VNLMRAFPFV ILMIAMIPA TRAIVGSTIG PVAASLVLSV SGLFYFARLV EQNLREVPKG VIEAAAAMGA PPIAIVCKVL LNEARAGMVS S ITVLAIGL ...文字列: MADLTFQQAV STIVGMKDEI FRALGETFVM VGLSTTFAVI FGTLLGVLLF VTSSRQLHYN KLVNFLLDNL VNLMRAFPFV ILMIAMIPA TRAIVGSTIG PVAASLVLSV SGLFYFARLV EQNLREVPKG VIEAAAAMGA PPIAIVCKVL LNEARAGMVS S ITVLAIGL LSYSAAAGMI GGGGLGDLAI RYGYYRYQTE VIIFIVALLV LLVILIQSTG NALARKLDKR UniProtKB: ABC transporter, permease protein |
-分子 #2: Lipoprotein
| 分子 | 名称: Lipoprotein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Neisseria meningitidis serogroup B (strain MC58) (髄膜炎菌) Neisseria meningitidis serogroup B (strain MC58) (髄膜炎菌)株: MC58 |
| 分子量 | 理論値: 33.010246 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKTFFKTLSA AALALILAAC GGQKDSAPAA SASAAADNGA AKKEIVFGTT VGDFGDMVKE QIQAELEKKG YTVKLVEFTD YVRPNLALA EGELDINVFQ HKPYLDDFKK EHNLDITEVF QVPTAPLGLY PGKLKSLEEV KDGSTVSAPN DPSNFARVLV M LDELGWIK ...文字列: MKTFFKTLSA AALALILAAC GGQKDSAPAA SASAAADNGA AKKEIVFGTT VGDFGDMVKE QIQAELEKKG YTVKLVEFTD YVRPNLALA EGELDINVFQ HKPYLDDFKK EHNLDITEVF QVPTAPLGLY PGKLKSLEEV KDGSTVSAPN DPSNFARVLV M LDELGWIK LKDGINPLTA SKADIAENLK NIKIVELEAA QLPRSRADVD FAVVNGNYAI SSGMKLTEAL FQEPSFAYVN WS AVKTADK DSQWLKDVTE AYNSDAFKAY AHKRFEGYKS PAAWNEGAAK KLEHHHHHHH HHH UniProtKB: Lipoprotein |
-分子 #3: ABC transporter, ATP-binding protein
| 分子 | 名称: ABC transporter, ATP-binding protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Neisseria meningitidis serogroup B (strain MC58) (髄膜炎菌) Neisseria meningitidis serogroup B (strain MC58) (髄膜炎菌)株: MC58 |
| 分子量 | 理論値: 29.866152 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGHHHHHHHH HHSSGHIDDD DKHMIILDKV SKHYQTRDKT RFAAVEPTSL EIRDGEIFGL MGYSGAGKST LLRLINLLER PDSGKVNVC GQELTALDAA ALRQARQNIG MVFQQFNLLS NRTVADNVAF PLEIAGWPSE KIKARVKECL EIVGLTERAG H YPAQLSGG ...文字列: MGHHHHHHHH HHSSGHIDDD DKHMIILDKV SKHYQTRDKT RFAAVEPTSL EIRDGEIFGL MGYSGAGKST LLRLINLLER PDSGKVNVC GQELTALDAA ALRQARQNIG MVFQQFNLLS NRTVADNVAF PLEIAGWPSE KIKARVKECL EIVGLTERAG H YPAQLSGG QKQRVGIARA LAPKPQVILA DEPTSALDPA TTRSVLECLE DINKRFNVTI VIVTHEMSVI RRLCDRAALL DK GKVVEIV EVRGNQIHAQ SDIGRELIRE D UniProtKB: Cell division ATP-binding protein FtsE |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: correlation coefficient |
|---|---|
| 得られたモデル |  PDB-7mbz: |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)