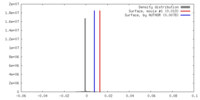
登録情報 データベース : EMDB / ID : EMD-23739タイトル Cryo-EM structure of MLL1-NCP (H3K4M) complex, mode02 Mode02 複合体 : MLL1 in complex with RbBP5, WDR5, SET1, and ASH2L bound to the nucleosome (Mode02)複合体 : RbBP5, WDR5, SET1, and ASH2Lタンパク質・ペプチド : Retinoblastoma-binding protein 5タンパク質・ペプチド : WD repeat-containing protein 5タンパク質・ペプチド : Histone-lysine N-methyltransferase 2Aタンパク質・ペプチド : Set1/Ash2 histone methyltransferase complex subunit ASH2複合体 : nucleosomeタンパク質・ペプチド : Histone H3タンパク質・ペプチド : Histone H4タンパク質・ペプチド : Histone H2Aタンパク質・ペプチド : Histone H2B 1.1DNA : DNA (146-MER)DNA : DNA (146-MER) / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
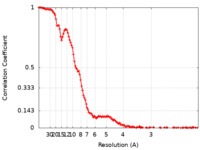
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト) / Xenopus laevis (アフリカツメガエル) / synthetic construct (人工物) 手法 / / 解像度 : 4.02 Å Park SH / Ayoub A 資金援助 Organization Grant number 国 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) GM082856 National Institutes of Health/National Cancer Institute (NIH/NCI) CA250329
ジャーナル : Biochemistry / 年 : 2022タイトル : Regulation of MLL1 Methyltransferase Activity in Two Distinct Nucleosome Binding Modes.著者 : Alex Ayoub / Sang Ho Park / Young-Tae Lee / Uhn-Soo Cho / Yali Dou / 要旨 : Cryo-EM structures of the KMT2A/MLL1 core complex bound on nucleosome core particles (NCPs) suggest unusual rotational dynamics of the MLL1 complex approaching its physiological substrate. However, ... Cryo-EM structures of the KMT2A/MLL1 core complex bound on nucleosome core particles (NCPs) suggest unusual rotational dynamics of the MLL1 complex approaching its physiological substrate. However, the functional implication of such dynamics remains unclear. Here, we show that the MLL1 core complex also shows high rotational dynamics bound on the NCP carrying the catalytically inert histone H3 lysine 4 to methionine (K4M) mutation. There are two major binding modes of the MLL1 complex on the NCP. Importantly, disruption of only one of the binding modes compromised the overall MLL1 activity in an NCP-specific manner. We propose that the MLL1 core complex probably exists in an equilibrium of poised and active binding modes. The high rotational dynamics of the MLL1 complex on the NCP is a feature that can be exploited for loci-specific regulation of H3K4 methylation in higher eukaryotes. 履歴 登録 2021年4月1日 - ヘッダ(付随情報) 公開 2021年12月29日 - マップ公開 2021年12月29日 - 更新 2025年6月4日 - 現状 2025年6月4日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト) /
Homo sapiens (ヒト) /  データ登録者
データ登録者 米国, 2件
米国, 2件  引用
引用 ジャーナル: Biochemistry / 年: 2022
ジャーナル: Biochemistry / 年: 2022
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_23739.map.gz
emd_23739.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-23739-v30.xml
emd-23739-v30.xml emd-23739.xml
emd-23739.xml EMDBヘッダ
EMDBヘッダ emd_23739_fsc.xml
emd_23739_fsc.xml FSCデータファイル
FSCデータファイル emd_23739.png
emd_23739.png emd-23739.cif.gz
emd-23739.cif.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-23739
http://ftp.pdbj.org/pub/emdb/structures/EMD-23739 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23739
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23739 emd_23739_validation.pdf.gz
emd_23739_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_23739_full_validation.pdf.gz
emd_23739_full_validation.pdf.gz emd_23739_validation.xml.gz
emd_23739_validation.xml.gz emd_23739_validation.cif.gz
emd_23739_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23739
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23739 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23739
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23739 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_23739.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_23739.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)