+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23515 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | XLF, XRCC4 and LigIV BRCT in NHEJ Short-range synaptic complex | |||||||||||||||||||||
 マップデータ マップデータ | XLF, XRCC4 and LigIV BRCT in NHEJ Short-range synaptic complex | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||
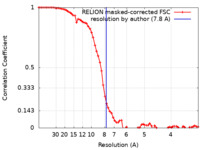
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.8 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | He Y / Chen S | |||||||||||||||||||||
| 資金援助 |  米国, 6件 米国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: Structural basis of long-range to short-range synaptic transition in NHEJ. 著者: Siyu Chen / Linda Lee / Tasmin Naila / Susan Fishbain / Annie Wang / Alan E Tomkinson / Susan P Lees-Miller / Yuan He /   要旨: DNA double-strand breaks (DSBs) are a highly cytotoxic form of DNA damage and the incorrect repair of DSBs is linked to carcinogenesis. The conserved error-prone non-homologous end joining (NHEJ) ...DNA double-strand breaks (DSBs) are a highly cytotoxic form of DNA damage and the incorrect repair of DSBs is linked to carcinogenesis. The conserved error-prone non-homologous end joining (NHEJ) pathway has a key role in determining the effects of DSB-inducing agents that are used to treat cancer as well as the generation of the diversity in antibodies and T cell receptors. Here we applied single-particle cryo-electron microscopy to visualize two key DNA-protein complexes that are formed by human NHEJ factors. The Ku70/80 heterodimer (Ku), the catalytic subunit of the DNA-dependent protein kinase (DNA-PKcs), DNA ligase IV (LigIV), XRCC4 and XLF form a long-range synaptic complex, in which the DNA ends are held approximately 115 Å apart. Two DNA end-bound subcomplexes comprising Ku and DNA-PKcs are linked by interactions between the DNA-PKcs subunits and a scaffold comprising LigIV, XRCC4, XLF, XRCC4 and LigIV. The relative orientation of the DNA-PKcs molecules suggests a mechanism for autophosphorylation in trans, which leads to the dissociation of DNA-PKcs and the transition into the short-range synaptic complex. Within this complex, the Ku-bound DNA ends are aligned for processing and ligation by the XLF-anchored scaffold, and a single catalytic domain of LigIV is stably associated with a nick between the two Ku molecules, which suggests that the joining of both strands of a DSB involves both LigIV molecules. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23515.map.gz emd_23515.map.gz | 23 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23515-v30.xml emd-23515-v30.xml emd-23515.xml emd-23515.xml | 19.3 KB 19.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23515_fsc.xml emd_23515_fsc.xml | 6.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23515.png emd_23515.png | 86.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23515 http://ftp.pdbj.org/pub/emdb/structures/EMD-23515 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23515 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23515 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23515_validation.pdf.gz emd_23515_validation.pdf.gz | 315.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23515_full_validation.pdf.gz emd_23515_full_validation.pdf.gz | 314.8 KB | 表示 | |
| XML形式データ |  emd_23515_validation.xml.gz emd_23515_validation.xml.gz | 9.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23515 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23515 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23515 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23515 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23515.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23515.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | XLF, XRCC4 and LigIV BRCT in NHEJ Short-range synaptic complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.66 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : XLF, XRCC4 and LigIV BRCT in NHEJ Short-range synaptic complex
| 全体 | 名称: XLF, XRCC4 and LigIV BRCT in NHEJ Short-range synaptic complex |
|---|---|
| 要素 |
|
-超分子 #1: XLF, XRCC4 and LigIV BRCT in NHEJ Short-range synaptic complex
| 超分子 | 名称: XLF, XRCC4 and LigIV BRCT in NHEJ Short-range synaptic complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: XLF-XRCC4-LigIV BRCT |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 280 KDa |
-分子 #1: XRCC4_HUMAN DNA repair protein XRCC4
| 分子 | 名称: XRCC4_HUMAN DNA repair protein XRCC4 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: MERKISRIHL VSEPSITHFL QVSWEKTLES GFVITLTDGH SAWTGTVSES EISQEADDMA MEKGKYVGEL RKALLSGAGP ADVYTFNFSK ESCYFFFEKN LKDVSFRLGS FNLEKVENPA EVIRELICYC LDTIAENQAK NEHLQKENER LLRDWNDVQG RFEKCVSAKE ...文字列: MERKISRIHL VSEPSITHFL QVSWEKTLES GFVITLTDGH SAWTGTVSES EISQEADDMA MEKGKYVGEL RKALLSGAGP ADVYTFNFSK ESCYFFFEKN LKDVSFRLGS FNLEKVENPA EVIRELICYC LDTIAENQAK NEHLQKENER LLRDWNDVQG RFEKCVSAKE ALETDLYKRF ILVLNEKKTK IRSLHNKLLN AAQEREKDIK QEGETAICSE MTADRDPVYD ESTDEESENQ TDLSGLASAA VSKDDSIISS LDVTDIAPSR KRRQRMQRNL GTEPKMAPQE NQLQEKENSR PDSSLPETSK KEHISAENMS LETLRNSSPE DLFDEI |
-分子 #2: NHEJ1_HUMAN Non-homologous end-joining factor 1
| 分子 | 名称: NHEJ1_HUMAN Non-homologous end-joining factor 1 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: MEELEQGLLM QPWAWLQLAE NSLLAKVFIT KQGYALLVSD LQQVWHEQVD TSVVSQRAKE LNKRLTAPPA AFLCHLDNLL RPLLKDAAHP SEATFSCDCV ADALILRVRS ELSGLPFYWN FHCMLASPSL VSQHLIRPLM GMSLALQCQV RELATLLHMK DLEIQDYQES ...文字列: MEELEQGLLM QPWAWLQLAE NSLLAKVFIT KQGYALLVSD LQQVWHEQVD TSVVSQRAKE LNKRLTAPPA AFLCHLDNLL RPLLKDAAHP SEATFSCDCV ADALILRVRS ELSGLPFYWN FHCMLASPSL VSQHLIRPLM GMSLALQCQV RELATLLHMK DLEIQDYQES GATLIRDRLK TEPFEENSFL EQFMIEKLPE ACSIGDGKPF VMNLQDLYMA VTTQEVQVGQ KHQGAGDPHT SNSASLQGID SQCVNQPEQL VSSAPTLSAP EKESTGTSGP LQRPQLSKVK RKKPRGLFS |
-分子 #3: DNLI4_HUMAN DNA ligase 4
| 分子 | 名称: DNLI4_HUMAN DNA ligase 4 / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: SNIFEDVEFC VMSGTDSQPK PDLENRIAEF GGYIVQNPGP DTYCVIAGSE NIRVKNIILS NKHDVVKPAW LLECFKTKSF VPWQPRFMIH MCPSTKEHFA REYDCYGDSY FIDTDLNQLK EVFSGIKNSN EQTPEEMASL IADLEYRYSW DCSPLSMFRR HTVYLDSYAV ...文字列: SNIFEDVEFC VMSGTDSQPK PDLENRIAEF GGYIVQNPGP DTYCVIAGSE NIRVKNIILS NKHDVVKPAW LLECFKTKSF VPWQPRFMIH MCPSTKEHFA REYDCYGDSY FIDTDLNQLK EVFSGIKNSN EQTPEEMASL IADLEYRYSW DCSPLSMFRR HTVYLDSYAV INDLSTKNEG TRLAIKALEL RFHGAKVVSC LAEGVSHVII GEDHSRVADF KAFRRTFKRK FKILKESWVT DSIDKCELQE ENQYLI |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.9 構成要素:
| |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R3.5/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 200.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: OTHER / 前処理 - 気圧: 101.325 kPa | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 11520 pixel / デジタル化 - サイズ - 縦: 8184 pixel / 撮影したグリッド数: 2 / 実像数: 32723 / 平均露光時間: 0.0426 sec. / 平均電子線量: 46.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: DARK FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 30000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation coefficient |
|---|
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)