+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2346 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bacteriophage phi6 procapsids with different composition of accessory proteins | |||||||||
 マップデータ マップデータ | recombinant phi6 procapsid composed of P1 and P4 subunits (i.e. without the P2 and P7 subunits) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | dsRNA virus / Cystoviridae / procapsid / RNA polymerase / P7 subunit / location / occupancy | |||||||||
| 生物種 |  Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 12.4 Å | |||||||||
 データ登録者 データ登録者 | Nemecek D / Qiao J / Mindich L / Steven AC / Heymann JB | |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2012 ジャーナル: J Virol / 年: 2012タイトル: Packaging accessory protein P7 and polymerase P2 have mutually occluding binding sites inside the bacteriophage 6 procapsid. 著者: Daniel Nemecek / Jian Qiao / Leonard Mindich / Alasdair C Steven / J Bernard Heymann /  要旨: Bacteriophage 6 is a double-stranded RNA (dsRNA) virus whose genome is packaged sequentially as three single-stranded RNA (ssRNA) segments into an icosahedral procapsid which serves as a compartment ...Bacteriophage 6 is a double-stranded RNA (dsRNA) virus whose genome is packaged sequentially as three single-stranded RNA (ssRNA) segments into an icosahedral procapsid which serves as a compartment for genome replication and transcription. The procapsid shell consists of 60 copies each of P1(A) and P1(B), two nonequivalent conformers of the P1 protein. Hexamers of the packaging ATPase P4 are mounted over the 5-fold vertices, and monomers of the RNA-dependent RNA polymerase (P2) attach to the inner surface, near the 3-fold axes. A fourth protein, P7, is needed for packaging and also promotes assembly. We used cryo-electron microscopy to localize P7 by difference mapping of procapsids with different protein compositions. We found that P7 resides on the interior surface of the P1 shell and appears to be monomeric. Its binding sites are arranged around the 3-fold axes, straddling the interface between two P1(A) subunits. Thus, P7 may promote assembly by stabilizing an initiation complex. Only about 20% of the 60 P7 binding sites were occupied in our preparations. P7 density overlaps P2 density similarly mapped, implying mutual occlusion. The known structure of the 12 homolog fits snugly into the P7 density. Both termini-which have been implicated in RNA binding-are oriented toward the adjacent 5-fold vertex, the entry pathway of ssRNA segments. Thus, P7 may promote packaging either by interacting directly with incoming RNA or by modulating the structure of the translocation pore. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2346.map.gz emd_2346.map.gz | 227.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2346-v30.xml emd-2346-v30.xml emd-2346.xml emd-2346.xml | 9.4 KB 9.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-2346.png EMD-2346.png | 2.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2346 http://ftp.pdbj.org/pub/emdb/structures/EMD-2346 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2346 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2346 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2346_validation.pdf.gz emd_2346_validation.pdf.gz | 298.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2346_full_validation.pdf.gz emd_2346_full_validation.pdf.gz | 297.4 KB | 表示 | |
| XML形式データ |  emd_2346_validation.xml.gz emd_2346_validation.xml.gz | 7.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2346 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2346 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2346 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2346 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2346.map.gz / 形式: CCP4 / 大きさ: 238.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2346.map.gz / 形式: CCP4 / 大きさ: 238.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | recombinant phi6 procapsid composed of P1 and P4 subunits (i.e. without the P2 and P7 subunits) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.63 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
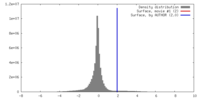
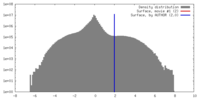
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : recombinant phi6 procapsid composed of P1 and P4 subunits
| 全体 | 名称: recombinant phi6 procapsid composed of P1 and P4 subunits |
|---|---|
| 要素 |
|
-超分子 #1000: recombinant phi6 procapsid composed of P1 and P4 subunits
| 超分子 | 名称: recombinant phi6 procapsid composed of P1 and P4 subunits タイプ: sample / ID: 1000 / 集合状態: icosahedral shell without acessory proteins / Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 12.6 MDa |
-超分子 #1: Pseudomonas phage phi6
| 超分子 | 名称: Pseudomonas phage phi6 / タイプ: virus / ID: 1 / Name.synonym: bacteriophage phi6 / NCBI-ID: 10879 / 生物種: Pseudomonas phage phi6 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes / Syn species name: bacteriophage phi6 |
|---|---|
| 宿主 | 生物種:  Pseudomonas syringae (バクテリア) / 別称: BACTERIA(EUBACTERIA) Pseudomonas syringae (バクテリア) / 別称: BACTERIA(EUBACTERIA) |
| Host system | 生物種:  |
| 分子量 | 理論値: 12.6 MDa |
| ウイルス殻 | Shell ID: 1 / 直径: 450 Å / T番号(三角分割数): 2 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 10mM Tris, 5mM MgCl2 |
| グリッド | 詳細: Q-foil 2/2/300 mesh copper grid |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 120 K / 装置: FEI VITROBOT MARK I / 手法: Blot for 2 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG |
|---|---|
| 日付 | 2011年2月20日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 6.35 µm / 実像数: 4047 / 平均電子線量: 15 e/Å2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 38800 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.05 µm / 最小 デフォーカス(公称値): 0.65 µm / 倍率(公称値): 38000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | particles were selected manually, CTF was determined from whole micrographs |
|---|---|
| CTF補正 | 詳細: CTF was determined from whole micrographs |
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 12.4 Å / 解像度の算出法: FSC 0.33 CUT-OFF / ソフトウェア - 名称: Bsoft / 使用した粒子像数: 4047 |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)