+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23154 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain EM map of SARS-CoV-2 spike protein (trimer) with Fab COV2-2489 (NTD) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell / membrane fusion / Attachment and Entry / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / receptor ligand activity / symbiont-mediated suppression of host innate immune response / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 25.0 Å | |||||||||
 データ登録者 データ登録者 | Binshtein E / Crowe JE | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2021 ジャーナル: bioRxiv / 年: 2021タイトル: Neutralizing and protective human monoclonal antibodies recognizing the N-terminal domain of the SARS-CoV-2 spike protein. 著者: Naveenchandra Suryadevara / Swathi Shrihari / Pavlo Gilchuk / Laura A VanBlargan / Elad Binshtein / Seth J Zost / Rachel S Nargi / Rachel E Sutton / Emma S Winkler / Elaine C Chen / Mallorie ...著者: Naveenchandra Suryadevara / Swathi Shrihari / Pavlo Gilchuk / Laura A VanBlargan / Elad Binshtein / Seth J Zost / Rachel S Nargi / Rachel E Sutton / Emma S Winkler / Elaine C Chen / Mallorie E Fouch / Edgar Davidson / Benjamin J Doranz / Robert H Carnahan / Larissa B Thackray / Michael S Diamond / James E Crowe 要旨: Most human monoclonal antibodies (mAbs) neutralizing SARS-CoV-2 recognize the spike (S) protein receptor-binding domain and block virus interactions with the cellular receptor angiotensin-converting ...Most human monoclonal antibodies (mAbs) neutralizing SARS-CoV-2 recognize the spike (S) protein receptor-binding domain and block virus interactions with the cellular receptor angiotensin-converting enzyme 2. We describe a panel of human mAbs binding to diverse epitopes on the N-terminal domain (NTD) of S protein from SARS-CoV-2 convalescent donors and found a minority of these possessed neutralizing activity. Two mAbs (COV2-2676 and COV2-2489) inhibited infection of authentic SARS-CoV-2 and recombinant VSV/SARS-CoV-2 viruses. We mapped their binding epitopes by alanine-scanning mutagenesis and selection of functional SARS-CoV-2 S neutralization escape variants. Mechanistic studies showed that these antibodies neutralize in part by inhibiting a post-attachment step in the infection cycle. COV2-2676 and COV2-2489 offered protection either as prophylaxis or therapy, and Fc effector functions were required for optimal protection. Thus, natural infection induces a subset of potent NTD-specific mAbs that leverage neutralizing and Fc-mediated activities to protect against SARS-CoV-2 infection using multiple functional attributes. #1:  ジャーナル: Cell(Cambridge,Mass.) / 年: 2021 ジャーナル: Cell(Cambridge,Mass.) / 年: 2021タイトル: Neutralizing and protective human monoclonal antibodies recognizing the N-terminal domain of the SARS-CoV-2 spike protein. 著者: Suryadevara N / Shrihari S / Gilchuk P / VanBlargan LA / Binshtein E / Zost SJ / Nargi RS / Sutton RE / Winkler ES / Chen EC / Fouch ME / Davidson E / Doranz BJ / Chen RE / Shi PY / Carnahan ...著者: Suryadevara N / Shrihari S / Gilchuk P / VanBlargan LA / Binshtein E / Zost SJ / Nargi RS / Sutton RE / Winkler ES / Chen EC / Fouch ME / Davidson E / Doranz BJ / Chen RE / Shi PY / Carnahan RH / Thackray LB / Diamond MS / Crowe Jr JE | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23154.map.gz emd_23154.map.gz | 7.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23154-v30.xml emd-23154-v30.xml emd-23154.xml emd-23154.xml | 14.8 KB 14.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23154.png emd_23154.png | 40.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23154 http://ftp.pdbj.org/pub/emdb/structures/EMD-23154 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23154 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23154 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23154_validation.pdf.gz emd_23154_validation.pdf.gz | 288 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23154_full_validation.pdf.gz emd_23154_full_validation.pdf.gz | 287.6 KB | 表示 | |
| XML形式データ |  emd_23154_validation.xml.gz emd_23154_validation.xml.gz | 5.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23154 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23154 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23154 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23154 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23154.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23154.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.36 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
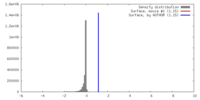
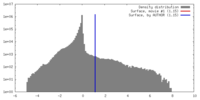
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : SARS-COV-2 spike protein (trimer) with Fab COV2-2489
| 全体 | 名称: SARS-COV-2 spike protein (trimer) with Fab COV2-2489 |
|---|---|
| 要素 |
|
-超分子 #1: SARS-COV-2 spike protein (trimer) with Fab COV2-2489
| 超分子 | 名称: SARS-COV-2 spike protein (trimer) with Fab COV2-2489 タイプ: complex / ID: 1 / 親要素: 0 |
|---|
-超分子 #2: SARS-COV-2 spike protein (trimer)
| 超分子 | 名称: SARS-COV-2 spike protein (trimer) / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: Fab COV2-2489
| 超分子 | 名称: Fab COV2-2489 / タイプ: complex / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| 染色 | タイプ: NEGATIVE / 材質: UF | |||||||||
| グリッド | 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 撮影したグリッド数: 1 / 実像数: 221 / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.2 mm 最大 デフォーカス(公称値): 1.9000000000000001 µm 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)