+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23015 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | GluK2/K5 with L-Glu | |||||||||
 マップデータ マップデータ | GluK2/K5 with L-Glu (full-length map) | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of synaptic vesicle fusion to presynaptic active zone membrane / protein retention in ER lumen / mossy fiber rosette / detection of cold stimulus involved in thermoception / Activation of Na-permeable kainate receptors / kainate selective glutamate receptor complex / Activation of Ca-permeable Kainate Receptor / negative regulation of synaptic transmission, glutamatergic / regulation of short-term neuronal synaptic plasticity / inhibitory postsynaptic potential ...regulation of synaptic vesicle fusion to presynaptic active zone membrane / protein retention in ER lumen / mossy fiber rosette / detection of cold stimulus involved in thermoception / Activation of Na-permeable kainate receptors / kainate selective glutamate receptor complex / Activation of Ca-permeable Kainate Receptor / negative regulation of synaptic transmission, glutamatergic / regulation of short-term neuronal synaptic plasticity / inhibitory postsynaptic potential / glutamate receptor activity / ubiquitin conjugating enzyme binding / receptor clustering / modulation of excitatory postsynaptic potential / extracellularly glutamate-gated ion channel activity / regulation of JNK cascade / kainate selective glutamate receptor activity / ionotropic glutamate receptor complex / behavioral fear response / neuronal action potential / positive regulation of synaptic transmission / glutamate-gated receptor activity / glutamate-gated calcium ion channel activity / ligand-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / presynaptic modulation of chemical synaptic transmission / dendrite cytoplasm / hippocampal mossy fiber to CA3 synapse / bioluminescence / regulation of membrane potential / SNARE binding / excitatory postsynaptic potential / generation of precursor metabolites and energy / synaptic transmission, glutamatergic / establishment of localization in cell / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / PDZ domain binding / cellular response to glucose stimulus / postsynaptic density membrane / regulation of long-term neuronal synaptic plasticity / modulation of chemical synaptic transmission / terminal bouton / SH3 domain binding / intracellular calcium ion homeostasis / positive regulation of neuron apoptotic process / presynaptic membrane / scaffold protein binding / chemical synaptic transmission / perikaryon / postsynaptic membrane / neuron apoptotic process / negative regulation of neuron apoptotic process / postsynaptic density / axon / neuronal cell body / glutamatergic synapse / dendrite / ubiquitin protein ligase binding / synapse / endoplasmic reticulum / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
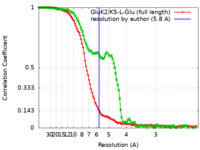
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.8 Å | |||||||||
 データ登録者 データ登録者 | Khanra N / Brown PMGE / Perozzo AM / Bowie D / Meyerson JR | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: Architecture and structural dynamics of the heteromeric GluK2/K5 kainate receptor. 著者: Nandish Khanra / Patricia Mge Brown / Amanda M Perozzo / Derek Bowie / Joel R Meyerson /   要旨: Kainate receptors (KARs) are L-glutamate-gated ion channels that regulate synaptic transmission and modulate neuronal circuits. KARs have strict assembly rules and primarily function as heteromeric ...Kainate receptors (KARs) are L-glutamate-gated ion channels that regulate synaptic transmission and modulate neuronal circuits. KARs have strict assembly rules and primarily function as heteromeric receptors in the brain. A longstanding question is how KAR heteromer subunits organize and coordinate together to fulfill their signature physiological roles. Here we report structures of the GluK2/GluK5 heteromer in apo, antagonist-bound, and desensitized states. The receptor assembles with two copies of each subunit, ligand binding domains arranged as two heterodimers and GluK5 subunits proximal to the channel. Strikingly, during desensitization, GluK2, but not GluK5, subunits undergo major structural rearrangements to facilitate channel closure. We show how the large conformational differences between antagonist-bound and desensitized states are mediated by the linkers connecting the pore helices to the ligand binding domains. This work presents the first KAR heteromer structure, reveals how its subunits are organized, and resolves how the heteromer can accommodate functionally distinct closed channel structures. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23015.map.gz emd_23015.map.gz | 117.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23015-v30.xml emd-23015-v30.xml emd-23015.xml emd-23015.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23015_fsc.xml emd_23015_fsc.xml emd_23015_fsc_2.xml emd_23015_fsc_2.xml | 11.5 KB 11.2 KB | 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23015.png emd_23015.png | 56.8 KB | ||
| その他 |  emd_23015_additional_1.map.gz emd_23015_additional_1.map.gz emd_23015_additional_2.map.gz emd_23015_additional_2.map.gz | 96.9 MB 88.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23015 http://ftp.pdbj.org/pub/emdb/structures/EMD-23015 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23015 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23015 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23015_validation.pdf.gz emd_23015_validation.pdf.gz | 465.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23015_full_validation.pdf.gz emd_23015_full_validation.pdf.gz | 465.4 KB | 表示 | |
| XML形式データ |  emd_23015_validation.xml.gz emd_23015_validation.xml.gz | 12.2 KB | 表示 | |
| CIF形式データ |  emd_23015_validation.cif.gz emd_23015_validation.cif.gz | 16.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23015 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23015 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23015 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23015 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ks3MC  7ks0C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10660 (タイトル: GluK2/K5 with L-Glu / Data size: 3.4 TB / Data #1: GluK2/K5 with L-Glu [micrographs - multiframe] / Data #2: GluK2/K5 with L-Glu [micrographs - multiframe] EMPIAR-10660 (タイトル: GluK2/K5 with L-Glu / Data size: 3.4 TB / Data #1: GluK2/K5 with L-Glu [micrographs - multiframe] / Data #2: GluK2/K5 with L-Glu [micrographs - multiframe]Data #3: GluK2/K5 with L-Glu [picked particles - single frame - processed] Data #4: GluK2/K5 with L-Glu [picked particles - single frame - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23015.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23015.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GluK2/K5 with L-Glu (full-length map) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.096 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: GluK2/K5 with L-Glu (ATD map)
| ファイル | emd_23015_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GluK2/K5 with L-Glu (ATD map) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: GluK2/K5 with L-Glu (LBD-TMD map)
| ファイル | emd_23015_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GluK2/K5 with L-Glu (LBD-TMD map) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GluK2/K5 with L-Glu
| 全体 | 名称: GluK2/K5 with L-Glu |
|---|---|
| 要素 |
|
-超分子 #1: GluK2/K5 with L-Glu
| 超分子 | 名称: GluK2/K5 with L-Glu / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293S GnTI- Homo sapiens (ヒト) / 組換細胞: HEK293S GnTI- |
-分子 #1: Glutamate receptor ionotropic, kainate 5,Green fluorescent protei...
| 分子 | 名称: Glutamate receptor ionotropic, kainate 5,Green fluorescent protein chimera タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 123.676812 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MPAELLLLLI VAFANPSCQV LSSLRMAAIL DDQTVCGRGE RLALALAREQ INGIIEVPAK ARVEVDIFEL QRDSQYETTD TMCQILPKG VVSVLGPSSS PASASTVSHI CGEKEIPHIK VGPEETPRLQ YLRFASVSLY PSNEDVSLAV SRILKSFNYP S ASLICAKA ...文字列: MPAELLLLLI VAFANPSCQV LSSLRMAAIL DDQTVCGRGE RLALALAREQ INGIIEVPAK ARVEVDIFEL QRDSQYETTD TMCQILPKG VVSVLGPSSS PASASTVSHI CGEKEIPHIK VGPEETPRLQ YLRFASVSLY PSNEDVSLAV SRILKSFNYP S ASLICAKA ECLLRLEELV RGFLISKETL SVRMLDDSRD PTPLLKEIRD DKVSTIIIDA NASISHLVLR KASELGMTSA FY KYILTTM DFPILHLDGI VEDSSNILGF SMFNTSHPFY PEFVRSLNMS WRENCEASTY PGPALSAALM FDAVHVVVSA VRE LNRSQE IGVKPLACTS ANIWPHGTSL MNYLRMVEYD GLTGRVEFNS KGQRTNYTLR ILEKSRQGHR EIGVWYSNRT LAMN ATTLD INLSQTLANK TLVVTTILEN PYVMRRPNFQ ALSGNERFEG FCVDMLRELA ELLRFRYRLR LVEDGLYGAP EPNGS WTGM VGELINRKAD LAVAAFTITA EREKVIDFSK PFMTLGISIL YRVHMGRKPG YFSFLDPFSP AVWLFMLLAY LAVSVV LFL AARLSPYEWY NPHPSLRARP HILENQYTLG NSLWFPVGGF MQQGSEIMPR ALSTRIVSGV WWAFTLIIIS SYTANLA AF LTVQRMEVPV ESADDLADQT NIEYGTIHAG STMTFFQNSR YQTYQRMWNY MQSKQPSVFV KSTEEGIARV LNSRYAFL L ESTMNEYHRR LNCNLTQIGG LLDTKGYGIG MPLGSPFRDE ITLAILQLQE NNRLEILKRK WWEGGRCPKE EDHRAKGLG MENIGGIFVV LIAGLIIAVF VAVMEFIYKS RAEAKRMKGL VPRGSAAAAM VSKGEELFTG VVPILVELDG DVNGHKFSVS GEGEGDATY GKLTLKFICT TGKLPVPWPT LVTTLTYGVQ CFSRYPDHMK QHDFFKSAMP EGYVQERTIF FKDDGNYKTR A EVKFEGDT LVNRIELKGI DFKEDGNILG HKLEYNYNSH NVYIMADKQK NGIKVNFKIR HNIEDGSVQL ADHYQQNTPI GD GPVLLPD NHYLSTQSKL SKDPNEKRDH MVLLEFVTAA GITLGMDELY KSGLRTETSQ VAPA |
-分子 #2: Glutamate receptor ionotropic, kainate 2
| 分子 | 名称: Glutamate receptor ionotropic, kainate 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 105.981617 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKIISPVLSN LVFSRSIKVL LCLLWIGYSQ GTTHVLRFGG IFEYVESGPM GAEELAFRFA VNTINRNRTL LPNTTLTYDT QKINLYDSF EASKKACDQL SLGVAAIFGP SHSSSANAVQ SICNALGVPH IQTRWKHQVS DNKDSFYVSL YPDFSSLSRA I LDLVQFFK ...文字列: MKIISPVLSN LVFSRSIKVL LCLLWIGYSQ GTTHVLRFGG IFEYVESGPM GAEELAFRFA VNTINRNRTL LPNTTLTYDT QKINLYDSF EASKKACDQL SLGVAAIFGP SHSSSANAVQ SICNALGVPH IQTRWKHQVS DNKDSFYVSL YPDFSSLSRA I LDLVQFFK WKTVTVVYDD STGLIRLQEL IKAPSRYNLR LKIRQLPADT KDAKPLLKEM KRGKEFHVIF DCSHEMAAGI LK QALAMGM MTEYYHYIFT TLDLFALDVE PYRYSGVNMT GFRILNTENT QVSSIIEKWS MERLQAPPKP DSGLLDGFMT TDA ALMYDA VHVVSVAVQQ FPQMTVSSLQ CNRHKPWRFG TRFMSLIKEA HWEGLTGRIT FNKTNGLRTD FDLDVISLKE EGLE KIGTW DPASGLNMTE SQKGKPANIT DSLSNRSLIV TTILEEPYVL FKKSDKPLYG NDRFEGYCID LLRELSTILG FTYEI RLVE DGKYGAQDDV NGQWNGMVRE LIDHKADLAV APLAITYVRE KVIDFSKPFM TLGISILYRK PNGTNPGVFS FLNPLS PDI WMYVLLAYLG VSVVLFVIAR FSPYEWYNPH PSNPDSDVVE NNFTLLNSFW FGVGALMQQG SELMPKALST RIVGGIW WF FTLIIISSYT ANLAAFLTVE RMESPIDSAD DLAKQTKIEY GAVEDGATMT FFKKSKISTY DKMWAFMSSR RQSVLVKS N EEGIQRVLTS DYAFLMESTT IEFVTQRNCN LTQIGGLIDS KGYGVGTPMG SPYRDKITIA ILQLQEEGKL HMMKEKWWR GNGCPEEESK EASALGVQNI GGIFIVLAAG LVLSVFVAVG EFLYKSKKNA QLEKRSFCSA MVEELRMSLK CQRRLKHKPQ APVIVKTEE VINMHTFNDR RLPGKETMAS GLRSAWSHPQ FEKGGGSGGG SGGGSWSHPQ FEK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.7 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV | |||||||||
| 詳細 | 1 mM L-Glu |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 51.5 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)