+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22804 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of SRR2899884.46167H+MEDI8852L fab in complex with Victoria HA | |||||||||||||||
 マップデータ マップデータ | Sharpened Map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / host cell surface receptor binding / apical plasma membrane / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Influenza A virus (A型インフルエンザウイルス) / Influenza A virus (A型インフルエンザウイルス) /   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.41 Å | |||||||||||||||
 データ登録者 データ登録者 | Gorman J / Kwong PD | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Front Immunol / 年: 2021 ジャーナル: Front Immunol / 年: 2021タイトル: Sequence-Signature Optimization Enables Improved Identification of Human HV6-1-Derived Class Antibodies That Neutralize Diverse Influenza A Viruses. 著者: Gwo-Yu Chuang / Chen-Hsiang Shen / Crystal Sao-Fong Cheung / Jason Gorman / Adrian Creanga / M Gordon Joyce / Kwanyee Leung / Reda Rawi / Lingshu Wang / Eun Sung Yang / Yongping Yang / ...著者: Gwo-Yu Chuang / Chen-Hsiang Shen / Crystal Sao-Fong Cheung / Jason Gorman / Adrian Creanga / M Gordon Joyce / Kwanyee Leung / Reda Rawi / Lingshu Wang / Eun Sung Yang / Yongping Yang / Baoshan Zhang / Yi Zhang / Masaru Kanekiyo / Tongqing Zhou / Brandon J DeKosky / Barney S Graham / John R Mascola / Peter D Kwong /  要旨: Sequence signatures of multidonor broadly neutralizing influenza antibodies can be used to quantify the prevalence of B cells with virus-neutralizing potential to accelerate development of broadly ...Sequence signatures of multidonor broadly neutralizing influenza antibodies can be used to quantify the prevalence of B cells with virus-neutralizing potential to accelerate development of broadly protective vaccine strategies. Antibodies of the same class share similar recognition modes and developmental pathways, and several antibody classes have been identified that neutralize diverse group 1- and group 2-influenza A viruses and have been observed in multiple human donors. One such multidonor antibody class, the HV6-1-derived class, targets the stem region of hemagglutinin with extraordinary neutralization breadth. Here, we use an iterative process to combine informatics, biochemical, and structural analyses to delineate an improved sequence signature for HV6-1-class antibodies. Based on sequence and structure analyses of known HV6-1 class antibodies, we derived a more inclusive signature (version 1), which we used to search for matching B-cell transcripts from published next-generation sequencing datasets of influenza vaccination studies. We expressed selected antibodies, evaluated their function, and identified amino acid-level requirements from which to refine the sequence signature (version 2). The cryo-electron microscopy structure for one of the signature-identified antibodies in complex with hemagglutinin confirmed motif recognition to be similar to known HV6-1-class members, MEDI8852 and 56.a.09, despite differences in recognition-loop length. Threading indicated the refined signature to have increased accuracy, and signature-identified heavy chains, when paired with the light chain of MEDI8852, showed neutralization comparable to the most potent members of the class. Incorporating sequences of additional class members thus enables an improved sequence signature for HV6-1-class antibodies, which can identify class members with increased accuracy. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22804.map.gz emd_22804.map.gz | 155.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22804-v30.xml emd-22804-v30.xml emd-22804.xml emd-22804.xml | 24.5 KB 24.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_22804.png emd_22804.png | 110.1 KB | ||
| マスクデータ |  emd_22804_msk_1.map emd_22804_msk_1.map | 166.4 MB |  マスクマップ マスクマップ | |
| その他 |  emd_22804_additional_1.map.gz emd_22804_additional_1.map.gz emd_22804_half_map_1.map.gz emd_22804_half_map_1.map.gz emd_22804_half_map_2.map.gz emd_22804_half_map_2.map.gz | 38.8 MB 154.6 MB 154.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22804 http://ftp.pdbj.org/pub/emdb/structures/EMD-22804 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22804 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22804 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22804_validation.pdf.gz emd_22804_validation.pdf.gz | 873.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22804_full_validation.pdf.gz emd_22804_full_validation.pdf.gz | 872.9 KB | 表示 | |
| XML形式データ |  emd_22804_validation.xml.gz emd_22804_validation.xml.gz | 15.4 KB | 表示 | |
| CIF形式データ |  emd_22804_validation.cif.gz emd_22804_validation.cif.gz | 17.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22804 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22804 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22804 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22804 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22804.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22804.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened Map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07325 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22804_msk_1.map emd_22804_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
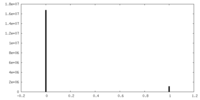
| 投影像・断面図 |
| ||||||||||||
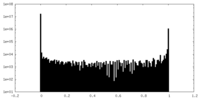
| 密度ヒストグラム |
-追加マップ: Unsharpened Map
| ファイル | emd_22804_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened Map | ||||||||||||
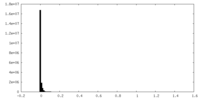
| 投影像・断面図 |
| ||||||||||||
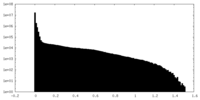
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_22804_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_22804_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of SRR2899884.46167H+MEDI8852L fab in complex w...
| 全体 | 名称: Cryo-EM structure of SRR2899884.46167H+MEDI8852L fab in complex with Victoria HA |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of SRR2899884.46167H+MEDI8852L fab in complex w...
| 超分子 | 名称: Cryo-EM structure of SRR2899884.46167H+MEDI8852L fab in complex with Victoria HA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|
-超分子 #2: Cryo-EM structure of SRR2899884.46167H+MEDI8852L fab in complex w...
| 超分子 | 名称: Cryo-EM structure of SRR2899884.46167H+MEDI8852L fab in complex with Victoria HA タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Hemagglutinin
| 分子 | 名称: Hemagglutinin / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
| 分子量 | 理論値: 38.515629 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKTIIALSHI LCLVFAQKLP GNDNSTATLC LGHHAVPNGT IVKTITNDQI EVTNATELVQ NSSIGEICDS PHQILDGENC TLIDALLGD PQCDGFQNKK WDLFVERSKA YSNCYPYDVP DYASLRSLVA SSGTLEFNNE SFNWTGVTQN GTSSACIRRS N NSFFSRLN ...文字列: MKTIIALSHI LCLVFAQKLP GNDNSTATLC LGHHAVPNGT IVKTITNDQI EVTNATELVQ NSSIGEICDS PHQILDGENC TLIDALLGD PQCDGFQNKK WDLFVERSKA YSNCYPYDVP DYASLRSLVA SSGTLEFNNE SFNWTGVTQN GTSSACIRRS N NSFFSRLN WLTQLNFKYP ALNVTMPNNE QFDKLYIWGV HHPVTDKDQI FLYAQSSGRI TVSTKRSQQA VIPNIGYRPR IR NIPSRIS IYWTIVKPGD ILLINSTGNL IAPRGYFKIR SGKSSIMRSD APIGKCNSEC ITPNGSIPND KPFQNVNRIT YGA CPRYVK QSTLKLATGM RNVPEKQTR |
-分子 #2: Fusion protein of Hemagglutinin and Envelope glycoprotein
| 分子 | 名称: Fusion protein of Hemagglutinin and Envelope glycoprotein タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 25.259078 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GIFGAIAGFI ENGWEGMVDG WYGFRHQNSE GRGQAADLKS TQAAIDQING KLNRLIGKTN EKFHQIEKEF SEVEGRIQDL EKYVEDTKI DLWSYNAELL VALENQHTID LTDSEMNKLF EKTKKQLREN AEDMGNGCFK IYHKCDNACI GSIRNGTYDH D VYRDEALN ...文字列: GIFGAIAGFI ENGWEGMVDG WYGFRHQNSE GRGQAADLKS TQAAIDQING KLNRLIGKTN EKFHQIEKEF SEVEGRIQDL EKYVEDTKI DLWSYNAELL VALENQHTID LTDSEMNKLF EKTKKQLREN AEDMGNGCFK IYHKCDNACI GSIRNGTYDH D VYRDEALN NRFQIKGVSG RLVPRGSPGS GYIPEAPRDG QAYVRKDGEW VLLSTFLGHH HHHH |
-分子 #3: Fab heavy chain
| 分子 | 名称: Fab heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.292277 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QIQLQQSGPG LVKPSQTLSL TCSISGDTVT NNYAAWDWIR QSPTRGLEWL GRTFYRSKWY KEYALSVKSR LTISPDTSKN QISLQLSSV TPEDTAVYYC ARAGITIFGL ITGGLDYWGQ GSLVTVSSAS TKGPSVFPLA PSSKSTSGGT AALGCLVKDY F PEPVTVSW ...文字列: QIQLQQSGPG LVKPSQTLSL TCSISGDTVT NNYAAWDWIR QSPTRGLEWL GRTFYRSKWY KEYALSVKSR LTISPDTSKN QISLQLSSV TPEDTAVYYC ARAGITIFGL ITGGLDYWGQ GSLVTVSSAS TKGPSVFPLA PSSKSTSGGT AALGCLVKDY F PEPVTVSW NSGALTSGVH TFPAVLQSSG LYSLSSVVTV PSSSLGTQTY ICNVNHKPSN TKVDKKVEP |
-分子 #4: Fab light chain
| 分子 | 名称: Fab light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.450832 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MTQSPSSLSA SVGDRVTITC RTSQSLSSYT HWYQQKPGKA PKLLIYAASS RGSGVPSRFS GSGSGTDFTL TISSLQPEDF ATYYCQQSR TFGQGTKVEI KRTVAAPSVF IFPPSDEQLK SGTASVVCLL NNFYPREAKV QWKVDNALQS GNSQESVTEQ D SKDSTYSL ...文字列: MTQSPSSLSA SVGDRVTITC RTSQSLSSYT HWYQQKPGKA PKLLIYAASS RGSGVPSRFS GSGSGTDFTL TISSLQPEDF ATYYCQQSR TFGQGTKVEI KRTVAAPSVF IFPPSDEQLK SGTASVVCLL NNFYPREAKV QWKVDNALQS GNSQESVTEQ D SKDSTYSL SSTLTLSKAD YEKHKVYACE VTHQGLSSPV TKSFNRGE |
-分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 7 / コピー数: 15 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 式: PBS |
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 10024 / 平均露光時間: 10.0 sec. / 平均電子線量: 71.06 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X