+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2181 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron microscopy map of SOS1 antiporter | |||||||||
 マップデータ マップデータ | Reconstruction of SOS1 antiporter | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | membrane protein / plant salt tolerance / protein structure | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 25.9 Å | |||||||||
 データ登録者 データ登録者 | Nunez Ramirez R / Sanchez Barrena MJ / Villalta I / Vega JF / Pardo JM / Quintero FJ / Martinez Salazar J / Albert A | |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2012 ジャーナル: J Mol Biol / 年: 2012タイトル: Structural insights on the plant salt-overly-sensitive 1 (SOS1) Na(+)/H(+) antiporter. 著者: Rafael Núñez-Ramírez / María José Sánchez-Barrena / Irene Villalta / Juan F Vega / Jose M Pardo / Francisco J Quintero / Javier Martinez-Salazar / Armando Albert /  要旨: The Arabidopsisthaliana Na(+)/H(+) antiporter salt-overly-sensitive 1 (SOS1) is essential to maintain low intracellular levels of toxic Na(+) under salt stress. Available data show that the plant ...The Arabidopsisthaliana Na(+)/H(+) antiporter salt-overly-sensitive 1 (SOS1) is essential to maintain low intracellular levels of toxic Na(+) under salt stress. Available data show that the plant SOS2 protein kinase and its interacting activator, the SOS3 calcium-binding protein, function together in decoding calcium signals elicited by salt stress and regulating the phosphorylation state and the activity of SOS1. Molecular genetic studies have shown that the activation implies a domain reorganization of the antiporter cytosolic moiety, indicating that there is a clear relationship between function and molecular structure of the antiporter. To provide information on this issue, we have carried out in vivo and in vitro studies on the oligomerization state of SOS1. In addition, we have performed electron microscopy and single-particle reconstruction of negatively stained full-length and active SOS1. Our studies show that the protein is a homodimer that contains a membrane domain similar to that found in other antiporters of the family and an elongated, large, and structured cytosolic domain. Both the transmembrane (TM) and cytosolic moieties contribute to the dimerization of the antiporter. The close contacts between the TM and the cytosolic domains provide a link between regulation and transport activity of the antiporter. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2181.map.gz emd_2181.map.gz | 1.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2181-v30.xml emd-2181-v30.xml emd-2181.xml emd-2181.xml | 10 KB 10 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2181.tif emd_2181.tif | 2.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2181 http://ftp.pdbj.org/pub/emdb/structures/EMD-2181 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2181 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2181 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2181_validation.pdf.gz emd_2181_validation.pdf.gz | 203.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2181_full_validation.pdf.gz emd_2181_full_validation.pdf.gz | 202.3 KB | 表示 | |
| XML形式データ |  emd_2181_validation.xml.gz emd_2181_validation.xml.gz | 4.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2181 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2181 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2181 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2181 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2181.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2181.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of SOS1 antiporter | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
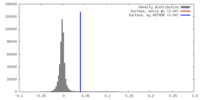
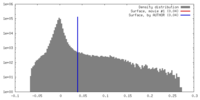
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Antiporter SOS1
| 全体 | 名称: Antiporter SOS1 |
|---|---|
| 要素 |
|
-超分子 #1000: Antiporter SOS1
| 超分子 | 名称: Antiporter SOS1 / タイプ: sample / ID: 1000 詳細: This sample is a membrane protein and is purified with lipids and detergents surrounding the membrane domain 集合状態: Dimer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 360 KDa / 理論値: 254 KDa 手法: Gel filtration, Electron microscopy image analysis, Dynamic light scattering |
-分子 #1: Na+/H+ antiporter SOS1
| 分子 | 名称: Na+/H+ antiporter SOS1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 127 KDa |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8.5 詳細: 50 mM phosphate buffer pH 8.5, 300 mM NaCl, 10% glycerol, 0.05% DDM, 250 mM imidazole |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: Purified SOS1 was adsorbed to glow-discharged carbon coated grids and stained with 2% uranyl formate |
| グリッド | 詳細: 400 mesh carbon coated copper grid, glow discharged in moderate vacuum |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2100 |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 120.000-150.000 times magnification and confirmed by inspection of the Fourier transform |
| 日付 | 2010年11月1日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ORIUS SC200 (2k x 2k) 実像数: 90 / 平均電子線量: 15 e/Å2 / ビット/ピクセル: 14 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 200 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 20000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 1.0 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 20000 |
| 試料ステージ | 試料ホルダーモデル: JEOL |
- 画像解析
画像解析
| 詳細 | Single images of SOS1 were manually extracted using EMAN. 2D reference-free classification, 2D averaging, 3D classification and reconstruction were performed using maximum-likelihood methods implemented in XMIPP package. |
|---|---|
| CTF補正 | 詳細: Each micrograph. Estimation using CTFFIND3 and correction using Bsoft |
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 25.9 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: XMIPP / 使用した粒子像数: 5600 |
| 最終 2次元分類 | クラス数: 96 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)