+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21426 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single Particle Cryo-EM Structure of the Natively Isolated Sec61 complex, TMCO1, Nicalin, TMEM147, and CCDC47 Containing Translocon | |||||||||
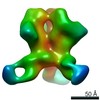
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
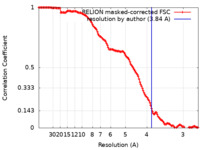
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.84 Å | |||||||||
 データ登録者 データ登録者 | McGilvray PT / Anghel SA / Sundaram A / Trnka MJ / Zhong F / Hu H / Burlingame AL / Keenan RJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: An ER translocon for multi-pass membrane protein biogenesis. 著者: Philip T McGilvray / S Andrei Anghel / Arunkumar Sundaram / Frank Zhong / Michael J Trnka / James R Fuller / Hong Hu / Alma L Burlingame / Robert J Keenan /  要旨: Membrane proteins with multiple transmembrane domains play critical roles in cell physiology, but little is known about the machinery coordinating their biogenesis at the endoplasmic reticulum. Here ...Membrane proteins with multiple transmembrane domains play critical roles in cell physiology, but little is known about the machinery coordinating their biogenesis at the endoplasmic reticulum. Here we describe a ~ 360 kDa ribosome-associated complex comprising the core Sec61 channel and five accessory factors: TMCO1, CCDC47 and the Nicalin-TMEM147-NOMO complex. Cryo-electron microscopy reveals a large assembly at the ribosome exit tunnel organized around a central membrane cavity. Similar to protein-conducting channels that facilitate movement of transmembrane segments, cytosolic and luminal funnels in TMCO1 and TMEM147, respectively, suggest routes into the central membrane cavity. High-throughput mRNA sequencing shows selective translocon engagement with hundreds of different multi-pass membrane proteins. Consistent with a role in multi-pass membrane protein biogenesis, cells lacking different accessory components show reduced levels of one such client, the glutamate transporter EAAT1. These results identify a new human translocon and provide a molecular framework for understanding its role in multi-pass membrane protein biogenesis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21426.map.gz emd_21426.map.gz | 176 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21426-v30.xml emd-21426-v30.xml emd-21426.xml emd-21426.xml | 22.9 KB 22.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21426_fsc.xml emd_21426_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21426.png emd_21426.png | 75.3 KB | ||
| マスクデータ |  emd_21426_msk_1.map emd_21426_msk_1.map | 476.8 MB |  マスクマップ マスクマップ | |
| その他 |  emd_21426_half_map_1.map.gz emd_21426_half_map_1.map.gz emd_21426_half_map_2.map.gz emd_21426_half_map_2.map.gz | 175.3 MB 174.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21426 http://ftp.pdbj.org/pub/emdb/structures/EMD-21426 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21426 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21426 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21426_validation.pdf.gz emd_21426_validation.pdf.gz | 78.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21426_full_validation.pdf.gz emd_21426_full_validation.pdf.gz | 77.6 KB | 表示 | |
| XML形式データ |  emd_21426_validation.xml.gz emd_21426_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21426 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21426 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21426 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21426 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21426.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21426.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.36 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
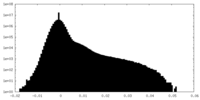
| ファイル |  emd_21426_msk_1.map emd_21426_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
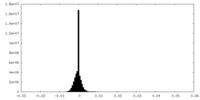
| ファイル | emd_21426_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
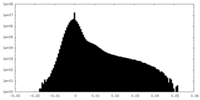
| ファイル | emd_21426_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Native complex of a translating ribosome and a novel translocon c...
+超分子 #1: Native complex of a translating ribosome and a novel translocon c...
+超分子 #2: Translocon
+超分子 #3: Sec61 Complex
+超分子 #4: TMCO1
+超分子 #5: NOMO-Nicalin-TMEM147 Complex
+超分子 #6: CCDC47
+超分子 #7: Sec61 alpha
+超分子 #8: Sec61 beta
+超分子 #9: Sec61 gamma
+超分子 #10: TMEM147
+超分子 #11: Nicalin
+超分子 #12: NOMO
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 2.0 nm / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV 詳細: Two filter papers were added to each arm, 2.5 microliters of sample were added to grids, samples were blotted for 11 seconds, and 0.5 second of drain time was allowed before vitrification.. | |||||||||||||||
| 詳細 | Sample was well-dispersed on a thin (~2 nm) carbon film. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 5562 / 平均露光時間: 3.8 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X