+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21185 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | De novo designed tetrahedral nanoparticle T33_dn10 displaying 4 copies of BG505-SOSIP trimer on the surface | |||||||||
 マップデータ マップデータ | De novo designed tetrahedral nanoparticle T33_dn10 presenting BG505-SOSIP, CryoEM Map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Nanoparticle / Rosetta / De novo protein design / HIV Env / DE NOVO PROTEIN | |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.25 Å | |||||||||
 データ登録者 データ登録者 | Antanasijevic A / Ward AB | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2020 ジャーナル: PLoS Pathog / 年: 2020タイトル: Structural and functional evaluation of de novo-designed, two-component nanoparticle carriers for HIV Env trimer immunogens. 著者: Aleksandar Antanasijevic / George Ueda / Philip J M Brouwer / Jeffrey Copps / Deli Huang / Joel D Allen / Christopher A Cottrell / Anila Yasmeen / Leigh M Sewall / Ilja Bontjer / Thomas J ...著者: Aleksandar Antanasijevic / George Ueda / Philip J M Brouwer / Jeffrey Copps / Deli Huang / Joel D Allen / Christopher A Cottrell / Anila Yasmeen / Leigh M Sewall / Ilja Bontjer / Thomas J Ketas / Hannah L Turner / Zachary T Berndsen / David C Montefiori / Per Johan Klasse / Max Crispin / David Nemazee / John P Moore / Rogier W Sanders / Neil P King / David Baker / Andrew B Ward /    要旨: Two-component, self-assembling nanoparticles represent a versatile platform for multivalent presentation of viral antigens. Computational design of protein nanoparticles with differing sizes and ...Two-component, self-assembling nanoparticles represent a versatile platform for multivalent presentation of viral antigens. Computational design of protein nanoparticles with differing sizes and geometries enables combination with antigens of choice to test novel multimerization concepts in immunization strategies where the goal is to improve the induction and maturation of neutralizing antibody lineages. Here, we describe detailed antigenic, structural, and functional characterization of computationally designed tetrahedral, octahedral, and icosahedral nanoparticle immunogens displaying trimeric HIV envelope glycoprotein (Env) ectodomains. Env trimers, based on subtype A (BG505) or consensus group M (ConM) sequences and engineered with SOSIP stabilizing mutations, were fused to an underlying trimeric building block of each nanoparticle. Initial screening yielded one icosahedral and two tetrahedral nanoparticle candidates, capable of presenting twenty or four copies of the Env trimer. A number of analyses, including detailed structural characterization by cryo-EM, demonstrated that the nanoparticle immunogens possessed the intended structural and antigenic properties. When the immunogenicity of ConM-SOSIP trimers presented on a two-component tetrahedral nanoparticle or as soluble proteins were compared in rabbits, the two immunogens elicited similar serum antibody binding titers against the trimer component. Neutralizing antibody titers were slightly elevated in the animals given the nanoparticle immunogen and were initially more focused to the trimer apex. Altogether, our findings indicate that tetrahedral nanoparticles can be successfully applied for presentation of HIV Env trimer immunogens; however, the optimal implementation to different immunization strategies remains to be determined. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21185.map.gz emd_21185.map.gz | 303.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21185-v30.xml emd-21185-v30.xml emd-21185.xml emd-21185.xml | 22.1 KB 22.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21185_fsc.xml emd_21185_fsc.xml | 15.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21185.png emd_21185.png | 128.1 KB | ||
| マスクデータ |  emd_21185_msk_1.map emd_21185_msk_1.map | 325 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-21185.cif.gz emd-21185.cif.gz | 7 KB | ||
| その他 |  emd_21185_half_map_1.map.gz emd_21185_half_map_1.map.gz emd_21185_half_map_2.map.gz emd_21185_half_map_2.map.gz | 258 MB 258.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21185 http://ftp.pdbj.org/pub/emdb/structures/EMD-21185 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21185 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21185 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21185_validation.pdf.gz emd_21185_validation.pdf.gz | 845 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21185_full_validation.pdf.gz emd_21185_full_validation.pdf.gz | 844.6 KB | 表示 | |
| XML形式データ |  emd_21185_validation.xml.gz emd_21185_validation.xml.gz | 22.5 KB | 表示 | |
| CIF形式データ |  emd_21185_validation.cif.gz emd_21185_validation.cif.gz | 29.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21185 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21185 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21185 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21185 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21185.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21185.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | De novo designed tetrahedral nanoparticle T33_dn10 presenting BG505-SOSIP, CryoEM Map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_21185_msk_1.map emd_21185_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
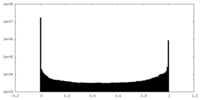
| 密度ヒストグラム |
-ハーフマップ: De novo designed tetrahedral nanoparticle T33 dn10 presenting BG505-SOSIP,...
| ファイル | emd_21185_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | De novo designed tetrahedral nanoparticle T33_dn10 presenting BG505-SOSIP, CryoEM Half-Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: De novo designed tetrahedral nanoparticle T33 dn10 presenting BG505-SOSIP,...
| ファイル | emd_21185_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | De novo designed tetrahedral nanoparticle T33_dn10 presenting BG505-SOSIP, CryoEM Half-Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : De novo designed tetrahedral nanoparticle T33_dn10 displaying 4 c...
| 全体 | 名称: De novo designed tetrahedral nanoparticle T33_dn10 displaying 4 copies of BG505-SOSIP trimer on the surface. Nanoparticle consists of 12 copies of both subunits (BG505-SOSIP-T33_dn10A and T33_dn10B) |
|---|---|
| 要素 |
|
-超分子 #1: De novo designed tetrahedral nanoparticle T33_dn10 displaying 4 c...
| 超分子 | 名称: De novo designed tetrahedral nanoparticle T33_dn10 displaying 4 copies of BG505-SOSIP trimer on the surface. Nanoparticle consists of 12 copies of both subunits (BG505-SOSIP-T33_dn10A and T33_dn10B) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Nanoparticle is assembled by combining equimolar amounts of BG505-SOSIP-T33_dn10A and T33_dn10B and subsequent incubation. Nanoparticle is purified from unassembled components by SEC |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: T33_dn10B
| 分子 | 名称: T33_dn10B / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 31.46683 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIEEVVAEMI DILAESSKKS IEELARAADN KTTEKAVAEA IEEIARLATA AIQLIEALAK NLASEEFMAR AISAIAELAK KAIEAIYRL ADNHTTDTFM ARAIAAIANL AVTAILAIAA LASNHTTEEF MARAISAIAE LAKKAIEAIY RLADNHTTDK F MAAAIEAI ...文字列: MIEEVVAEMI DILAESSKKS IEELARAADN KTTEKAVAEA IEEIARLATA AIQLIEALAK NLASEEFMAR AISAIAELAK KAIEAIYRL ADNHTTDTFM ARAIAAIANL AVTAILAIAA LASNHTTEEF MARAISAIAE LAKKAIEAIY RLADNHTTDK F MAAAIEAI ALLATLAILA IALLASNHTT EKFMARAIMA IAILAAKAIE AIYRLADNHT SPTYIEKAIE AIEKIARKAI KA IEMLAKN ITTEEYKEKA KKIIDIIRKL AKMAIKKLED NRTLEHHHHH H |
-分子 #2: BG505-SOSIP-T33_dn10A
| 分子 | 名称: BG505-SOSIP-T33_dn10A / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 88.855289 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKRGLCCVLL LCGAVFVSPS QEIHARFRRG ARAENLWVTV YYGVPVWKDA ETTLFCASDA KAYETKKHNV WATHCCVPTD PNPQEIHLE NVTEEFNMWK NNMVEQMHTD IISLWDQSLK PCVKLTPLCV TLQCTNVTNN ITDDMRGELK NCSFNMTTEL R DKKQKVYS ...文字列: MKRGLCCVLL LCGAVFVSPS QEIHARFRRG ARAENLWVTV YYGVPVWKDA ETTLFCASDA KAYETKKHNV WATHCCVPTD PNPQEIHLE NVTEEFNMWK NNMVEQMHTD IISLWDQSLK PCVKLTPLCV TLQCTNVTNN ITDDMRGELK NCSFNMTTEL R DKKQKVYS LFYRLDVVQI NENQGNRSNN SNKEYRLINC NTSAITQACP KVSFEPIPIH YCAPAGFAIL KCKDKKFNGT GP CTNVSTV QCTHGIKPVV STQLLLNGSL AEEEVIIRSE NITNNAKNIL VQLNESVQIN CTRPNNNTVK SIRIGPGQWF YYT GDIIGD IRQAHCNVSK ATWNETLGKV VKQLRKHFGN NTIIRFANSS GGDLEVTTHS FNCGGEFFYC NTSGLFNSTW ISNT SVQGS NSTGSNDSIT LPCRIKQIIN MWQRIGQAMY APPIQGVIRC VSNITGLILT RDGGSTNSTT ETFRPGGGDM RDNWR SELY KYKVVKIEPL GVAPTRCKRR VVGRRRRRRA VGIGAVSLGF LGAAGSTMGA ASMTLTVQAR NLLSGIVQQQ SNLLRA PEC QQHLLKDTHW GIKQLQARVL AVEHYLRDQQ LLGIWGCSGK LICCTNVPWN SSWSNRNLSE IWDNMTWLQW DKEISNY TQ IIYGLLEESQ NQQEKNEQGS GSGSGSGGEE AELAYLLGEL AYKLGEYRIA IRAYRIALKR DPNNAEAWYN LGNAYYKQ G DYDEAIEYYQ KALELDPNNA EAWYNLGNAY YKQGDYDEAI EYYEKALELD PENLEALQNL LNAMDKQG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.6 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: TBS buffer, pH 7.4 | |||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 11 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 10 sec. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV 詳細: Blotting time varied in the 3-7 s range, Blotting force set to 0, Wait time of 10s.. | |||||||||
| 詳細 | Nanoparticle is assembled by combining equimolar amounts of BG505-SOSIP-T33_dn10A and T33_dn10B and subsequent incubation. Nanoparticle is purified from unassembled components by SEC. Sample is then concentrated to 1.6mg/ml in TBS buffer. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 798 / 平均露光時間: 11.25 sec. / 平均電子線量: 49.39 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 36000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Model refinement was performed by iterative rounds of Rosetta relaxed refinement and manual refinement in Coot. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-6vfk: |
 ムービー
ムービー コントローラー
コントローラー





















 Z
Z Y
Y X
X