+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20956 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Portal vertex structure of bacteriophage T4 | |||||||||
 マップデータ マップデータ | low-pass filtered and B-factor sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | portal protein assembly / gp20 / gp23 / portal vertex /  VIRUS (ウイルス) VIRUS (ウイルス) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont genome ejection through host cell envelope, contractile tail mechanism / T=13 icosahedral viral capsid / viral portal complex / viral genome packaging / viral release from host cell /  カプシド / host cell plasma membrane / カプシド / host cell plasma membrane /  生体膜 生体膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Enterobacteria phage T4 (ファージ) / Enterobacteria phage T4 (ファージ) /   Escherichia virus T4 (T4ファージ) Escherichia virus T4 (T4ファージ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.5 Å クライオ電子顕微鏡法 / 解像度: 4.5 Å | |||||||||
 データ登録者 データ登録者 | Fang Q / Fokine A | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Structural morphing in a symmetry-mismatched viral vertex. 著者: Qianglin Fang / Wei-Chun Tang / Pan Tao / Marthandan Mahalingam / Andrei Fokine / Michael G Rossmann / Venigalla B Rao /  要旨: Large biological structures are assembled from smaller, often symmetric, sub-structures. However, asymmetry among sub-structures is fundamentally important for biological function. An extreme form of ...Large biological structures are assembled from smaller, often symmetric, sub-structures. However, asymmetry among sub-structures is fundamentally important for biological function. An extreme form of asymmetry, a 12-fold-symmetric dodecameric portal complex inserted into a 5-fold-symmetric capsid vertex, is found in numerous icosahedral viruses, including tailed bacteriophages, herpesviruses, and archaeal viruses. This vertex is critical for driving capsid assembly, DNA packaging, tail attachment, and genome ejection. Here, we report the near-atomic in situ structure of the symmetry-mismatched portal vertex from bacteriophage T4. Remarkably, the local structure of portal morphs to compensate for symmetry-mismatch, forming similar interactions in different capsid environments while maintaining strict symmetry in the rest of the structure. This creates a unique and unusually dynamic symmetry-mismatched vertex that is central to building an infectious virion. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20956.map.gz emd_20956.map.gz | 16.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20956-v30.xml emd-20956-v30.xml emd-20956.xml emd-20956.xml | 13 KB 13 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20956.png emd_20956.png | 275.6 KB | ||
| Filedesc metadata |  emd-20956.cif.gz emd-20956.cif.gz | 5.5 KB | ||
| その他 |  emd_20956_additional_1.map.gz emd_20956_additional_1.map.gz emd_20956_additional_2.map.gz emd_20956_additional_2.map.gz | 24.3 MB 916.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20956 http://ftp.pdbj.org/pub/emdb/structures/EMD-20956 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20956 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20956 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20956.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20956.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | low-pass filtered and B-factor sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.44 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: unfiltered map
| ファイル | emd_20956_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unfiltered map | ||||||||||||
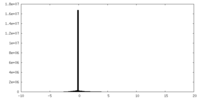
| 投影像・断面図 |
| ||||||||||||
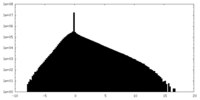
| 密度ヒストグラム |
-添付マップデータ: emd 20956 additional 2.map
| ファイル | emd_20956_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
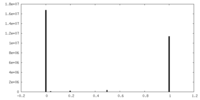
| 投影像・断面図 |
| ||||||||||||
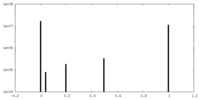
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Escherichia virus T4
| 全体 | 名称:   Escherichia virus T4 (T4ファージ) Escherichia virus T4 (T4ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Escherichia virus T4
| 超分子 | 名称: Escherichia virus T4 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 10665 / 生物種: Escherichia virus T4 / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|
-分子 #1: Major capsid protein
| 分子 | 名称: Major capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 30 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) |
| 分子量 | 理論値: 56.074242 KDa |
| 配列 | 文字列: MTIKTKAELL NKWKPLLEGE GLPEIANSKQ AIIAKIFENQ EKDFQTAPEY KDEKIAQAFG SFLTEAEIGG DHGYNATNIA AGQTSGAVT QIGPAVMGMV RRAIPNLIAF DICGVQPMNS PTGQVFALRA VYGKDPVAAG AKEAFHPMYG PDAMFSGQGA A KKFPALAA ...文字列: MTIKTKAELL NKWKPLLEGE GLPEIANSKQ AIIAKIFENQ EKDFQTAPEY KDEKIAQAFG SFLTEAEIGG DHGYNATNIA AGQTSGAVT QIGPAVMGMV RRAIPNLIAF DICGVQPMNS PTGQVFALRA VYGKDPVAAG AKEAFHPMYG PDAMFSGQGA A KKFPALAA STQTTVGDIY THFFQETGTV YLQASVQVTI DAGATDAAKL DAEIKKQMEA GALVEIAEGM ATSIAELQEG FN GSTDNPW NEMGFRIDKQ VIEAKSRQLK AAYSIELAQD LRAVHGMDAD AELSGILATE IMLEINREVV DWINYSAQVG KSG MTLTPG SKAGVFDFQD PIDIRGARWA GESFKALLFQ IDKEAVEIAR QTGRGEGNFI IASRNVVNVL ASVDTGISYA AQGL ATGFS TDTTKSVFAG VLGGKYRVYI DQYAKQDYFT VGYKGPNEMD AGIYYAPYVA LTPLRGSDPK NFQPVMGFKT RYGIG INPF AESAAQAPAS RIQSGMPSIL NSLGKNAYFR RVYVKGI UniProtKB: Major capsid protein |
-分子 #2: Portal protein
| 分子 | 名称: Portal protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) |
| 分子量 | 理論値: 61.117082 KDa |
| 配列 | 文字列: MKFNVLSLFA PWAKMDERNF KDQEKEDLVS ITAPKLDDGA REFEVSSNEA ASPYNAAFQT IFGSYEPGMK TTRELIDTYR NLMNNYEVD NAVSEIVSDA IVYEDDTEVV ALNLDKSKFS PKIKNMMLDE FSDVLNHLSF QRKGSDHFRR WYVDSRIFFH K IIDPKRPK ...文字列: MKFNVLSLFA PWAKMDERNF KDQEKEDLVS ITAPKLDDGA REFEVSSNEA ASPYNAAFQT IFGSYEPGMK TTRELIDTYR NLMNNYEVD NAVSEIVSDA IVYEDDTEVV ALNLDKSKFS PKIKNMMLDE FSDVLNHLSF QRKGSDHFRR WYVDSRIFFH K IIDPKRPK EGIKELRRLD PRQVQYVREI ITETEAGTKI VKGYKEYFIY DTAHESYACD GRMYEAGTKI KIPKAAVVYA HS GLVDCCG KNIIGYLHRA VKPANQLKLL EDAVVIYRIT RAPDRRVWYV DTGNMPARKA AEHMQHVMNT MKNRVVYDAS TGK IKNQQH NMSMTEDYWL QRRDGKAVTE VDTLPGADNT GNMEDIRWFR QALYMALRVP LSRIPQDQQG GVMFDSGTSI TRDE LTFAK FIRELQHKFE EVFLDPLKTN LLLKGIITED EWNDEINNIK IEFHRDSYFA ELKEAEILER RINMLTMAEP FIGKY ISHR TAMKDILQMT DEEIEQEAKQ IEEESKEARF QDPDQEQEDF UniProtKB: Portal protein |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 23.1 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: EMDB MAP |
|---|---|
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 4.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 53608 |
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-6uzc: |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X