+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20620 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EM structure of MPEG-1 (L425K, alpha conformation) soluble pre-pore complex | |||||||||
 マップデータ マップデータ | MPEG-1 (L425K, alpha conformation) soluble pre-pore complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | MPEG-1/perforin-2 / MACPF / macrophage / pore forming proteins / immunity / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dendritic cell antigen processing and presentation / antigen processing and presentation of exogenous peptide antigen / phagolysosome membrane / antibacterial innate immune response / wide pore channel activity / antigen processing and presentation of exogenous peptide antigen via MHC class I / phagocytic vesicle / phagocytic vesicle membrane / cytoplasmic vesicle / defense response to Gram-negative bacterium ...dendritic cell antigen processing and presentation / antigen processing and presentation of exogenous peptide antigen / phagolysosome membrane / antibacterial innate immune response / wide pore channel activity / antigen processing and presentation of exogenous peptide antigen via MHC class I / phagocytic vesicle / phagocytic vesicle membrane / cytoplasmic vesicle / defense response to Gram-negative bacterium / adaptive immune response / defense response to Gram-positive bacterium / defense response to bacterium / extracellular region 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
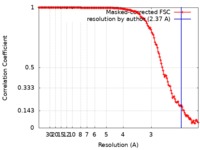
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.37 Å | |||||||||
 データ登録者 データ登録者 | Pang SS / Bayly-Jones C | |||||||||
| 資金援助 |  オーストラリア, 1件 オーストラリア, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: The cryo-EM structure of the acid activatable pore-forming immune effector Macrophage-expressed gene 1. 著者: Siew Siew Pang / Charles Bayly-Jones / Mazdak Radjainia / Bradley A Spicer / Ruby H P Law / Adrian W Hodel / Edward S Parsons / Susan M Ekkel / Paul J Conroy / Georg Ramm / Hariprasad ...著者: Siew Siew Pang / Charles Bayly-Jones / Mazdak Radjainia / Bradley A Spicer / Ruby H P Law / Adrian W Hodel / Edward S Parsons / Susan M Ekkel / Paul J Conroy / Georg Ramm / Hariprasad Venugopal / Phillip I Bird / Bart W Hoogenboom / Ilia Voskoboinik / Yann Gambin / Emma Sierecki / Michelle A Dunstone / James C Whisstock /    要旨: Macrophage-expressed gene 1 (MPEG1/Perforin-2) is a perforin-like protein that functions within the phagolysosome to damage engulfed microbes. MPEG1 is thought to form pores in target membranes, ...Macrophage-expressed gene 1 (MPEG1/Perforin-2) is a perforin-like protein that functions within the phagolysosome to damage engulfed microbes. MPEG1 is thought to form pores in target membranes, however, its mode of action remains unknown. We use cryo-Electron Microscopy (cryo-EM) to determine the 2.4 Å structure of a hexadecameric assembly of MPEG1 that displays the expected features of a soluble prepore complex. We further discover that MPEG1 prepore-like assemblies can be induced to perforate membranes through acidification, such as would occur within maturing phagolysosomes. We next solve the 3.6 Å cryo-EM structure of MPEG1 in complex with liposomes. These data reveal that a multi-vesicular body of 12 kDa (MVB12)-associated β-prism (MABP) domain binds membranes such that the pore-forming machinery of MPEG1 is oriented away from the bound membrane. This unexpected mechanism of membrane interaction suggests that MPEG1 remains bound to the phagolysosome membrane while simultaneously forming pores in engulfed bacterial targets. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20620.map.gz emd_20620.map.gz | 141.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20620-v30.xml emd-20620-v30.xml emd-20620.xml emd-20620.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20620_fsc.xml emd_20620_fsc.xml | 14 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20620.png emd_20620.png | 177.8 KB | ||
| マスクデータ |  emd_20620_msk_1.map emd_20620_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-20620.cif.gz emd-20620.cif.gz | 6 KB | ||
| その他 |  emd_20620_additional.map.gz emd_20620_additional.map.gz emd_20620_half_map_1.map.gz emd_20620_half_map_1.map.gz emd_20620_half_map_2.map.gz emd_20620_half_map_2.map.gz | 122.1 MB 226.2 MB 226.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20620 http://ftp.pdbj.org/pub/emdb/structures/EMD-20620 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20620 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20620 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20620_validation.pdf.gz emd_20620_validation.pdf.gz | 808.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20620_full_validation.pdf.gz emd_20620_full_validation.pdf.gz | 808.2 KB | 表示 | |
| XML形式データ |  emd_20620_validation.xml.gz emd_20620_validation.xml.gz | 21.7 KB | 表示 | |
| CIF形式データ |  emd_20620_validation.cif.gz emd_20620_validation.cif.gz | 28.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20620 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20620 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20620 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20620 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20620.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20620.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MPEG-1 (L425K, alpha conformation) soluble pre-pore complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_20620_msk_1.map emd_20620_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: MPEG-1 (L425K, alpha conformation) soluble pre-pore complex unsharpened...
| ファイル | emd_20620_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MPEG-1 (L425K, alpha conformation) soluble pre-pore complex unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: MPEG-1 (L425K, alpha conformation) soluble pre-pore complex, half...
| ファイル | emd_20620_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MPEG-1 (L425K, alpha conformation) soluble pre-pore complex, half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: MPEG-1 (L425K, alpha conformation) soluble pre-pore complex, half...
| ファイル | emd_20620_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MPEG-1 (L425K, alpha conformation) soluble pre-pore complex, half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MPEG-1 (L425K, alpha conformation) soluble pre-pore
| 全体 | 名称: MPEG-1 (L425K, alpha conformation) soluble pre-pore |
|---|---|
| 要素 |
|
-超分子 #1: MPEG-1 (L425K, alpha conformation) soluble pre-pore
| 超分子 | 名称: MPEG-1 (L425K, alpha conformation) soluble pre-pore / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 2.3 MDa |
-分子 #1: Macrophage-expressed gene 1 protein
| 分子 | 名称: Macrophage-expressed gene 1 protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 16 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 71.126148 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KSGKPSGEMD EVGVQKCKNA LKLPVLEVLP GGGWDNLRNV DMGRVMELTY SNCRTTEDGQ YIIPDEIFTI PQKQSNLEMN SEILESWAN YQSSTSYSIN TELSLFSKVN GKFSTEFQRM KTLQVKDQAI TTRVQVRNLV YTVKINPTLE LSSGFRKELL D ISDRLENN ...文字列: KSGKPSGEMD EVGVQKCKNA LKLPVLEVLP GGGWDNLRNV DMGRVMELTY SNCRTTEDGQ YIIPDEIFTI PQKQSNLEMN SEILESWAN YQSSTSYSIN TELSLFSKVN GKFSTEFQRM KTLQVKDQAI TTRVQVRNLV YTVKINPTLE LSSGFRKELL D ISDRLENN QTRMATYLAE LLVLNYGTHV TTSVDAGAAL IQEDHLRASF LQDSQSSRSA VTASAGLAFQ NTVNFKFEEN YT SQNVLTK SYLSNRTNSR VQSIGGVPFY PGITLQAWQQ GITNHLVAID RSGLPLHFFI NPNMLPDLPG PLVKKVSKTV ETA VKRYYT FNTYPGCTDL NSPNFNFQAN TDDGSCEGKM TNFSFGGVYQ ECTQLSGNRD VLLCQKLEQK NPLTGDFSCP SGYS PVHLL SQIHEEGYNH LECHRKCTLK VFCKTVCEDV FQVAKAEFRA FWCVASSQVP ENSGLLFGGL FSSKSINPMT NAQSC PAGY FPLRLFENLK VCVSQDYELG SRFAVPFGGF FSCTVGNPLV DPAISRDLGA PSLKKCPGGF SQHPALISDG CQVSYC VKS GLFTGGSLPP ARLPPFTRPP LMSQAATNTV IVTNSENARS WIKDSQTHQW RLGEPIELRR AMNVIHGDGG GLSHHHH HH UniProtKB: Macrophage-expressed gene 1 protein |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 16 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 |
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 44.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)