+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20446 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of AdnA(D934A)-AdnB(D1014A) in complex with AMPPNP and DNA | |||||||||
 マップデータ マップデータ | AdnAB-D934A-D1014A mutant in complex with AMPPNP and DNA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA / DNA BINDING PROTEIN / DNA BINDING PROTEIN-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA helicase complex / DNA 3'-5' helicase / recombinational repair / exonuclease activity / 3'-5' DNA helicase activity / DNA helicase activity / DNA helicase / hydrolase activity / DNA repair / DNA binding ...DNA helicase complex / DNA 3'-5' helicase / recombinational repair / exonuclease activity / 3'-5' DNA helicase activity / DNA helicase activity / DNA helicase / hydrolase activity / DNA repair / DNA binding / ATP binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Mycolicibacterium smegmatis (バクテリア) / Mycolicibacterium smegmatis (バクテリア) /   Mycobacterium smegmatis (バクテリア) Mycobacterium smegmatis (バクテリア) | |||||||||
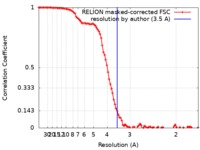
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Jia N / Unciuleac M / Shuman S / Patel DJ | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2019 ジャーナル: Proc Natl Acad Sci U S A / 年: 2019タイトル: Structures and single-molecule analysis of bacterial motor nuclease AdnAB illuminate the mechanism of DNA double-strand break resection. 著者: Ning Jia / Mihaela C Unciuleac / Chaoyou Xue / Eric C Greene / Dinshaw J Patel / Stewart Shuman /  要旨: Mycobacterial AdnAB is a heterodimeric helicase-nuclease that initiates homologous recombination by resecting DNA double-strand breaks (DSBs). The AdnA and AdnB subunits are each composed of an N- ...Mycobacterial AdnAB is a heterodimeric helicase-nuclease that initiates homologous recombination by resecting DNA double-strand breaks (DSBs). The AdnA and AdnB subunits are each composed of an N-terminal motor domain and a C-terminal nuclease domain. Here we report cryoelectron microscopy (cryo-EM) structures of AdnAB in three functional states: in the absence of DNA and in complex with forked duplex DNAs before and after cleavage of the 5' single-strand DNA (ssDNA) tail by the AdnA nuclease. The structures reveal the path of the 5' ssDNA through the AdnA nuclease domain and the mechanism of 5' strand cleavage; the path of the 3' tracking strand through the AdnB motor and the DNA contacts that couple ATP hydrolysis to mechanical work; the position of the AdnA iron-sulfur cluster subdomain at the Y junction and its likely role in maintaining the split trajectories of the unwound 5' and 3' strands. Single-molecule DNA curtain analysis of DSB resection reveals that AdnAB is highly processive but prone to spontaneous pausing at random sites on duplex DNA. A striking property of AdnAB is that the velocity of DSB resection slows after the enzyme experiences a spontaneous pause. Our results highlight shared as well as distinctive properties of AdnAB vis-à-vis the RecBCD and AddAB clades of bacterial DSB-resecting motor nucleases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20446.map.gz emd_20446.map.gz | 7.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20446-v30.xml emd-20446-v30.xml emd-20446.xml emd-20446.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20446_fsc.xml emd_20446_fsc.xml | 10 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20446.png emd_20446.png | 58.2 KB | ||
| Filedesc metadata |  emd-20446.cif.gz emd-20446.cif.gz | 6.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20446 http://ftp.pdbj.org/pub/emdb/structures/EMD-20446 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20446 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20446 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20446_validation.pdf.gz emd_20446_validation.pdf.gz | 407 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20446_full_validation.pdf.gz emd_20446_full_validation.pdf.gz | 406.6 KB | 表示 | |
| XML形式データ |  emd_20446_validation.xml.gz emd_20446_validation.xml.gz | 11.1 KB | 表示 | |
| CIF形式データ |  emd_20446_validation.cif.gz emd_20446_validation.cif.gz | 14.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20446 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20446 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20446 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20446 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20446.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20446.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | AdnAB-D934A-D1014A mutant in complex with AMPPNP and DNA | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8613 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : AdnAB-D934A-D1014A mutant in complex with AMPPNP and DNA
| 全体 | 名称: AdnAB-D934A-D1014A mutant in complex with AMPPNP and DNA |
|---|---|
| 要素 |
|
-超分子 #1: AdnAB-D934A-D1014A mutant in complex with AMPPNP and DNA
| 超分子 | 名称: AdnAB-D934A-D1014A mutant in complex with AMPPNP and DNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 理論値: 200 KDa |
-超分子 #2: UvrD/REP helicase
| 超分子 | 名称: UvrD/REP helicase / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア) |
-超分子 #3: ATP-dependent DNA helicase (UvrD/REP)
| 超分子 | 名称: ATP-dependent DNA helicase (UvrD/REP) / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア) |
-超分子 #4: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: UvrD/REP helicase
| 分子 | 名称: UvrD/REP helicase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium smegmatis (バクテリア) Mycobacterium smegmatis (バクテリア) |
| 分子量 | 理論値: 118.084531 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTQVASPVVQ ARYSPVELSA ALGLFPPTDE QAAVIAAPPG PLVVIAGAGA GKTETMAARV VWLVANGFAT PSQVLGLTFT RKAAGQLLR RVRTRLARLA GAGLAPGSGA SDESATVSTY HAFAGTLLRE HGLLLPVEPD TRLLSETELW QLAYDVVCAH P GHLDTEKT ...文字列: MTQVASPVVQ ARYSPVELSA ALGLFPPTDE QAAVIAAPPG PLVVIAGAGA GKTETMAARV VWLVANGFAT PSQVLGLTFT RKAAGQLLR RVRTRLARLA GAGLAPGSGA SDESATVSTY HAFAGTLLRE HGLLLPVEPD TRLLSETELW QLAYDVVCAH P GHLDTEKT PAAVTAMVLR LSGALAEHLV DTDQLRDTHV ELERLVHTLP AGPYQRDRGP SQWLLRMLAT QTERTELVPL ID ALHQRMR AEKVMDFGMQ MAAAARLAAR FPQVGEQLRQ RFRVVLLDEY QDTGHAQRIA LSSLFGGGAD DGLALTAVGD PIQ SIYGWR GASATNLPRF TTDFPYSDGT PAPTLELRTS WRNPPSTLHV ANAVSEEARR RSVAVRALRP RPDAEPGTIR CALL NNVAA ERDWVADHLA RAYHGAIGRG EAAPTAAVLV RRNADAAPMA EALTARGVPV EVVGVAGLLA VPEVADLVAM LRLIA DPTA GSAVMRILTG PRWRFGARDI AALWRRAVEL DDRPKGELGT ADIVAQAAPD ADTACVADAI CDPGDAERYS PAGYER IVA LGRELTMLRA HLGHPLPELV AEVRRVLGLD AEARAARPVA AGWAGTENLD RFSDLVSDFA GHAGASVSAL LAYLDAA VE VENGLAPAEL TVSHDRVQIL TVHAAKGLEW QVVAVPHLSA RVFPSTTQAR TWLTDASDLP PLLRGDRATE SEIGVPVL D TSDIYDRKIL SDKISDHKKS LDQRRVDEER RLLYVAITRA EDTLLLSGHH WGATESKPRG PSEFLCELKT ILEEATAAG TPCGEIEHWA PDPAPGETNP LRDQVVEALW PPVASADDHV HRGAQLVAAA MAGEVSAEAD QEGWAADVDA LLAERERPPQ QEDTELPGQ LSVSTLVELS RDPKAALTRL RRRLPQRPDP HALLGTTFHE WVQRYFHAER LFDLDDLPGA VDSDSGRAVE E SLAELQDA FVKSPWAART PVEVEVPFDM VLGETVVRGR IDAVFAEPDG TTMVLAWKTG DPPETPEAKE HAAVQLAVYR LA WAAMRGC PPESVRAAFH YVRSGQTVIP ETLPGAEELV KLLAAAPTET AEEADRIT UniProtKB: DNA helicase |
-分子 #2: ATP-dependent DNA helicase (UvrD/REP)
| 分子 | 名称: ATP-dependent DNA helicase (UvrD/REP) / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium smegmatis (バクテリア) Mycobacterium smegmatis (バクテリア) |
| 分子量 | 理論値: 110.899562 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTTRPAESAP QTASTLLEPG SNGVVRLLGG PGTGKSSLLV DTAVQHILAG ADPESVLLLT GSARLRTAAR AAITARLLGA GTVGVVREP LVRTVHSYAF AVLRLAAQRN GDPPPRLITS AEQDGIIREL LAGDLEDGHR SPVGWPEQLW PALTTAGFAT E LRDLMARC ...文字列: MTTRPAESAP QTASTLLEPG SNGVVRLLGG PGTGKSSLLV DTAVQHILAG ADPESVLLLT GSARLRTAAR AAITARLLGA GTVGVVREP LVRTVHSYAF AVLRLAAQRN GDPPPRLITS AEQDGIIREL LAGDLEDGHR SPVGWPEQLW PALTTAGFAT E LRDLMARC TERGVDPIAL QRLGRTAKRP EWLAAGRFAQ AYEQIMLLRS AVGMAAPQAT VPALGAAELV GAALEALGAD DE LLDTERN RIKLLLVDDA QHLDPQAARL VRALAAGTGL TVIAGDPDQS VFGYRGADPV LLRDDTHPAI TLTQSYRCAP EIA SAITGL GQRLPGVSDT RHWTGNPQRE GTVTVRLAAS THAEGTMIAD ALRRAHLVDG IPWSQMAVIV RSVPRVGTAL ARAL TAAGV PVQDNGTDVP VGRQPAAAAL LTVLDVTATG HLDADSAVAL LTGPIGRVDP VTLRQLRRAL RRADGSQPPR DFGDL LVDA IEREPKGLSA EHARTLRRLR AVLTAARRSD ASGADPRYTL WQAWHASGLQ RRWLAASERG GSVGAQADRD LDAVTT LFD VADQYVNRTA GASLRGLVDH VTRLGAAVAR TEPETAAEAV AVLSVHGALA GEWDFVVIAG VQEGLWPNMI PRGGVLG TQ HLVDVLDGVA DMTDRTVSTR APLVAEERRL LMAAMGRART RVMITAVDSD TGDESLLPSP FCAEISAWAT EPVAEPPL V APRVLAPSAL VGRLRAVVCA PDGAVDDDAR ACAAAQLARL AAAGVPGADP SQWHAMTSLT TEEPLWSEPG HVVTLSPST LQMLTDCPLR WLLERHGGDD GRDVRSTVGS LVHALVSEPG KTESQLVNEL EKVWDDLPYD AKWYSDNELA RHRAMLETFT RWREDTRRQ LTEVATEIPV EGIVVEPGEN TPGVRVRGRL DRLERDEAGR LVVVALKTGK SPVTKDDAQN HAQLAMYQLA V AAGLLDDG DEPGGGKLVY LGKAGAAGAT EREQDPLTPD KRAEWLETVG EAAAATAGPR FVARVNNGCA NCPVRSSCPA QA NGDRP UniProtKB: DNA helicase |
-分子 #3: DNA (70-MER)
| 分子 | 名称: DNA (70-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア) |
| 分子量 | 理論値: 21.477703 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DC)(DT)(DA) (DA)(DT)(DG)(DC)(DG)(DA)(DG)(DC)(DA)(DC) (DT)(DG)(DC)(DT)(DA)(DT)(DT)(DC)(DC) (DC)(DT)(DA)(DG)(DC)(DA)(DG)(DT)(DG)(DC) (DT) (DC)(DG)(DC)(DA)(DT) ...文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DC)(DT)(DA) (DA)(DT)(DG)(DC)(DG)(DA)(DG)(DC)(DA)(DC) (DT)(DG)(DC)(DT)(DA)(DT)(DT)(DC)(DC) (DC)(DT)(DA)(DG)(DC)(DA)(DG)(DT)(DG)(DC) (DT) (DC)(DG)(DC)(DA)(DT)(DT)(DA)(DG) (DA)(DT)(DT)(DT)(DT)(DG)(DT)(DT)(DT)(DT) (DT)(DT) (DT)(DA)(DG)(DC)(DG)(DG)(DT) (DT)(DT)(DT) |
-分子 #4: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 4 / コピー数: 1 / 式: ANP |
|---|---|
| 分子量 | 理論値: 506.196 Da |
| Chemical component information |  ChemComp-ANP: |
-分子 #5: IRON/SULFUR CLUSTER
| 分子 | 名称: IRON/SULFUR CLUSTER / タイプ: ligand / ID: 5 / コピー数: 1 / 式: SF4 |
|---|---|
| 分子量 | 理論値: 351.64 Da |
| Chemical component information |  ChemComp-FS1: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 構成要素 - 式: Tris / 詳細: 20 mM Tris-HCl, pH 7.5, 150 mM NaCl |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 2.16 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー