+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20351 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | VC-Tn6677 multisubunit CRISPR/Cas effector, close conformation. | |||||||||
 マップデータ マップデータ | post-processed map with B-factor automatically calculated by Relion3 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR/Cas / Cascade / RNA BINDING PROTEIN / RNA BINDING PROTEIN-RNA-DNA complex | |||||||||
| 生物種 |  | |||||||||
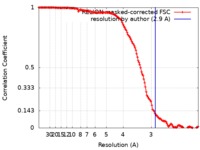
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Halpin-Healy T / Klompe S | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Structural basis of DNA targeting by a transposon-encoded CRISPR-Cas system. 著者: Tyler S Halpin-Healy / Sanne E Klompe / Samuel H Sternberg / Israel S Fernández /  要旨: Bacteria use adaptive immune systems encoded by CRISPR and Cas genes to maintain genomic integrity when challenged by pathogens and mobile genetic elements. Type I CRISPR-Cas systems typically ...Bacteria use adaptive immune systems encoded by CRISPR and Cas genes to maintain genomic integrity when challenged by pathogens and mobile genetic elements. Type I CRISPR-Cas systems typically target foreign DNA for degradation via joint action of the ribonucleoprotein complex Cascade and the helicase-nuclease Cas3, but nuclease-deficient type I systems lacking Cas3 have been repurposed for RNA-guided transposition by bacterial Tn7-like transposons. How CRISPR- and transposon-associated machineries collaborate during DNA targeting and insertion remains unknown. Here we describe structures of a TniQ-Cascade complex encoded by the Vibrio cholerae Tn6677 transposon using cryo-electron microscopy, revealing the mechanistic basis of this functional coupling. The cryo-electron microscopy maps enabled de novo modelling and refinement of the transposition protein TniQ, which binds to the Cascade complex as a dimer in a head-to-tail configuration, at the interface formed by Cas6 and Cas7 near the 3' end of the CRISPR RNA (crRNA). The natural Cas8-Cas5 fusion protein binds the 5' crRNA handle and contacts the TniQ dimer via a flexible insertion domain. A target DNA-bound structure reveals critical interactions necessary for protospacer-adjacent motif recognition and R-loop formation. This work lays the foundation for a structural understanding of how DNA targeting by TniQ-Cascade leads to downstream recruitment of additional transposase proteins, and will guide protein engineering efforts to leverage this system for programmable DNA insertions in genome-engineering applications. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20351.map.gz emd_20351.map.gz | 228.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20351-v30.xml emd-20351-v30.xml emd-20351.xml emd-20351.xml | 26.1 KB 26.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20351_fsc.xml emd_20351_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20351.png emd_20351.png | 238.2 KB | ||
| マスクデータ |  emd_20351_msk_1.map emd_20351_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-20351.cif.gz emd-20351.cif.gz | 6.9 KB | ||
| その他 |  emd_20351_additional_1.map.gz emd_20351_additional_1.map.gz emd_20351_additional_2.map.gz emd_20351_additional_2.map.gz emd_20351_half_map_1.map.gz emd_20351_half_map_1.map.gz emd_20351_half_map_2.map.gz emd_20351_half_map_2.map.gz | 193.8 MB 16.5 MB 194.5 MB 194.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20351 http://ftp.pdbj.org/pub/emdb/structures/EMD-20351 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20351 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20351 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20351_validation.pdf.gz emd_20351_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20351_full_validation.pdf.gz emd_20351_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_20351_validation.xml.gz emd_20351_validation.xml.gz | 21.5 KB | 表示 | |
| CIF形式データ |  emd_20351_validation.cif.gz emd_20351_validation.cif.gz | 28.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20351 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20351 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20351 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20351 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20351.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20351.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | post-processed map with B-factor automatically calculated by Relion3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0605 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_20351_msk_1.map emd_20351_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
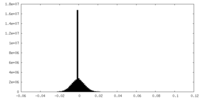
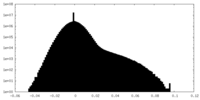
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: unsharpened map
| ファイル | emd_20351_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
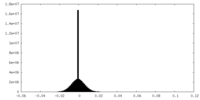
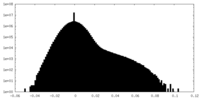
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: masked post-processed map with B-factor automatically calculated by...
| ファイル | emd_20351_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | masked post-processed map with B-factor automatically calculated by Relion3 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_20351_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_20351_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : VC-Tn667 transposon encoded CRISPR/Cas effector
| 全体 | 名称: VC-Tn667 transposon encoded CRISPR/Cas effector |
|---|---|
| 要素 |
|
-超分子 #1: VC-Tn667 transposon encoded CRISPR/Cas effector
| 超分子 | 名称: VC-Tn667 transposon encoded CRISPR/Cas effector / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 600 KDa |
-分子 #1: cas7 type I-F CRISPR-associated protein Csy3
| 分子 | 名称: cas7 type I-F CRISPR-associated protein Csy3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 39.588664 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KLPTNLAYER SIDPSDVCFF VVWPDDRKTP LTYNSRTLLG QMEAKSLAYD VSGQPIKSAT AEALAQGNPH QVDFCHVPYG ASHIECSFS VSFSSELRQP YKCNSSKVKQ TLVQLVELYE TKIGWTELAT RYLMNICNGK WLWKNTRKAY CWNIVLTPWP W NGEKVGFE ...文字列: KLPTNLAYER SIDPSDVCFF VVWPDDRKTP LTYNSRTLLG QMEAKSLAYD VSGQPIKSAT AEALAQGNPH QVDFCHVPYG ASHIECSFS VSFSSELRQP YKCNSSKVKQ TLVQLVELYE TKIGWTELAT RYLMNICNGK WLWKNTRKAY CWNIVLTPWP W NGEKVGFE DIRTNYTSRQ DFKNNKNWSA IVEMIKTAFS STDGLAIFEV RATLHLPTNA MVRPSQVFTE KEAAAAAAAA TQ NSRVFQS TTIDGERSPI LGAFKTGAAI ATIDDWYPEA TEPLRVGRFG VHREDVTCYR HPSTGKDFFS ILQQAEHYIE VLS ANKTPA QETINDMHFL MANLIKGGMF QHKGD |
-分子 #2: cas5_8 naturally occurring fusion protein
| 分子 | 名称: cas5_8 naturally occurring fusion protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 57.064711 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: LKEIIASNPD DLTTELKRAF RPLTPHIAID GNEIDALTIL VNLTDKAKCK QKLRDEKWWA SCINCVNYRQ SHNPKFPDIR SEGVIRTQA LGELPSFLLS SSKIPPYHWS YSHDSKYVNK SAFLTNEFCW DGEISCLGEL LKDADHPLWN TLKKLGCSQK T CKAMAKQL ...文字列: LKEIIASNPD DLTTELKRAF RPLTPHIAID GNEIDALTIL VNLTDKAKCK QKLRDEKWWA SCINCVNYRQ SHNPKFPDIR SEGVIRTQA LGELPSFLLS SSKIPPYHWS YSHDSKYVNK SAFLTNEFCW DGEISCLGEL LKDADHPLWN TLKKLGCSQK T CKAMAKQL ADITLTTINV TLAPNYLTQI SLPDSDTSYI SLSPVASLSM QSHFHQRLQD ENRHSAITRF SRTTNMGVTA MT CGGAFRM LKSGAKFSSP PHHRLNNGSF LVLPNIRVCG ATALSSPVTV GIPSLTAFFG FVHAFERNIN RTTSSFRVES FAI CVHQLH VEKRGLTAEF VEKGDGTISA PATRDDWQCD VVFSLILNTN FAQHIDQDTL VTSLPKRLAR GSAKIAIDDF KHIN SFSTL ETAIESLPIE AGRWLSLYAQ SNNNLSDLLA AMTEDHQLMA SCVGYHLLEE PKDKPNSLRG YKHAIAECII GLINS ITFS SETDPNTIFW SLKNYQNYLV VQPRSIN |
-分子 #3: type I-F CRISPR-associated endoribonuclease Cas6/Csy4
| 分子 | 名称: type I-F CRISPR-associated endoribonuclease Cas6/Csy4 タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 22.813025 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KWYYKTITFL PELCNNESLA AKCLRVLHGF NYQYETRNIG VSFPLWCDAT VGKKISFVSK NKIELDLLLK QHYFVQMEQL QYFHISNTV LVPEDCTYVS FRRCQSIDKL TAAGLARKIR RLEKRALSRG EAFDPSSFAQ KEHTAIAHYH SLGESSKQTN R NFRLNIRM ...文字列: KWYYKTITFL PELCNNESLA AKCLRVLHGF NYQYETRNIG VSFPLWCDAT VGKKISFVSK NKIELDLLLK QHYFVQMEQL QYFHISNTV LVPEDCTYVS FRRCQSIDKL TAAGLARKIR RLEKRALSRG EAFDPSSFAQ KEHTAIAHYH SLGESSKQTN R NFRLNIRM LSEQPREGNS IFSSYGLANS ENSFQPVPL |
-分子 #4: TniQ monomer I
| 分子 | 名称: TniQ monomer I / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 41.51716 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AMFLQRPKPY SDESLESFFI RVANKNGYGD VHRFLEATKR FLQDIDHNGY QTFPTDITRI NPYSAKNSSS ARTASFLKLA QLTFNEPPE LLGLAINRTN MKYSPSTSAV VRGAEVFPRS LLRTHSIPCC PLCLRENGYA SYLWHFQGYE YCHSHNVPLI T TCSGHEAA ...文字列: AMFLQRPKPY SDESLESFFI RVANKNGYGD VHRFLEATKR FLQDIDHNGY QTFPTDITRI NPYSAKNSSS ARTASFLKLA QLTFNEPPE LLGLAINRTN MKYSPSTSAV VRGAEVFPRS LLRTHSIPCC PLCLRENGYA SYLWHFQGYE YCHSHNVPLI T TCSGHEAA CTVSNWLAGH ESKPLPNLPK SYRWGLVHWW MGIKDSDHFS FVQFFSNWPR SFHSIIEDEV EFNLEHAVVS TS ELRLKDL LGRLFFGSIR LPERNLQHNI ILGELLCYLE NRLWQDKGLI ANLKMNALEA TVMLNCSLDQ IASMVEQRIL KPN RKSKDV TDYLFHFGDI FCLWLAEFQS DEFNRSFYVS RW |
-分子 #5: TniQ monomer 2
| 分子 | 名称: TniQ monomer 2 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 42.315031 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AMFLQRPKPY SDESLESFFI RVANKNGYGD VHRFLEATKR FLQDIDHNGY QTFPTDITRI NPYSAKNSSS ARTASFLKLA QLTFNEPPE LLGLAINRTN MKYSPSTSAV VRGAEVFPRS LLRTHSIPCC PLCLRENGYA SYLWHFQGYE YCHSHNVPLI T TCSCGKEF ...文字列: AMFLQRPKPY SDESLESFFI RVANKNGYGD VHRFLEATKR FLQDIDHNGY QTFPTDITRI NPYSAKNSSS ARTASFLKLA QLTFNEPPE LLGLAINRTN MKYSPSTSAV VRGAEVFPRS LLRTHSIPCC PLCLRENGYA SYLWHFQGYE YCHSHNVPLI T TCSCGKEF DYRVSEAACT VSNWLAGHES KPLPNLPKSY RWGLVHWWMG IKDDHFSFVQ FFSNWPRSFH SIIEDEVEFN LE HAVVSTS ELRLKDLLGR LFFGSIRLPE RNLQHNIILG ELLCYLENRL WQDKGLIANL KMNALEATVM LNCSLDQIAS MVE QRILKP NAAAAAAAAA DVTDYLFHFG DIFCLWLAAF QSDEFNRSFY VSR |
-分子 #6: guide RNA
| 分子 | 名称: guide RNA / タイプ: rna / ID: 6 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 19.237338 KDa |
| 配列 | 文字列: CUGAUAACUU ACAGGACGCU UUGGCUUCAU UGCUUUUCAG GUGAACUGCC GAGUAGGUAG |
-分子 #7: Targeting strand ssDNA
| 分子 | 名称: Targeting strand ssDNA / タイプ: dna / ID: 7 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 8.391409 KDa |
| 配列 | 文字列: (DA)(DT)(DG)(DA)(DA)(DG)(DC)(DC)(DA)(DA) (DG)(DG)(DC)(DG)(DT)(DC)(DC)(DT)(DG)(DT) (DA)(DA)(DG)(DG)(DC)(DG)(DG) |
-分子 #8: Non-targeting strand ssDNA
| 分子 | 名称: Non-targeting strand ssDNA / タイプ: dna / ID: 8 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 2.65175 KDa |
| 配列 | 文字列: (DC)(DC)(DG)(DC)(DC)(DT)(DT)(DA)(DC) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | 詳細: unspecified | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 4 K / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X