+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1cqe | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PROSTAGLANDIN H2 SYNTHASE-1 COMPLEX WITH FLURBIPROFEN | |||||||||
 要素 要素 | PROTEIN (PROSTAGLANDIN H2 SYNTHASE-1) | |||||||||
 キーワード キーワード | OXIDOREDUCTASE / DIOXYGENASE / PEROXIDASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報prostaglandin-endoperoxide synthase / prostaglandin-endoperoxide synthase activity / cyclooxygenase pathway / oxidoreductase activity, acting on single donors with incorporation of molecular oxygen, incorporation of two atoms of oxygen / prostaglandin biosynthetic process / peroxidase activity / regulation of blood pressure / response to oxidative stress / neuron projection / heme binding ...prostaglandin-endoperoxide synthase / prostaglandin-endoperoxide synthase activity / cyclooxygenase pathway / oxidoreductase activity, acting on single donors with incorporation of molecular oxygen, incorporation of two atoms of oxygen / prostaglandin biosynthetic process / peroxidase activity / regulation of blood pressure / response to oxidative stress / neuron projection / heme binding / endoplasmic reticulum membrane / protein homodimerization activity / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 |  X線回折 / X線回折 /  多重同系置換 / 解像度: 3.1 Å 多重同系置換 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Picot, D. / Loll, P.J. / Mulichak, A.M. / Garavito, R.M. | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 1994 ジャーナル: Nature / 年: 1994タイトル: The X-ray crystal structure of the membrane protein prostaglandin H2 synthase-1. 著者: Picot, D. / Loll, P.J. / Garavito, R.M. #1:  ジャーナル: Biochemistry / 年: 1996 ジャーナル: Biochemistry / 年: 1996タイトル: Synthesis and Use of Iodinated Nonsteroidal Antiinflammatory Drug Analogs as Crystallographic Probes of the Prostaglandin H2 Synthase Cyclooxygenase Active Site 著者: Loll, P.J. / Picot, D. / Ekabo, O. / Garavito, R.M. #2:  ジャーナル: Nat.Struct.Biol. / 年: 1995 ジャーナル: Nat.Struct.Biol. / 年: 1995タイトル: The Structural Basis of Aspirin Activity Inferred from the Crystal Structure of Inactivated Prostaglandin H2 Synthase 著者: Loll, P.J. / Picot, D. / Garavito, R.M. #3:  ジャーナル: J.Mol.Biol. / 年: 1992 ジャーナル: J.Mol.Biol. / 年: 1992タイトル: X-Ray Crystal Structure of Canine Myeloperoxidase at 3 Angstroms Resolution 著者: Zeng, J. / Fenna, R.E. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1cqe.cif.gz 1cqe.cif.gz | 244.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1cqe.ent.gz pdb1cqe.ent.gz | 196.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1cqe.json.gz 1cqe.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1cqe_validation.pdf.gz 1cqe_validation.pdf.gz | 1023.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1cqe_full_validation.pdf.gz 1cqe_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  1cqe_validation.xml.gz 1cqe_validation.xml.gz | 27.1 KB | 表示 | |
| CIF形式データ |  1cqe_validation.cif.gz 1cqe_validation.cif.gz | 40.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cq/1cqe https://data.pdbj.org/pub/pdb/validation_reports/cq/1cqe ftp://data.pdbj.org/pub/pdb/validation_reports/cq/1cqe ftp://data.pdbj.org/pub/pdb/validation_reports/cq/1cqe | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||
| 単位格子 |
| ||||||||||
| 非結晶学的対称性 (NCS) | NCS oper: (Code: given Matrix: (-0.99514, -0.06069, 0.0775), ベクター: |
- 要素
要素
-タンパク質 , 1種, 2分子 AB
| #1: タンパク質 | 分子量: 66595.320 Da / 分子数: 2 / 由来タイプ: 天然 詳細: PROTEIN IS GLYCOSYLATED AT RESIDUES ASN68, ASN144 AND ASN410 由来: (天然)  参照: UniProt: P05979, prostaglandin-endoperoxide synthase |
|---|
-糖 , 2種, 11分子 
| #2: 多糖 | 2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose #3: 糖 | ChemComp-BOG / |
|---|
-非ポリマー , 3種, 134分子 
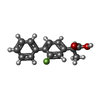



| #4: 化合物 | | #5: 化合物 | #6: 水 | ChemComp-HOH / | |
|---|
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 4 X線回折 / 使用した結晶の数: 4 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.9 Å3/Da / 溶媒含有率: 73.08 % | ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 6.7 詳細: CRYSTALLIZED BY HANGING DROP VAPOR DIFFUSION METHOD USING 1:1 RATIO OF PROTEIN SOLUTION (10 MG/ML IN 20 MM SODIUM PHOSPHATE BUFFER PH6.7, 100-200 MM NACL, 0.6% BETA-OCTYL GLUCOPYRANOSIDE, 0.1 ...詳細: CRYSTALLIZED BY HANGING DROP VAPOR DIFFUSION METHOD USING 1:1 RATIO OF PROTEIN SOLUTION (10 MG/ML IN 20 MM SODIUM PHOSPHATE BUFFER PH6.7, 100-200 MM NACL, 0.6% BETA-OCTYL GLUCOPYRANOSIDE, 0.1 MM FLURBIPORFEN) AND RESERVOIR OF 4-8% PEG 4000., VAPOR DIFFUSION, HANGING DROP | ||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 277 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: ELLIOTT GX-21 / 波長: 1.5418 回転陽極 / タイプ: ELLIOTT GX-21 / 波長: 1.5418 |
| 検出器 | 検出器: AREA DETECTOR |
| 放射 | モノクロメーター: GRAPHITE / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 3.1→20 Å / Num. obs: 32349 / % possible obs: 96 % / Observed criterion σ(I): 2 / Rmerge(I) obs: 0.076 |
| 反射 シェル | 解像度: 3.1→3.2 Å |
| 反射 | *PLUS Num. measured all: 112745 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  多重同系置換 / 解像度: 3.1→20 Å / 交差検証法: THROUGHOUT / σ(F): 2 多重同系置換 / 解像度: 3.1→20 Å / 交差検証法: THROUGHOUT / σ(F): 2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3.1→20 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 3.1→3.2 Å / Total num. of bins used: 8 /
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / バージョン: 3.1 / 分類: refinement X-PLOR / バージョン: 3.1 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj





