+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | consensus map of the V-K CRISPR-associated Transposon Integration Assembly | |||||||||
 マップデータ マップデータ | CAST V-K R1 global refinement, consensus map used as reference for composite map. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR-associated Transposon genome editing transposition / DNA BINDING PROTEIN | |||||||||
| 生物種 |  Scytonema hofmannii (バクテリア) Scytonema hofmannii (バクテリア) | |||||||||
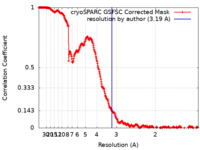
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.19 Å | |||||||||
 データ登録者 データ登録者 | Tenjo-Castano F / Mesa P / Montoya G | |||||||||
| 資金援助 |  デンマーク, European Union, 2件 デンマーク, European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2024 ジャーナル: Mol Cell / 年: 2024タイトル: Conformational landscape of the type V-K CRISPR-associated transposon integration assembly. 著者: Francisco Tenjo-Castaño / Nicholas Sofos / Luisa S Stutzke / Piero Temperini / Anders Fuglsang / Tillmann Pape / Pablo Mesa / Guillermo Montoya /  要旨: CRISPR-associated transposons (CASTs) are mobile genetic elements that co-opt CRISPR-Cas systems for RNA-guided DNA transposition. CASTs integrate large DNA cargos into the attachment (att) site ...CRISPR-associated transposons (CASTs) are mobile genetic elements that co-opt CRISPR-Cas systems for RNA-guided DNA transposition. CASTs integrate large DNA cargos into the attachment (att) site independently of homology-directed repair and thus hold promise for eukaryotic genome engineering. However, the functional diversity and complexity of CASTs hinder an understanding of their mechanisms. Here, we present the high-resolution cryoelectron microscopy (cryo-EM) structure of the reconstituted ∼1 MDa post-transposition complex of the type V-K CAST, together with different assembly intermediates and diverse TnsC filament lengths, thus enabling the recapitulation of the integration complex formation. The results of mutagenesis experiments probing the roles of specific residues and TnsB-binding sites show that transposition activity can be enhanced and suggest that the distance between the PAM and att sites is determined by the lengths of the TnsB C terminus and the TnsC filament. This singular model of RNA-guided transposition provides a foundation for repurposing the system for genome-editing applications. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19286.map.gz emd_19286.map.gz | 99.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19286-v30.xml emd-19286-v30.xml emd-19286.xml emd-19286.xml | 24.2 KB 24.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19286_fsc.xml emd_19286_fsc.xml | 28.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19286.png emd_19286.png | 75.9 KB | ||
| マスクデータ |  emd_19286_msk_1.map emd_19286_msk_1.map | 106.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-19286.cif.gz emd-19286.cif.gz | 7 KB | ||
| その他 |  emd_19286_half_map_1.map.gz emd_19286_half_map_1.map.gz emd_19286_half_map_2.map.gz emd_19286_half_map_2.map.gz | 98.6 MB 98.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19286 http://ftp.pdbj.org/pub/emdb/structures/EMD-19286 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19286 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19286 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19286_validation.pdf.gz emd_19286_validation.pdf.gz | 1010.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19286_full_validation.pdf.gz emd_19286_full_validation.pdf.gz | 1010.1 KB | 表示 | |
| XML形式データ |  emd_19286_validation.xml.gz emd_19286_validation.xml.gz | 21.1 KB | 表示 | |
| CIF形式データ |  emd_19286_validation.cif.gz emd_19286_validation.cif.gz | 31.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19286 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19286 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19286 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19286 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19286.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19286.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CAST V-K R1 global refinement, consensus map used as reference for composite map. | ||||||||||||||||||||||||||||||||||||
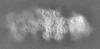
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.728 Å | ||||||||||||||||||||||||||||||||||||
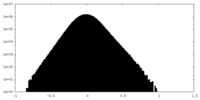
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_19286_msk_1.map emd_19286_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
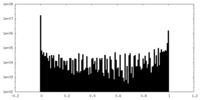
| 密度ヒストグラム |
-ハーフマップ: CAST V-K R1 global refinement, Half-A map.
| ファイル | emd_19286_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CAST V-K R1 global refinement, Half-A map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: CAST V-K R1 global refinement, Half-B map.
| ファイル | emd_19286_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CAST V-K R1 global refinement, Half-B map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Type V-K CRISPR-associated transposon post-transposition state af...
+超分子 #1: Type V-K CRISPR-associated transposon post-transposition state af...
+分子 #1: sgRNA
+分子 #2: Non-target strand - LE
+分子 #3: Target strand - LE
+分子 #4: LE
+分子 #5: RE
+分子 #6: Non-target strand - RE
+分子 #7: Target strand
+分子 #8: Cas12k
+分子 #9: S15
+分子 #10: TniQ
+分子 #11: TnsC
+分子 #12: TnsB
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: UltrAuFoil R0./1 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | プロトコル: AB INITIO MODEL |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)