+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM reconstruction of SARS-CoV-2 spike in complex with nanobody tri-TMH (partially open conformation) | ||||||||||||
 マップデータ マップデータ | Map filtered to local resolution | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | SARS-CoV-2 spike / nanobody / trimodular nanobody / VIRAL PROTEIN | ||||||||||||
| 生物種 |   | ||||||||||||
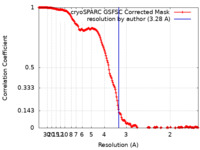
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.28 Å | ||||||||||||
 データ登録者 データ登録者 | Rissanen I / Hannula L / Huiskonen JT | ||||||||||||
| 資金援助 |  フィンランド, 3件 フィンランド, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Microbiol Spectr / 年: 2024 ジャーナル: Microbiol Spectr / 年: 2024タイトル: Nanobody engineering for SARS-CoV-2 neutralization and detection. 著者: Liina Hannula / Suvi Kuivanen / Jonathan Lasham / Ravi Kant / Lauri Kareinen / Mariia Bogacheva / Tomas Strandin / Tarja Sironen / Jussi Hepojoki / Vivek Sharma / Petri Saviranta / Anja Kipar ...著者: Liina Hannula / Suvi Kuivanen / Jonathan Lasham / Ravi Kant / Lauri Kareinen / Mariia Bogacheva / Tomas Strandin / Tarja Sironen / Jussi Hepojoki / Vivek Sharma / Petri Saviranta / Anja Kipar / Olli Vapalahti / Juha T Huiskonen / Ilona Rissanen /     要旨: In response to the ongoing COVID-19 pandemic, the quest for coronavirus inhibitors has inspired research on a variety of small proteins beyond conventional antibodies, including robust single-domain ...In response to the ongoing COVID-19 pandemic, the quest for coronavirus inhibitors has inspired research on a variety of small proteins beyond conventional antibodies, including robust single-domain antibody fragments, i.e., "nanobodies." Here, we explore the potential of nanobody engineering in the development of antivirals and diagnostic tools. Through fusion of nanobody domains that target distinct binding sites, we engineered multimodular nanobody constructs that neutralize wild-type SARS-CoV-2 and the Alpha and Delta variants at high potency, with IC values as low as 50 pM. Despite simultaneous binding to distinct epitopes, Beta and Omicron variants were more resistant to neutralization by the multimodular nanobodies, which highlights the importance of accounting for antigenic drift in the design of biologics. To further explore the applications of nanobody engineering in outbreak management, we present an assay based on fusions of nanobodies with fragments of NanoLuc luciferase that can detect sub-nanomolar quantities of the SARS-CoV-2 spike protein in a single step. Our work showcases the potential of nanobody engineering to combat emerging infectious diseases. IMPORTANCE: Nanobodies, small protein binders derived from the camelid antibody, are highly potent inhibitors of respiratory viruses that offer several advantages over conventional antibodies as ...IMPORTANCE: Nanobodies, small protein binders derived from the camelid antibody, are highly potent inhibitors of respiratory viruses that offer several advantages over conventional antibodies as candidates for specific therapies, including high stability and low production costs. In this work, we leverage the unique properties of nanobodies and apply them as building blocks for new therapeutic and diagnostic tools. We report ultra-potent SARS-CoV-2 inhibition by engineered nanobodies comprising multiple modules in structure-guided combinations and develop nanobodies that carry signal molecules, allowing rapid detection of the SARS-CoV-2 spike protein. Our results highlight the potential of engineered nanobodies in the development of effective countermeasures, both therapeutic and diagnostic, to manage outbreaks of emerging viruses. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19064.map.gz emd_19064.map.gz | 49 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19064-v30.xml emd-19064-v30.xml emd-19064.xml emd-19064.xml | 17.8 KB 17.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19064_fsc.xml emd_19064_fsc.xml | 19.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19064.png emd_19064.png | 50 KB | ||
| マスクデータ |  emd_19064_msk_1.map emd_19064_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-19064.cif.gz emd-19064.cif.gz | 6.3 KB | ||
| その他 |  emd_19064_half_map_1.map.gz emd_19064_half_map_1.map.gz emd_19064_half_map_2.map.gz emd_19064_half_map_2.map.gz | 475.6 MB 475.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19064 http://ftp.pdbj.org/pub/emdb/structures/EMD-19064 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19064 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19064 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19064_validation.pdf.gz emd_19064_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19064_full_validation.pdf.gz emd_19064_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_19064_validation.xml.gz emd_19064_validation.xml.gz | 26.3 KB | 表示 | |
| CIF形式データ |  emd_19064_validation.cif.gz emd_19064_validation.cif.gz | 34.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19064 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19064 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19064 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19064 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19064.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19064.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map filtered to local resolution | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
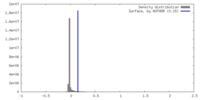
| 密度 |
| ||||||||||||||||||||||||||||||||||||
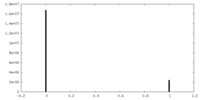
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_19064_msk_1.map emd_19064_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
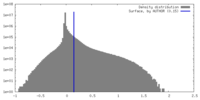
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_19064_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
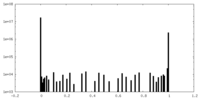
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_19064_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 spike in complex with nanobody tri-TMH
| 全体 | 名称: SARS-CoV-2 spike in complex with nanobody tri-TMH |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 spike in complex with nanobody tri-TMH
| 超分子 | 名称: SARS-CoV-2 spike in complex with nanobody tri-TMH / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: SARS-CoV-2 spike protein
| 分子 | 名称: SARS-CoV-2 spike protein / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: TGQCVNLTTR TQLPPAYTNS FTRGVYYPDK VFRSSVLHST QDLFLPFFSN VTWFHAIHVS GTNGTKRFDN PVLPFNDGVY FASTEKSNII RGWIFGTTLD SKTQSLLIVN NATNVVIKVC EFQFCNDPFL GVYYHKNNKS WMESEFRVYS SANNCTFEYV SQPFLMDLEG ...文字列: TGQCVNLTTR TQLPPAYTNS FTRGVYYPDK VFRSSVLHST QDLFLPFFSN VTWFHAIHVS GTNGTKRFDN PVLPFNDGVY FASTEKSNII RGWIFGTTLD SKTQSLLIVN NATNVVIKVC EFQFCNDPFL GVYYHKNNKS WMESEFRVYS SANNCTFEYV SQPFLMDLEG KQGNFKNLRE FVFKNIDGYF KIYSKHTPIN LVRDLPQGFS ALEPLVDLPI GINITRFQTL LALHRSYLTP GDSSSGWTAG AAAYYVGYLQ PRTFLLKYNE NGTITDAVDC ALDPLSETKC TLKSFTVEKG IYQTSNFRVQ PTESIVRFPN ITNLCPFGEV FNATRFASVY AWNRKRISNC VADYSVLYNS ASFSTFKCYG VSPTKLNDLC FTNVYADSFV IRGDEVRQIA PGQTGKIADY NYKLPDDFTG CVIAWNSNNL DSKVGGNYNY LYRLFRKSNL KPFERDISTE IYQAGSTPCN GVEGFNCYFP LQSYGFQPTN GVGYQPYRVV VLSFELLHAP ATVCGPKKST NLVKNKCVNF NFNGLTGTGV LTESNKKFLP FQQFGRDIAD TTDAVRDPQT LEILDITPCS FGGVSVITPG TNTSNQVAVL YQDVNCTEVP VAIHADQLTP TWRVYSTGSN VFQTRAGCLI GAEHVNNSYE CDIPIGAGIC ASYQTQTNSP GSASSVASQS IIAYTMSLGA ENSVAYSNNS IAIPTNFTIS VTTEILPVSM TKTSVDCTMY ICGDSTECSN LLLQYGSFCT QLNRALTGIA VEQDKNTQEV FAQVKQIYKT PPIKDFGGFN FSQILPDPSK PSKRSFIEDL LFNKVTLADA GFIKQYGDCL GDIAARDLIC AQKFNGLTVL PPLLTDEMIA QYTSALLAGT ITSGWTFGAG AALQIPFAMQ MAYRFNGIGV TQNVLYENQK LIANQFNSAI GKIQDSLSST ASALGKLQDV VNQNAQALNT LVKQLSSNFG AISSVLNDIL SRLDPPEAEV QIDRLITGRL QSLQTYVTQQ LIRAAEIRAS ANLAATKMSE CVLGQSKRVD FCGKGYHLMS FPQSAPHGVV FLHVTYVPAQ EKNFTTAPAI CHDGKAHFPR EGVFVSNGTH WFVTQRNFYE PQIITTDNTF VSGNCDVVIG IVNNTVYDPL QPELDSFKEE LDKYFKNHTS PDVDLGDISG INASVVNIQK EIDRLNEVAK NLNESLIDLQ ELGKYEQGTG YIPEAPRDGQ AYVRKDGEWV LLSTFLGSGL EVLFQGPGSG RGVPHIVMVD AYKRYKGSGH HHHHHHH |
-分子 #2: Tri-TMH multimodular nanobody
| 分子 | 名称: Tri-TMH multimodular nanobody / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRGSHHHHHH GMASMTGGQQ MGRDLYDDDD KDHPFTQVQL VETGGGLVQP GGSLRLSCAA SGFTFSSVYM NWVRQAPGKG PEWVSRISPN SGNIGYTDSV KGRFTISRDN AKNTLYLQMN NLKPEDTALY YCAIGLNLSS SSVRGQGTQV TVSSGGGGSG GGGSGGGGSG ...文字列: MRGSHHHHHH GMASMTGGQQ MGRDLYDDDD KDHPFTQVQL VETGGGLVQP GGSLRLSCAA SGFTFSSVYM NWVRQAPGKG PEWVSRISPN SGNIGYTDSV KGRFTISRDN AKNTLYLQMN NLKPEDTALY YCAIGLNLSS SSVRGQGTQV TVSSGGGGSG GGGSGGGGSG GGGSQVQLVE SGGGLVQAGG SLRLSCAASG FPVEVWRMEW YRQAPGKERE GVAAIESYGH GTRYADSVKG RFTISRDNAK NTVYLQMNSL KPEDTAVYYC NVYDDGQLAY HYDYWGQGTQ VTVSSGGGGS GGGGSGGGGS GGGGSQVQLV ESGGGLMQAG GSLRLSCAVS GRTFSTAAMG WFRQAPGKER EFVAAIRWSG GSAYYADSVK GRFTISRDKA KNTVYLQMNS LKYEDTAVYY CAQTHYVSYL LSDYATWPYD YWGQGTQVTV SS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 / 詳細: 10 mM Tris pH 8, 150 mM NaCl |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 274.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 実像数: 3073 / 平均露光時間: 5.0 sec. / 平均電子線量: 1.38 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)