+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
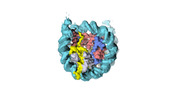
| タイトル | Human CENP-A nucleosome assembled on alpha-satellite DNA (partially unwrapped) | |||||||||
 マップデータ マップデータ | Human CENP-A nucleosome assembled on alpha-satellite DNA (partially unwrapped DNA) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Nucleosome / Centromere / centromeric / H3-like / CENP-A / Histones / Human / DNA / DNA BINDING PROTEIN | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
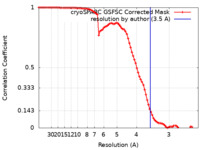
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Ali-Ahmad A / Sekulic N | |||||||||
| 資金援助 |  ノルウェー, 2件 ノルウェー, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: CENP-A and CENP-B collaborate to create an open centromeric chromatin state. 著者: Harsh Nagpal / Ahmad Ali-Ahmad / Yasuhiro Hirano / Wei Cai / Mario Halic / Tatsuo Fukagawa / Nikolina Sekulić / Beat Fierz /     要旨: Centromeres are epigenetically defined via the presence of the histone H3 variant CENP-A. Contacting CENP-A nucleosomes, the constitutive centromere associated network (CCAN) and the kinetochore ...Centromeres are epigenetically defined via the presence of the histone H3 variant CENP-A. Contacting CENP-A nucleosomes, the constitutive centromere associated network (CCAN) and the kinetochore assemble, connecting the centromere to spindle microtubules during cell division. The DNA-binding centromeric protein CENP-B is involved in maintaining centromere stability and, together with CENP-A, shapes the centromeric chromatin state. The nanoscale organization of centromeric chromatin is not well understood. Here, we use single-molecule fluorescence and cryoelectron microscopy (cryoEM) to show that CENP-A incorporation establishes a dynamic and open chromatin state. The increased dynamics of CENP-A chromatin create an opening for CENP-B DNA access. In turn, bound CENP-B further opens the chromatin fiber structure and induces nucleosomal DNA unwrapping. Finally, removal of CENP-A increases CENP-B mobility in cells. Together, our studies show that the two centromere-specific proteins collaborate to reshape chromatin structure, enabling the binding of centromeric factors and establishing a centromeric chromatin state. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18740.map.gz emd_18740.map.gz | 32.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18740-v30.xml emd-18740-v30.xml emd-18740.xml emd-18740.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18740_fsc.xml emd_18740_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18740.png emd_18740.png | 35.7 KB | ||
| マスクデータ |  emd_18740_msk_1.map emd_18740_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18740.cif.gz emd-18740.cif.gz | 5 KB | ||
| その他 |  emd_18740_half_map_1.map.gz emd_18740_half_map_1.map.gz emd_18740_half_map_2.map.gz emd_18740_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18740 http://ftp.pdbj.org/pub/emdb/structures/EMD-18740 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18740 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18740 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18740_validation.pdf.gz emd_18740_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18740_full_validation.pdf.gz emd_18740_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_18740_validation.xml.gz emd_18740_validation.xml.gz | 16.6 KB | 表示 | |
| CIF形式データ |  emd_18740_validation.cif.gz emd_18740_validation.cif.gz | 21.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18740 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18740 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18740 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18740 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18740.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18740.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human CENP-A nucleosome assembled on alpha-satellite DNA (partially unwrapped DNA) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2046 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18740_msk_1.map emd_18740_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18740_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18740_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human Cenp-A nucleosome assembled on alpha-satellite DNA (partial...
| 全体 | 名称: Human Cenp-A nucleosome assembled on alpha-satellite DNA (partially unwrapped DNA) |
|---|---|
| 要素 |
|
-超分子 #1: Human Cenp-A nucleosome assembled on alpha-satellite DNA (partial...
| 超分子 | 名称: Human Cenp-A nucleosome assembled on alpha-satellite DNA (partially unwrapped DNA) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Human histone CENP-A
| 分子 | 名称: Human histone CENP-A / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MARTKQTARK STGGKAPRKQ LATKAARKSA PATGGVKKPH RYRPGTVALR EIRKLQKSTH LLIRKLPFSR LAREICVKFT RGVDFNWQAQ ALLALQEAAE AFLVHLFEDA YLLTLHAGRV TLFPKDVQLA RRIRGLEEGL G |
-分子 #2: Human histone H4
| 分子 | 名称: Human histone H4 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGRGKGGKG LGKGGAKRHR KVLRDNIQGI TKPAIRRLAR RGGVKRISGL IYEETRGVLK VFLENVIRDA VTYTEHAKRK TVTAMDVVYA LKRQGRTLYG FGG |
-分子 #3: Human histone H2A
| 分子 | 名称: Human histone H2A / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPLGMSGRGK QGGKARAKAK SRSSRAGLQF PVGRVHRLLR KGNYAERVGA GAPVYMAAVL EYLTAEILEL AGNAARDNKK TRIIPRHLQL AIRNDEELNK LLGKVTIAQG GVLPNIQAVL LPKKTESHHK AKGK |
-分子 #4: Human histone H2B
| 分子 | 名称: Human histone H2B / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPEPAKSAPA PKKGSKKAVT KAQKKDGKKR KRSRKESYSV YVYKVLKQVH PDTGISSKAM GIMNSFVNDI FERIAGEASR LAHYNKRSTI TSREIQTAVR LLLPGELAKH AVSEGTKAVT KYTSSK |
-分子 #5: Human alpha-satellite DNA
| 分子 | 名称: Human alpha-satellite DNA / タイプ: dna / ID: 5 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GGAGGATTTC GTTGGAAACG GGATCAACTT CCCATAACTG AACGGAAGCA AACTCAGAAC ATTCTTTGTG ATGTTTGTAT TCAACTCACA GAGTTGAACC TTCCTTTGAT AGTTCAGGTT TGCAACACCC TTGTAGTAGA ATCTGCAAGT GTATATTTTG ACCACTTTGG A |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 57.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)