+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Streptococcus pneumoniae NADPH oxidase F397A mutant in complex with NADPH | |||||||||
 マップデータ マップデータ | Main map used for atomic model building and figures | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NADPH oxidase / ROS producing / flavoprotein / heme protein / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報2 iron, 2 sulfur cluster binding / flavin adenine dinucleotide binding / oxidoreductase activity / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.64 Å | |||||||||
 データ登録者 データ登録者 | Dubach VRA / San Segundo-Acosta P / Murphy BJ | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structural and mechanistic insights into Streptococcus pneumoniae NADPH oxidase. 著者: Victor R A Dubach / Pablo San Segundo-Acosta / Bonnie J Murphy /   要旨: Nicotinamide adenine dinucleotide phosphate (NADPH) oxidases (NOXs) have a major role in the physiology of eukaryotic cells by mediating reactive oxygen species production. Evolutionarily distant ...Nicotinamide adenine dinucleotide phosphate (NADPH) oxidases (NOXs) have a major role in the physiology of eukaryotic cells by mediating reactive oxygen species production. Evolutionarily distant proteins with the NOX catalytic core have been found in bacteria, including Streptococcus pneumoniae NOX (SpNOX), which is proposed as a model for studying NOXs because of its high activity and stability in detergent micelles. We present here cryo-electron microscopy structures of substrate-free and nicotinamide adenine dinucleotide (NADH)-bound SpNOX and of NADPH-bound wild-type and F397A SpNOX under turnover conditions. These high-resolution structures provide insights into the electron-transfer pathway and reveal a hydride-transfer mechanism regulated by the displacement of F397. We conducted structure-guided mutagenesis and biochemical analyses that explain the absence of substrate specificity toward NADPH and suggest the mechanism behind constitutive activity. Our study presents the structural basis underlying SpNOX enzymatic activity and sheds light on its potential in vivo function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18647.map.gz emd_18647.map.gz | 36.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18647-v30.xml emd-18647-v30.xml emd-18647.xml emd-18647.xml | 23.1 KB 23.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18647_fsc.xml emd_18647_fsc.xml | 7.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18647.png emd_18647.png | 99.1 KB | ||
| マスクデータ |  emd_18647_msk_1.map emd_18647_msk_1.map | 38.4 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18647.cif.gz emd-18647.cif.gz | 7.2 KB | ||
| その他 |  emd_18647_additional_1.map.gz emd_18647_additional_1.map.gz emd_18647_half_map_1.map.gz emd_18647_half_map_1.map.gz emd_18647_half_map_2.map.gz emd_18647_half_map_2.map.gz | 19.4 MB 35.7 MB 35.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18647 http://ftp.pdbj.org/pub/emdb/structures/EMD-18647 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18647 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18647 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18647_validation.pdf.gz emd_18647_validation.pdf.gz | 825.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18647_full_validation.pdf.gz emd_18647_full_validation.pdf.gz | 825.4 KB | 表示 | |
| XML形式データ |  emd_18647_validation.xml.gz emd_18647_validation.xml.gz | 14.7 KB | 表示 | |
| CIF形式データ |  emd_18647_validation.cif.gz emd_18647_validation.cif.gz | 18.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18647 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18647 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18647 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18647 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18647.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18647.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map used for atomic model building and figures | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1142 Å | ||||||||||||||||||||||||||||||||||||
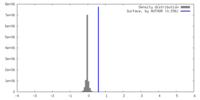
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18647_msk_1.map emd_18647_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
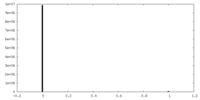
| 密度ヒストグラム |
-追加マップ: Raw unsharp map obtained after merging the half maps
| ファイル | emd_18647_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw unsharp map obtained after merging the half maps | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
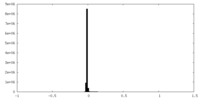
| 密度ヒストグラム |
-ハーフマップ: Half map A used during refinement and FSC...
| ファイル | emd_18647_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A used during refinement and FSC gold standard resolution estimation. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
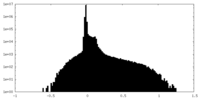
| 密度ヒストグラム |
-ハーフマップ: Half map B used during refinement and FSC...
| ファイル | emd_18647_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B used during refinement and FSC gold standard resolution estimation. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Protein-substrate complex of NADPH oxidase with NADPH
| 全体 | 名称: Protein-substrate complex of NADPH oxidase with NADPH |
|---|---|
| 要素 |
|
-超分子 #1: Protein-substrate complex of NADPH oxidase with NADPH
| 超分子 | 名称: Protein-substrate complex of NADPH oxidase with NADPH タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 46 KDa |
-分子 #1: FAD-binding FR-type domain-containing protein
| 分子 | 名称: FAD-binding FR-type domain-containing protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 45.984457 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EFSMKSVKGL LFIIASFILT LLTWMNTSPQ FMIPGLALTS LSLTFILATR LPLLESWFHS LEKVYTVHKF TAFLSIILLI FHNFSMGGL WGSRLAAQFG NLAIYIFASI ILVAYLGKYI QYEAWRWIHR LVYLAYILGL FHIYMIMGNR LLTFNLLSFL V GSYALLGL ...文字列: EFSMKSVKGL LFIIASFILT LLTWMNTSPQ FMIPGLALTS LSLTFILATR LPLLESWFHS LEKVYTVHKF TAFLSIILLI FHNFSMGGL WGSRLAAQFG NLAIYIFASI ILVAYLGKYI QYEAWRWIHR LVYLAYILGL FHIYMIMGNR LLTFNLLSFL V GSYALLGL LAGFYIIFLY QKISFPYLGK ITHLKRLNHD TREIQIHLSR PFNYQSGQFA FLKIFQEGFE SAPHPFSISG GH GQTLYFT VKTSGDHTKN IYDNLQAGSK VTLDRAYGHM IIEEGRENQV WIAGGIGITP FISYIREHPI LDKQVHFYYS FRG DENAVY LDLLRNYAQK NPNFELHLID STKDGYLNFE QKEVPEHATV YMCGPISMMK ALAKQIKKQN PKTELIYEGA KFK UniProtKB: FAD-binding FR-type domain-containing protein |
-分子 #2: NADPH DIHYDRO-NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE
| 分子 | 名称: NADPH DIHYDRO-NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE タイプ: ligand / ID: 2 / コピー数: 1 / 式: NDP |
|---|---|
| 分子量 | 理論値: 745.421 Da |
| Chemical component information |  ChemComp-NDP: |
-分子 #3: FLAVIN-ADENINE DINUCLEOTIDE
| 分子 | 名称: FLAVIN-ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: FAD |
|---|---|
| 分子量 | 理論値: 785.55 Da |
| Chemical component information |  ChemComp-FAD: |
-分子 #4: PROTOPORPHYRIN IX CONTAINING FE
| 分子 | 名称: PROTOPORPHYRIN IX CONTAINING FE / タイプ: ligand / ID: 4 / コピー数: 2 / 式: HEM |
|---|---|
| 分子量 | 理論値: 616.487 Da |
| Chemical component information |  ChemComp-HEM: |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 27 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 4 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 2 / 実像数: 45181 / 平均露光時間: 4.0 sec. / 平均電子線量: 70.0 e/Å2 詳細: Movies were collected in EER format. Hole selection was done with help from plasmon imaging (WJH Hagen, 2022). 2 grids were imaged over the course of 4 independent microscope sessions. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 215000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: experimental model 詳細: Initial model was the previousy resolved WT protein with NADPH bound |
|---|---|
| 詳細 | Initial model was fit using ChimeraX and refined with Coot and PHENIX |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: Cross-correlation coefficient |
| 得られたモデル |  PDB-8qta: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)