+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 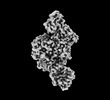 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of the apo SPARTA (BabAgo/TIR-APAZ) complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Prokaryotic Argonaute / TIR domain / RNA binding protein / DNA binding protein / IMMUNE SYSTEM | |||||||||
| 生物種 |  Bacillales bacterium (バクテリア) Bacillales bacterium (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.57 Å | |||||||||
 データ登録者 データ登録者 | Finocchio G / Koopal B / Potocnik A / Heijstek C / Jinek M / Swarts D | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2024 ジャーナル: Nucleic Acids Res / 年: 2024タイトル: Target DNA-dependent activation mechanism of the prokaryotic immune system SPARTA. 著者: Giada Finocchio / Balwina Koopal / Ana Potocnik / Clint Heijstek / Adrie H Westphal / Martin Jinek / Daan C Swarts /   要旨: In both prokaryotic and eukaryotic innate immune systems, TIR domains function as NADases that degrade the key metabolite NAD+ or generate signaling molecules. Catalytic activation of TIR domains ...In both prokaryotic and eukaryotic innate immune systems, TIR domains function as NADases that degrade the key metabolite NAD+ or generate signaling molecules. Catalytic activation of TIR domains requires oligomerization, but how this is achieved varies in distinct immune systems. In the Short prokaryotic Argonaute (pAgo)/TIR-APAZ (SPARTA) immune system, TIR NADase activity is triggered upon guide RNA-mediated recognition of invading DNA by an unknown mechanism. Here, we describe cryo-EM structures of SPARTA in the inactive monomeric and target DNA-activated tetrameric states. The monomeric SPARTA structure reveals that in the absence of target DNA, a C-terminal tail of TIR-APAZ occupies the nucleic acid binding cleft formed by the pAgo and TIR-APAZ subunits, inhibiting SPARTA activation. In the active tetrameric SPARTA complex, guide RNA-mediated target DNA binding displaces the C-terminal tail and induces conformational changes in pAgo that facilitate SPARTA-SPARTA dimerization. Concurrent release and rotation of one TIR domain allow it to form a composite NADase catalytic site with the other TIR domain within the dimer, and generate a self-complementary interface that mediates cooperative tetramerization. Combined, this study provides critical insights into the structural architecture of SPARTA and the molecular mechanism underlying target DNA-dependent oligomerization and catalytic activation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18486.map.gz emd_18486.map.gz | 89 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18486-v30.xml emd-18486-v30.xml emd-18486.xml emd-18486.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_18486.png emd_18486.png | 49 KB | ||
| Filedesc metadata |  emd-18486.cif.gz emd-18486.cif.gz | 5.9 KB | ||
| その他 |  emd_18486_half_map_1.map.gz emd_18486_half_map_1.map.gz emd_18486_half_map_2.map.gz emd_18486_half_map_2.map.gz | 165.1 MB 165.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18486 http://ftp.pdbj.org/pub/emdb/structures/EMD-18486 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18486 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18486 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18486_validation.pdf.gz emd_18486_validation.pdf.gz | 810.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18486_full_validation.pdf.gz emd_18486_full_validation.pdf.gz | 809.7 KB | 表示 | |
| XML形式データ |  emd_18486_validation.xml.gz emd_18486_validation.xml.gz | 14.9 KB | 表示 | |
| CIF形式データ |  emd_18486_validation.cif.gz emd_18486_validation.cif.gz | 17.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18486 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18486 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18486 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18486 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18486.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18486.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.65 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_18486_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
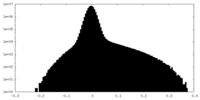
| 投影像・断面図 |
| ||||||||||||
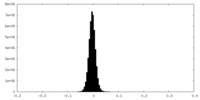
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18486_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
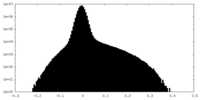
| 投影像・断面図 |
| ||||||||||||
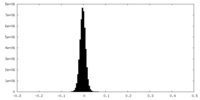
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of BabAgo with BabTIR-APAZ
| 全体 | 名称: Complex of BabAgo with BabTIR-APAZ |
|---|---|
| 要素 |
|
-超分子 #1: Complex of BabAgo with BabTIR-APAZ
| 超分子 | 名称: Complex of BabAgo with BabTIR-APAZ / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Bacillales bacterium (バクテリア) Bacillales bacterium (バクテリア) |
-分子 #1: Short prokaryotic Argonaute
| 分子 | 名称: Short prokaryotic Argonaute / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Bacillales bacterium (バクテリア) Bacillales bacterium (バクテリア) |
| 分子量 | 理論値: 57.911199 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKELIYIHEP NILFANGQKC ADPRDGLALF GPFTKIYGIK SGVVGTQYGL SIFKNYINHI QKPIYNANNI TRPMFPGFEA VFGCKWDAD NVVFKEVTKE EIEKILYTES NHKRTYDLVS LFINKIITAN KNEDEKVDVW FLVIPDEIYQ YCRPNSVLPK D LVQTKSLI ...文字列: MKELIYIHEP NILFANGQKC ADPRDGLALF GPFTKIYGIK SGVVGTQYGL SIFKNYINHI QKPIYNANNI TRPMFPGFEA VFGCKWDAD NVVFKEVTKE EIEKILYTES NHKRTYDLVS LFINKIITAN KNEDEKVDVW FLVIPDEIYQ YCRPNSVLPK D LVQTKSLI TKSKAKSFRY EPTLFENINK ELKEQEKEAI TYNYDAQFHD QLKARLLEHT IPTQILREST LAWRDFKNKF GA PKRDFSK IEGHLAWTIS TAAFYKAGGK PWKLSDIRSG VCYLGLVYKQ IEKSSNPKNA CCAAQMFLDN GDGTVFKGEV GPW YNQEKH EFHLNPKEAK ALLTQALNSY KEQNGVFPKE IFIHAKTKFN GQEWNAFQEV TPEGTNLVGV TITKTKPLKL FKSE GNYPI MRGNAFIVNE RSAFLWTVGY VPKTESTLSM EVPNPIFIEI NKGEADIEQV LKDVLALTKL NYNACIYADG VPVTL RFAD KIGEILTAST ELKAPPLAFK YYI |
-分子 #2: Toll/interleukin-1 receptor domain-containing protein
| 分子 | 名称: Toll/interleukin-1 receptor domain-containing protein タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Bacillales bacterium (バクテリア) Bacillales bacterium (バクテリア) |
| 分子量 | 理論値: 53.052355 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNARNKIFIS HAAPDDNDFT KWLALKLIAL GYEVWCDVLF LDKGADFWKV IDKEIREGAI KFLLATSEIA IKRDGVLKEI AVAEKVKKQ LKDDNFIIPL IIDENLSYDD LPPEIIRLNA VDFKKSWAVG LQDLLKALDD QKVEKNSPDP DKSNALYQQI F LHNKGIIE ...文字列: SNARNKIFIS HAAPDDNDFT KWLALKLIAL GYEVWCDVLF LDKGADFWKV IDKEIREGAI KFLLATSEIA IKRDGVLKEI AVAEKVKKQ LKDDNFIIPL IIDENLSYDD LPPEIIRLNA VDFKKSWAVG LQDLLKALDD QKVEKNSPDP DKSNALYQQI F LHNKGIIE REEIYDSNWF SILSFPKELR FHDYEKLMPK GFDVRELTYP AVRYKNYLCT FAWEYDFMHQ LPKTETYNSS QT IRIPTEE ILSGKYDSPF IGNFECQRLI VQLLNKAFEL RMKEKGVREY PMSNKMGYWF EKGKLEKDKF NKVLLVGKQK DKH WHFGIS AAGKLYPFPV LMISSHIFFT KDGKELIESK KIQHAARRRQ GKNWWNDDWR NKLLAFVKYL SDDENSFYLE VGSE EKIYI SNEPVQFVGK VSYNMPEKNN LKDEAEISDL NDLNEFDGEI FEETDSE |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 58.005 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL In silico モデル: Model generated with AlphaFold2 ColabFold |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.57 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 904721 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X