+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | S. cerevisia Niemann-Pick type C protein NCR1 in GDN at pH 7.5 | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | sterol transport / vacuole / lysosome / LIPID TRANSPORT / membrane protein | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Intestinal lipid absorption / LDL clearance / sterol transport / sterol binding / sphingolipid metabolic process / fungal-type vacuole membrane / cholesterol binding / cholesterol homeostasis / endoplasmic reticulum / membrane / plasma membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||
 データ登録者 データ登録者 | Frain KM / Dedic E / Nel L / Olesen E / Stokes D / Panyella Pedersen B | ||||||||||||
| 資金援助 |  米国, 米国,  デンマーク, 3件 デンマーク, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2024 ジャーナル: Proc Natl Acad Sci U S A / 年: 2024タイトル: Conformational changes in the Niemann-Pick type C1 protein NCR1 drive sterol translocation. 著者: Kelly M Frain / Emil Dedic / Lynette Nel / Anastasiia Bohush / Esben Olesen / Katja Thaysen / Daniel Wüstner / David L Stokes / Bjørn Panyella Pedersen /   要旨: The membrane protein Niemann-Pick type C1 (NPC1, named NCR1 in yeast) is central to sterol homeostasis in eukaryotes. NCR1 is localized to the vacuolar membrane, where it is suggested to carry ...The membrane protein Niemann-Pick type C1 (NPC1, named NCR1 in yeast) is central to sterol homeostasis in eukaryotes. NCR1 is localized to the vacuolar membrane, where it is suggested to carry sterols across the protective glycocalyx and deposit them into the vacuolar membrane. However, documentation of a vacuolar glycocalyx in fungi is lacking, and the mechanism for sterol translocation has remained unclear. Here, we provide evidence supporting the presence of a glycocalyx in isolated vacuoles and report four cryo-EM structures of NCR1 in two distinct conformations, named tense and relaxed. These two conformations illustrate the movement of sterols through a tunnel formed by the luminal domains, thus bypassing the barrier presented by the glycocalyx. Based on these structures and on comparison with other members of the Resistance-Nodulation-Division (RND) superfamily, we propose a transport model that links changes in the luminal domains with a cycle of protonation and deprotonation within the transmembrane region of the protein. Our model suggests that NPC proteins work by a generalized RND mechanism where the proton motive force drives conformational changes in the transmembrane domains that are allosterically coupled to luminal/extracellular domains to promote sterol transport. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18350.map.gz emd_18350.map.gz | 38.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18350-v30.xml emd-18350-v30.xml emd-18350.xml emd-18350.xml | 22.1 KB 22.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18350_fsc.xml emd_18350_fsc.xml | 8.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18350.png emd_18350.png | 22.3 KB | ||
| マスクデータ |  emd_18350_msk_1.map emd_18350_msk_1.map | 40.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18350.cif.gz emd-18350.cif.gz | 7.6 KB | ||
| その他 |  emd_18350_half_map_1.map.gz emd_18350_half_map_1.map.gz emd_18350_half_map_2.map.gz emd_18350_half_map_2.map.gz | 37.7 MB 37.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18350 http://ftp.pdbj.org/pub/emdb/structures/EMD-18350 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18350 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18350 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18350_validation.pdf.gz emd_18350_validation.pdf.gz | 832 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18350_full_validation.pdf.gz emd_18350_full_validation.pdf.gz | 831.6 KB | 表示 | |
| XML形式データ |  emd_18350_validation.xml.gz emd_18350_validation.xml.gz | 14.8 KB | 表示 | |
| CIF形式データ |  emd_18350_validation.cif.gz emd_18350_validation.cif.gz | 18.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18350 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18350 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18350 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18350 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18350.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18350.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.294 Å | ||||||||||||||||||||
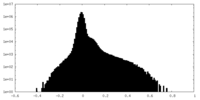
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18350_msk_1.map emd_18350_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
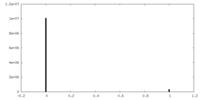
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18350_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
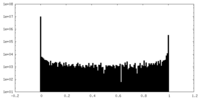
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18350_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
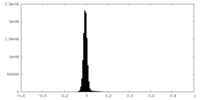
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : NCR1 pH 7.5 in GDN
| 全体 | 名称: NCR1 pH 7.5 in GDN |
|---|---|
| 要素 |
|
-超分子 #1: NCR1 pH 7.5 in GDN
| 超分子 | 名称: NCR1 pH 7.5 in GDN / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 132.6 KDa |
-分子 #1: NPC intracellular sterol transporter 1-related protein 1
| 分子 | 名称: NPC intracellular sterol transporter 1-related protein 1 タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 132.755094 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNVLWIIALV GQLMRLVQGT ATCAMYGNCG KKSVFGNELP CPVPRSFEPP VLSDETSKLL VEVCGEEWKE VRYACCTKDQ VVALRDNLQ KAQPLISSCP ACLKNFNNLF CHFTCAADQG RFVNITKVEK SKEDKDIVAE LDVFMNSSWA SEFYDSCKNI K FSATNGYA ...文字列: MNVLWIIALV GQLMRLVQGT ATCAMYGNCG KKSVFGNELP CPVPRSFEPP VLSDETSKLL VEVCGEEWKE VRYACCTKDQ VVALRDNLQ KAQPLISSCP ACLKNFNNLF CHFTCAADQG RFVNITKVEK SKEDKDIVAE LDVFMNSSWA SEFYDSCKNI K FSATNGYA MDLIGGGAKN YSQFLKFLGD AKPMLGGSPF QINYKYDLAN EEKEWQEFND EVYACDDAQY KCACSDCQES CP HLKPLKD GVCKVGPLPC FSLSVLIFYT ICALFAFMWY YLCKRKKNGA MIVDDDIVPE SGSLDESETN VFESFNNETN FFN GKLANL FTKVGQFSVE NPYKILITTV FSIFVFSFII FQYATLETDP INLWVSKNSE KFKEKEYFDD NFGPFYRTEQ IFVV NETGP VLSYETLHWW FDVENFITEE LQSSENIGYQ DLCFRPTEDS TCVIESFTQY FQGALPNKDS WKRELQECGK FPVNC LPTF QQPLKTNLLF SDDDILNAHA FVVTLLLTNH TQSANRWEER LEEYLLDLKV PEGLRISFNT EISLEKELNN NNDIST VAI SYLMMFLYAT WALRRKDGKT RLLLGISGLL IVLASIVCAA GFLTLFGLKS TLIIAEVIPF LILAIGIDNI FLITHEY DR NCEQKPEYSI DQKIISAIGR MSPSILMSLL CQTGCFLIAA FVTMPAVHNF AIYSTVSVIF NGVLQLTAYV SILSLYEK R SNYKQITGNE ETKESFLKTF YFKMLTQKRL IIIIFSAWFF TSLVFLPEIQ FGLDQTLAVP QDSYLVDYFK DVYSFLNVG PPVYMVVKNL DLTKRQNQQK ICGKFTTCER DSLANVLEQE RHRSTITEPL ANWLDDYFMF LNPQNDQCCR LKKGTDEVCP PSFPSRRCE TCFQQGSWNY NMSGFPEGKD FMEYLSIWIN APSDPCPLGG RAPYSTALVY NETSVSASVF RTAHHPLRSQ K DFIQAYSD GVRISSSFPE LDMFAYSPFY IFFVQYQTLG PLTLKLIGSA IILIFFISSV FLQNIRSSFL LALVVTMIIV DI GALMALL GISLNAVSLV NLIICVGLGV EFCVHIVRSF TVVPSETKKD ANSRVLYSLN TIGESVIKGI TLTKFIGVCV LAF AQSKIF DVFYFRMWFT LIIVAALHAL LFLPALLSLF GGESYRDDSI EAED UniProtKB: NPC intracellular sterol transporter 1-related protein 1 |
-分子 #3: CHOLESTEROL HEMISUCCINATE
| 分子 | 名称: CHOLESTEROL HEMISUCCINATE / タイプ: ligand / ID: 3 / コピー数: 2 / 式: Y01 |
|---|---|
| 分子量 | 理論値: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-分子 #4: ERGOSTEROL
| 分子 | 名称: ERGOSTEROL / タイプ: ligand / ID: 4 / コピー数: 1 / 式: ERG |
|---|---|
| 分子量 | 理論値: 396.648 Da |
| Chemical component information |  ChemComp-ERG: |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 1 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 9 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. | ||||||||||||
| 凍結 | 凍結剤: NITROGEN / チャンバー内湿度: 99 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5790 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 2 / 実像数: 15141 / 平均電子線量: 59.558 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X