+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 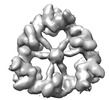 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
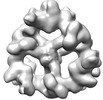
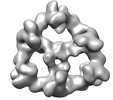
| タイトル | Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 CPH domain | |||||||||
 マップデータ マップデータ | postprocess map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cullin-RING RBR E3 Ligase / LIGASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cullin-RING-type E3 NEDD8 transferase / cellular response to chemical stress / NEDD8 transferase activity / cullin-RING ubiquitin ligase complex / Cul7-RING ubiquitin ligase complex / ubiquitin-dependent protein catabolic process via the C-end degron rule pathway / Loss of Function of FBXW7 in Cancer and NOTCH1 Signaling / positive regulation of protein autoubiquitination / protein neddylation / NEDD8 ligase activity ...cullin-RING-type E3 NEDD8 transferase / cellular response to chemical stress / NEDD8 transferase activity / cullin-RING ubiquitin ligase complex / Cul7-RING ubiquitin ligase complex / ubiquitin-dependent protein catabolic process via the C-end degron rule pathway / Loss of Function of FBXW7 in Cancer and NOTCH1 Signaling / positive regulation of protein autoubiquitination / protein neddylation / NEDD8 ligase activity / Cul5-RING ubiquitin ligase complex / negative regulation of response to oxidative stress / ubiquitin-ubiquitin ligase activity / Cul4A-RING E3 ubiquitin ligase complex / SCF ubiquitin ligase complex / Cul2-RING ubiquitin ligase complex / negative regulation of type I interferon production / Cul4B-RING E3 ubiquitin ligase complex / regulation of mitotic nuclear division / SCF-dependent proteasomal ubiquitin-dependent protein catabolic process / Cul3-RING ubiquitin ligase complex / Prolactin receptor signaling / protein monoubiquitination / cullin family protein binding / protein K48-linked ubiquitination / Nuclear events stimulated by ALK signaling in cancer / positive regulation of TORC1 signaling / T cell activation / Regulation of BACH1 activity / post-translational protein modification / Degradation of DVL / Recognition of DNA damage by PCNA-containing replication complex / Degradation of GLI1 by the proteasome / Negative regulation of NOTCH4 signaling / cellular response to amino acid stimulus / GSK3B and BTRC:CUL1-mediated-degradation of NFE2L2 / Vif-mediated degradation of APOBEC3G / Hedgehog 'on' state / DNA Damage Recognition in GG-NER / Degradation of GLI2 by the proteasome / GLI3 is processed to GLI3R by the proteasome / FBXL7 down-regulates AURKA during mitotic entry and in early mitosis / RING-type E3 ubiquitin transferase / Degradation of beta-catenin by the destruction complex / negative regulation of canonical Wnt signaling pathway / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / Dual Incision in GG-NER / Evasion by RSV of host interferon responses / NOTCH1 Intracellular Domain Regulates Transcription / Transcription-Coupled Nucleotide Excision Repair (TC-NER) / Formation of TC-NER Pre-Incision Complex / Regulation of expression of SLITs and ROBOs / Constitutive Signaling by NOTCH1 PEST Domain Mutants / Constitutive Signaling by NOTCH1 HD+PEST Domain Mutants / Formation of Incision Complex in GG-NER / microtubule cytoskeleton organization / Interleukin-1 signaling / Orc1 removal from chromatin / Dual incision in TC-NER / Gap-filling DNA repair synthesis and ligation in TC-NER / Regulation of RAS by GAPs / protein polyubiquitination / positive regulation of protein catabolic process / Regulation of RUNX2 expression and activity / cellular response to UV / KEAP1-NFE2L2 pathway / MAPK cascade / ubiquitin protein ligase activity / Antigen processing: Ubiquitination & Proteasome degradation / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / Neddylation / ubiquitin-dependent protein catabolic process / spermatogenesis / proteasome-mediated ubiquitin-dependent protein catabolic process / positive regulation of canonical NF-kappaB signal transduction / RNA polymerase II-specific DNA-binding transcription factor binding / Potential therapeutics for SARS / molecular adaptor activity / protein ubiquitination / DNA repair / ubiquitin protein ligase binding / DNA damage response / zinc ion binding / nucleoplasm / ATP binding / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
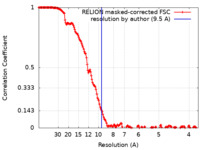
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.5 Å | |||||||||
 データ登録者 データ登録者 | Hopf LVM / Horn-Ghetko D / Schulman BA | |||||||||
| 資金援助 | European Union,  ドイツ, 2件 ドイツ, 2件
| |||||||||
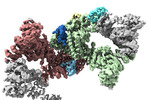
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Noncanonical assembly, neddylation and chimeric cullin-RING/RBR ubiquitylation by the 1.8 MDa CUL9 E3 ligase complex. 著者: Daniel Horn-Ghetko / Linus V M Hopf / Ishita Tripathi-Giesgen / Jiale Du / Sebastian Kostrhon / D Tung Vu / Viola Beier / Barbara Steigenberger / J Rajan Prabu / Luca Stier / Elias M Bruss / ...著者: Daniel Horn-Ghetko / Linus V M Hopf / Ishita Tripathi-Giesgen / Jiale Du / Sebastian Kostrhon / D Tung Vu / Viola Beier / Barbara Steigenberger / J Rajan Prabu / Luca Stier / Elias M Bruss / Matthias Mann / Yue Xiong / Brenda A Schulman /   要旨: Ubiquitin ligation is typically executed by hallmark E3 catalytic domains. Two such domains, 'cullin-RING' and 'RBR', are individually found in several hundred human E3 ligases, and collaborate with ...Ubiquitin ligation is typically executed by hallmark E3 catalytic domains. Two such domains, 'cullin-RING' and 'RBR', are individually found in several hundred human E3 ligases, and collaborate with E2 enzymes to catalyze ubiquitylation. However, the vertebrate-specific CUL9 complex with RBX1 (also called ROC1), of interest due to its tumor suppressive interaction with TP53, uniquely encompasses both cullin-RING and RBR domains. Here, cryo-EM, biochemistry and cellular assays elucidate a 1.8-MDa hexameric human CUL9-RBX1 assembly. Within one dimeric subcomplex, an E2-bound RBR domain is activated by neddylation of its own cullin domain and positioning from the adjacent CUL9-RBX1 in trans. Our data show CUL9 as unique among RBX1-bound cullins in dependence on the metazoan-specific UBE2F neddylation enzyme, while the RBR domain protects it from deneddylation. Substrates are recruited to various upstream domains, while ubiquitylation relies on both CUL9's neddylated cullin and RBR domains achieving self-assembled and chimeric cullin-RING/RBR E3 ligase activity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
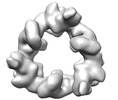
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18220.map.gz emd_18220.map.gz | 356.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18220-v30.xml emd-18220-v30.xml emd-18220.xml emd-18220.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18220_fsc.xml emd_18220_fsc.xml | 16.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18220.png emd_18220.png | 75.3 KB | ||
| Filedesc metadata |  emd-18220.cif.gz emd-18220.cif.gz | 6.5 KB | ||
| その他 |  emd_18220_additional_1.map.gz emd_18220_additional_1.map.gz emd_18220_half_map_1.map.gz emd_18220_half_map_1.map.gz emd_18220_half_map_2.map.gz emd_18220_half_map_2.map.gz | 304.4 MB 305.2 MB 305 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18220 http://ftp.pdbj.org/pub/emdb/structures/EMD-18220 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18220 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18220 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18220_validation.pdf.gz emd_18220_validation.pdf.gz | 787.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18220_full_validation.pdf.gz emd_18220_full_validation.pdf.gz | 786.8 KB | 表示 | |
| XML形式データ |  emd_18220_validation.xml.gz emd_18220_validation.xml.gz | 24 KB | 表示 | |
| CIF形式データ |  emd_18220_validation.cif.gz emd_18220_validation.cif.gz | 32.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18220 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18220 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18220 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18220 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8q7eC  8q7hC  8rhzC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18220.map.gz / 形式: CCP4 / 大きさ: 381.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18220.map.gz / 形式: CCP4 / 大きさ: 381.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | postprocess map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.885 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: refinement map
| ファイル | emd_18220_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | refinement map | ||||||||||||
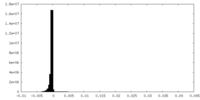
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18220_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
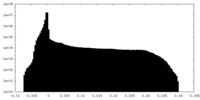
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18220_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
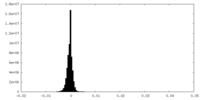
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Structure of the hexameric CUL9-RBX1 complex with deletion of CUL...
| 全体 | 名称: Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 CPH domain |
|---|---|
| 要素 |
|
-超分子 #1: Structure of the hexameric CUL9-RBX1 complex with deletion of CUL...
| 超分子 | 名称: Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 CPH domain タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
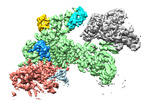
-分子 #1: Cullin-9
| 分子 | 名称: Cullin-9 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MVGERHAGDL MVPLGPRLQA YPEELIRQRP GHDGHPEYLI RWSVLKCGEV GKVGVEEGKA EHILMWLSAP EVYANCPGLL GERALSKGLQ HEPAGVSGSF PRDPGGLDEV AMGEMEADVQ ALVRRAARQL AESGTPSLTA AVLHTIHVLS AYASIGPLTG VFRETGALDL ...文字列: MVGERHAGDL MVPLGPRLQA YPEELIRQRP GHDGHPEYLI RWSVLKCGEV GKVGVEEGKA EHILMWLSAP EVYANCPGLL GERALSKGLQ HEPAGVSGSF PRDPGGLDEV AMGEMEADVQ ALVRRAARQL AESGTPSLTA AVLHTIHVLS AYASIGPLTG VFRETGALDL LMHMLCNPEP QIRRSAGKML QALAAHDAGS RAHVLLSLSQ QDGIEQHMDF DSRYTLLELF AETTSSEEHC MAFEGIHLPQ IPGKLLFSLV KRYLCVTSLL DQLNSSPELG AGDQSSPCAT REKSRGQREL EFSMAVGNLI SELVRSMGWA RNLSEQGMSP PRPTRSIFQP YISGPSLLLP TIVGSGSGSG SGTAFPSWDW NPMDGLYPLP YLQPEPQKNE RVGYLTQAEW WELLFFIKKL DLCEQQPIFQ NLWKNLDETL GEKALGEISV SVEMAESLLQ VLSSRFEGST LNDLLNSQIY TKYGLLSNEP SSSSTSRNHS CTPDPEEESK SEASFSEEET ESLKAKAEAP KTEAEPTKTR TETPMAQSDS QLFNQLLVTE GMTLPTEMKE AASEMARALR GPGPRSSLDQ HVAAVVATVQ ISSLDTNLQL SGLSALSQAV EEVTERDHPL VRPDRSLREK LVKMLVELLT NQVGEKMVVV QALRLLYLLM TKHEWRPLFA REGGIYAVLV CMQEYKTSVL VQQAGLAALK MLAVASSSEI PTFVTGRDSI HSLFDAQMTR EIFASIDSAT RPGSESLLLT VPAAVILMLN TEGCSSAARN GLLLLNLLLC NHHTLGDQII TQELRDTLFR HSGIAPRTEP MPTTRTILMM LLNRYSEPPG SPERAALETP IIQGQDGSPE LLIRSLVGGP SAELLLDLER VLCREGSPGG AVRPLLKRLQ QETQPFLLLL RTLDAPGPNK TLLLSVLRVI TRLLDFPEAM VLPWHEVLEP CLNCLSGPSS DSEIVQELTC FLHRLASMHK DYAVVLCCLG AKEILSKVLD KHSAQLLLGC ELRDLVTECE KYAQLYSNLT SSILAGSIQM VLGQIEDHRR THQPINIPFF DVFLRHLCQG SSVEVKEDKC WEKVEVSSNP HRASKLTDHN PKTYWESNGS TGSHYITLHM HRGVLVRQLT LLVASEDSSY MPARVVVFGG DSTSCIGTEL NTVNVMPSAS RVILLENLNR FWPIIQIRIK RCQQGGIDTR VRGVEVLGPK PTFWPLFREQ LCRRTCLFYT IRAQAWSRDI AEDHRRLLQL CPRLNRVLRH EQNFADRFLP DDEAAQALGK TCWEALVSPL VQNITSPDAE GVSALGWLLD QYLEQRETSR NPLSRAASFA SRVRRLCHLL VHVEPPPGPS PEPSTRPFSK NSKGRDRSPA PSPVLPSSSL RNITQCWLSV VQEQVSRFLA AAWRAPDFVP RYCKLYEHLQ RAGSELFGPR AAFMLALRSG FSGALLQQSF LTAAHMSEQF ARYIDQQIQG GLIGGAPGVE MLGQLQRHLE PIMVLSGLEL ATTFEHFYQH YMADRLLSFG SSWLEGAVLE QIGLCFPNRL PQLMLQSLST SEELQRQFHL FQLQRLDKLF LEQEDEEEKR LEEEEEEEEE EEAEKELFIE DPSPAISILV LSPRCWPVSP LCYLYHPRKC LPTEFCDALD RFSSFYSQSQ NHPVLDMGPH RRLQWTWLGR AELQFGKQIL HVSTVQMWLL LKFNQTEEVS VETLLKDSDL SPELLLQALV PLTSGNGPLT LHEGQDFPHG GVLRLHEPGP QRSGEALWLI PPQAYLNVEK DEGRTLEQKR NLLSCLLVRI LKAHGEKGLH IDQLVCLVLE AWQKGPNPPG TLGHTVAGGV ACTSTDVLSC ILHLLGQGYV KRRDDRPQIL MYAAPEPMGP CRGQADVPFC GSQSETSKPS PEAVATLASL QLPAGRTMSP QEVEGLMKQT VRQVQETLNL EPDVAQHLLA HSHWGAEQLL QSYSEDPEPL LLAAGLCVHQ AQAVPVRPDH CPVCVSPLGC DDDLPSLCCM HYCCKSCWNE YLTTRIEQNL VLNCTCPIAD CPAQPTGAFI RAIVSSPEVI SKYEKALLRG YVESCSNLTW CTNPQGCDRI LCRQGLGCGT TCSKCGWASC FNCSFPEAHY PASCGHMSQW VDDGGYYDGM SVEAQSKHLA KLISKRCPSC QAPIEKNEGC LHMTCAKCNH GFCWRCLKSW KPNHKDYYNC SAMVSKAARQ EKRFQDYNER CTFHHQAREF AVNLRNRVSA IHEVPPPRSF TFLNDACQGL EQARKVLAYA CVYSFYSQDA EYMDVVEQQT ENLELHTNAL QILLEETLLR CRDLASSLRL LRADCLSTGM ELLRRIQERL LAILQHSAQD FRVGLQSPSV EAWEAKGPNM PGSQPQASSG PEAEEEEEDD EDDVPEWQQD EFDEELDNDS FSYDESENLD QETFFFGDEE EDEDEAYD UniProtKB: Cullin-9 |
-分子 #2: E3 ubiquitin-protein ligase RBX1
| 分子 | 名称: E3 ubiquitin-protein ligase RBX1 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDVDTPSGTN SGAGKKRFEV KKWNAVALWA WDIVVDNCAI CRNHIMDLCI ECQANQ ASA TSEECTVAWG VCNHAFHFHC ISRWLKTRQV CPLDNREWEF QKYGH UniProtKB: E3 ubiquitin-protein ligase RBX1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 3.3000000000000003 µm 最小 デフォーカス(公称値): 1.2 µm |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X