| 登録情報 | データベース: EMDB / ID: EMD-16892
|
|---|
| タイトル | Cryo-EM KSB domain of RhiE from Burkholderia rhizoxinica |
|---|
 マップデータ マップデータ | |
|---|
 試料 試料 | - 複合体: Homodimeric complex of RhiE KSB domains
- タンパク質・ペプチド: RhiE protein,Polyketide synthase domain protein RhiE
|
|---|
 キーワード キーワード | Polyketide / TRANSFERASE |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
DIM/DIP cell wall layer assembly / fatty acid synthase activity / secondary metabolite biosynthetic process / phosphopantetheine binding / 3-oxoacyl-[acyl-carrier-protein] synthase activity / fatty acid biosynthetic process / plasma membrane / cytoplasm類似検索 - 分子機能 : / : / Polyketide synthase dehydratase domain / : / Polyketide and metazoan fatty acid synthase dehydratase (PKS/mFAS DH) domain profile. / PKS_PP_betabranch / Polyketide synthase dehydratase N-terminal domain / PKS_DH / Polyketide synthase, dehydratase domain / Polyketide synthase, dehydratase domain superfamily ...: / : / Polyketide synthase dehydratase domain / : / Polyketide and metazoan fatty acid synthase dehydratase (PKS/mFAS DH) domain profile. / PKS_PP_betabranch / Polyketide synthase dehydratase N-terminal domain / PKS_DH / Polyketide synthase, dehydratase domain / Polyketide synthase, dehydratase domain superfamily / Polyketide synthase, ketoreductase domain / KR domain / Polyketide synthase, C-terminal extension / Ketoacyl-synthetase C-terminal extension / PKS_KR / Ketosynthase family 3 (KS3) domain profile. / Polyketide synthase, phosphopantetheine-binding domain / Phosphopantetheine attachment site / Beta-ketoacyl synthase / Beta-ketoacyl synthase, active site / Ketosynthase family 3 (KS3) active site signature. / Polyketide synthase, beta-ketoacyl synthase domain / Beta-ketoacyl synthase, N-terminal / Beta-ketoacyl synthase, C-terminal / Beta-ketoacyl synthase, N-terminal domain / Beta-ketoacyl synthase, C-terminal domain / Thiolase-like / Phosphopantetheine attachment site / Phosphopantetheine attachment site. / Phosphopantetheine attachment site / ACP-like superfamily / Carrier protein (CP) domain profile. / Phosphopantetheine binding ACP domain / NAD(P)-binding domain superfamily類似検索 - ドメイン・相同性 RhiE protein / Polyketide synthase domain protein RhiE類似検索 - 構成要素 |
|---|
| 生物種 |  Mycetohabitans rhizoxinica (バクテリア) Mycetohabitans rhizoxinica (バクテリア) |
|---|
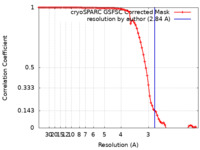
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.84 Å |
|---|
 データ登録者 データ登録者 | Capper MJ / Koehnke J |
|---|
| 資金援助 |  英国, 1件 英国, 1件 | Organization | Grant number | 国 |
|---|
| Biotechnology and Biological Sciences Research Council (BBSRC) | BB/W003090/1 |  英国 英国 |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Structural Analysis of a Chain-Branching Polyketide Synthase Module
著者: Dell M / Tran MA / Capper MJ / Sundaram S / Fiedler J / Koehnke J / Hellmich U / Hertwick C |
|---|
| 履歴 | | 登録 | 2023年3月22日 | - |
|---|
| ヘッダ(付随情報) 公開 | 2024年4月3日 | - |
|---|
| マップ公開 | 2024年4月3日 | - |
|---|
| 更新 | 2024年4月3日 | - |
|---|
| 現状 | 2024年4月3日 | 処理サイト: PDBe / 状態: 公開 |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Mycetohabitans rhizoxinica (バクテリア)
Mycetohabitans rhizoxinica (バクテリア) データ登録者
データ登録者 英国, 1件
英国, 1件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_16892.map.gz
emd_16892.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-16892-v30.xml
emd-16892-v30.xml emd-16892.xml
emd-16892.xml EMDBヘッダ
EMDBヘッダ emd_16892_fsc.xml
emd_16892_fsc.xml FSCデータファイル
FSCデータファイル emd_16892.png
emd_16892.png emd_16892_msk_1.map
emd_16892_msk_1.map マスクマップ
マスクマップ emd-16892.cif.gz
emd-16892.cif.gz emd_16892_additional_1.map.gz
emd_16892_additional_1.map.gz emd_16892_half_map_1.map.gz
emd_16892_half_map_1.map.gz emd_16892_half_map_2.map.gz
emd_16892_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-16892
http://ftp.pdbj.org/pub/emdb/structures/EMD-16892 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16892
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16892 emd_16892_validation.pdf.gz
emd_16892_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_16892_full_validation.pdf.gz
emd_16892_full_validation.pdf.gz emd_16892_validation.xml.gz
emd_16892_validation.xml.gz emd_16892_validation.cif.gz
emd_16892_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16892
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16892 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16892
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16892
 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_16892.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_16892.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_16892_msk_1.map
emd_16892_msk_1.map 試料の構成要素
試料の構成要素 Mycetohabitans rhizoxinica (バクテリア)
Mycetohabitans rhizoxinica (バクテリア) Mycetohabitans rhizoxinica (バクテリア)
Mycetohabitans rhizoxinica (バクテリア)
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X