+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Extended cowpea chlorotic mottle virus | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Icosahedral / extended / virus | |||||||||
| 機能・相同性 | Bromovirus coat protein / Bromovirus coat protein / T=3 icosahedral viral capsid / viral nucleocapsid / ribonucleoprotein complex / structural molecule activity / RNA binding / Capsid protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Cowpea chlorotic mottle virus (ウイルス) Cowpea chlorotic mottle virus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Harder OF / Barrass SV / Drabbels M / Lorenz UJ | |||||||||
| 資金援助 |  スイス, European Union, 2件 スイス, European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Fast viral dynamics revealed by microsecond time-resolved cryo-EM. 著者: Oliver F Harder / Sarah V Barrass / Marcel Drabbels / Ulrich J Lorenz /  要旨: Observing proteins as they perform their tasks has largely remained elusive, which has left our understanding of protein function fundamentally incomplete. To enable such observations, we have ...Observing proteins as they perform their tasks has largely remained elusive, which has left our understanding of protein function fundamentally incomplete. To enable such observations, we have recently proposed a technique that improves the time resolution of cryo-electron microscopy (cryo-EM) to microseconds. Here, we demonstrate that microsecond time-resolved cryo-EM enables observations of fast protein dynamics. We use our approach to elucidate the mechanics of the capsid of cowpea chlorotic mottle virus (CCMV), whose large-amplitude motions play a crucial role in the viral life cycle. We observe that a pH jump causes the extended configuration of the capsid to contract on the microsecond timescale. While this is a concerted process, the motions of the capsid proteins involve different timescales, leading to a curved reaction path. It is difficult to conceive how such a detailed picture of the dynamics could have been obtained with any other method, which highlights the potential of our technique. Crucially, our experiments pave the way for microsecond time-resolved cryo-EM to be applied to a broad range of protein dynamics that previously could not have been observed. This promises to fundamentally advance our understanding of protein function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16790.map.gz emd_16790.map.gz | 202.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16790-v30.xml emd-16790-v30.xml emd-16790.xml emd-16790.xml | 15.2 KB 15.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16790.png emd_16790.png | 243.8 KB | ||
| その他 |  emd_16790_half_map_1.map.gz emd_16790_half_map_1.map.gz emd_16790_half_map_2.map.gz emd_16790_half_map_2.map.gz | 199.3 MB 199.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16790 http://ftp.pdbj.org/pub/emdb/structures/EMD-16790 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16790 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16790 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16790_validation.pdf.gz emd_16790_validation.pdf.gz | 1022.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16790_full_validation.pdf.gz emd_16790_full_validation.pdf.gz | 1021.7 KB | 表示 | |
| XML形式データ |  emd_16790_validation.xml.gz emd_16790_validation.xml.gz | 15.7 KB | 表示 | |
| CIF形式データ |  emd_16790_validation.cif.gz emd_16790_validation.cif.gz | 18.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16790 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16790 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16790 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16790 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8cpyMC  8c38C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16790.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16790.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.452 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_16790_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
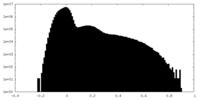
| 投影像・断面図 |
| ||||||||||||
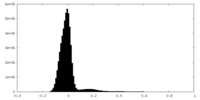
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16790_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
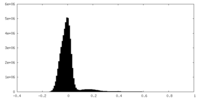
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cowpea chlorotic mottle virus
| 全体 | 名称:  Cowpea chlorotic mottle virus (ウイルス) Cowpea chlorotic mottle virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Cowpea chlorotic mottle virus
| 超分子 | 名称: Cowpea chlorotic mottle virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 12303 / 生物種: Cowpea chlorotic mottle virus / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| ウイルス殻 | Shell ID: 1 / T番号(三角分割数): 3 |
-分子 #1: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Cowpea chlorotic mottle virus (ウイルス) Cowpea chlorotic mottle virus (ウイルス) |
| 分子量 | 理論値: 20.366277 KDa |
| 配列 | 文字列: MSTVGTGKLT RAQRRAAARK NKRNTRVVQP VIVEPIASGQ GKAIKAWTGY SVSKWTASCA AAEAKVTSAI TISLPNELSS ERNKQLKVG RVLLWLGLLP SVSGTVKSCV TETQTTAAAS FQVALAVADN SKDVVAAMYP EAFKGITLEQ LTADLTIYLY S SAALTEGD ...文字列: MSTVGTGKLT RAQRRAAARK NKRNTRVVQP VIVEPIASGQ GKAIKAWTGY SVSKWTASCA AAEAKVTSAI TISLPNELSS ERNKQLKVG RVLLWLGLLP SVSGTVKSCV TETQTTAAAS FQVALAVADN SKDVVAAMYP EAFKGITLEQ LTADLTIYLY S SAALTEGD VIVHLEVEHV RPTFDDSFTP VY UniProtKB: Capsid protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 4.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
| 詳細 | Extended CCMV was prepared at pH 7.6 and incubated with NPE-caged-proton in a 6:1 ratio prior to plunge freezing. The grid was then irritated in a Linkam Cryo-stage with a UV laser, activating the NPE-caged-proton and reducing the sample pH to 4.5. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| アライメント法 | Coma free - Residual tilt: 150.0 mrad |
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 0.726 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.9 µm / 最小 デフォーカス(公称値): 0.3 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER |
| 得られたモデル |  PDB-8cpy: |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X