+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the RESC1-RESC2 complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RESC / RNA editing / cryo-EM structure / Trypanosoma brucei / RNA BINDING PROTEIN | |||||||||
| 機能・相同性 | mitochondrial mRNA processing / mitochondrial mRNA editing complex / mitochondrial RNA processing / mRNA modification / kinetoplast / mRNA binding / mitochondrion / Guide RNA associated protein, GAP2 / Uncharacterized protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
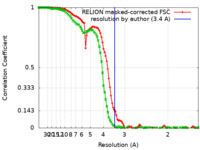
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Dolce LG / Weis F / Kowalinski E | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2023 ジャーナル: Nucleic Acids Res / 年: 2023タイトル: Structural basis for guide RNA selection by the RESC1-RESC2 complex. 著者: Luciano G Dolce / Yevheniia Nesterenko / Leon Walther / Félix Weis / Eva Kowalinski /   要旨: Kinetoplastid parasites, such as trypanosomes or leishmania, rely on RNA-templated RNA editing to mature mitochondrial cryptic pre-mRNAs into functional protein-coding transcripts. Processive pan- ...Kinetoplastid parasites, such as trypanosomes or leishmania, rely on RNA-templated RNA editing to mature mitochondrial cryptic pre-mRNAs into functional protein-coding transcripts. Processive pan-editing of multiple editing blocks within a single transcript is dependent on the 20-subunit RNA editing substrate binding complex (RESC) that serves as a platform to orchestrate the interactions between pre-mRNA, guide RNAs (gRNAs), the catalytic RNA editing complex (RECC), and a set of RNA helicases. Due to the lack of molecular structures and biochemical studies with purified components, neither the spacio-temporal interplay of these factors nor the selection mechanism for the different RNA components is understood. Here we report the cryo-EM structure of Trypanosoma brucei RESC1-RESC2, a central hub module of the RESC complex. The structure reveals that RESC1 and RESC2 form an obligatory domain-swapped dimer. Although the tertiary structures of both subunits closely resemble each other, only RESC2 selectively binds 5'-triphosphate-nucleosides, a defining characteristic of gRNAs. We therefore propose RESC2 as the protective 5'-end binding site for gRNAs within the RESC complex. Overall, our structure provides a starting point for the study of the assembly and function of larger RNA-bound kinetoplast RNA editing modules and might aid in the design of anti-parasite drugs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16592.map.gz emd_16592.map.gz | 80.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16592-v30.xml emd-16592-v30.xml emd-16592.xml emd-16592.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16592_fsc.xml emd_16592_fsc.xml emd_16592_fsc_2.xml emd_16592_fsc_2.xml | 10.7 KB 13.8 KB | 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16592.png emd_16592.png | 90.8 KB | ||
| マスクデータ |  emd_16592_msk_1.map emd_16592_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16592.cif.gz emd-16592.cif.gz | 6 KB | ||
| その他 |  emd_16592_additional_1.map.gz emd_16592_additional_1.map.gz emd_16592_additional_2.map.gz emd_16592_additional_2.map.gz emd_16592_half_map_1.map.gz emd_16592_half_map_1.map.gz emd_16592_half_map_2.map.gz emd_16592_half_map_2.map.gz | 91.3 MB 6 MB 80.8 MB 81 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16592 http://ftp.pdbj.org/pub/emdb/structures/EMD-16592 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16592 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16592 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16592_validation.pdf.gz emd_16592_validation.pdf.gz | 778.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16592_full_validation.pdf.gz emd_16592_full_validation.pdf.gz | 778.1 KB | 表示 | |
| XML形式データ |  emd_16592_validation.xml.gz emd_16592_validation.xml.gz | 17.6 KB | 表示 | |
| CIF形式データ |  emd_16592_validation.cif.gz emd_16592_validation.cif.gz | 23.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16592 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16592 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16592 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16592 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8cdpMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16592.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16592.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.81 Å | ||||||||||||||||||||||||||||||||||||
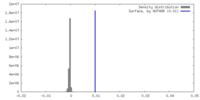
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16592_msk_1.map emd_16592_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
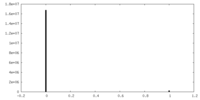
| 密度ヒストグラム |
-追加マップ: deepEMhancer post process
| ファイル | emd_16592_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | deepEMhancer post process | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
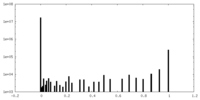
| 密度ヒストグラム |
-追加マップ: relion post process
| ファイル | emd_16592_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | relion post process | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
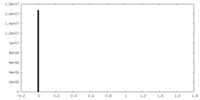
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16592_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16592_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RESC1-RESC2 complex
| 全体 | 名称: RESC1-RESC2 complex |
|---|---|
| 要素 |
|
-超分子 #1: RESC1-RESC2 complex
| 超分子 | 名称: RESC1-RESC2 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Guide_RNA_associated_protein_-_putative
| 分子 | 名称: Guide_RNA_associated_protein_-_putative / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 52.724535 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MKHHHHHHSA GLEVLFQGPD SMQNQGSVSQ GALNMRDQQA AAAENVTPER VWALWNEGNL FSLSLAQLQG FLSRCGVRTD PAAKKAAVV RQVEEYLHSK DTTVKGGGQG AASPQQHQQH GQQGGYGRWN QASVMQPETL LDLSQAGFYE GAANMVPKAF Q LLVSDTAP ...文字列: MKHHHHHHSA GLEVLFQGPD SMQNQGSVSQ GALNMRDQQA AAAENVTPER VWALWNEGNL FSLSLAQLQG FLSRCGVRTD PAAKKAAVV RQVEEYLHSK DTTVKGGGQG AASPQQHQQH GQQGGYGRWN QASVMQPETL LDLSQAGFYE GAANMVPKAF Q LLVSDTAP DVVVSRVNTT AFPGFPSNTE CYTLGASEKD VAIRSRYSKV LQWCCLNMSN LQMDGELYVD FGKLLLKPSV MR KNRRIVS SYTLQQRLQV NHPYTWVPTL PESCLSKIQE QFLQPEGFAP IGKGVQLTYS GTIKRSKDQL HVDLDNKGKV LAV NSAWVN LQTAWCTHAK GPDVRLLLRS RPPIRRQDVE LFASTPIIKL ADDDVADVLP PEHGQLVYLS EDETRLFERV SDRG VTITV REVKRQPLII LRDEEEDPRV EYSLSAHIPA NAAKATDVRA VGLTAFELAG RLAGLVAEDF VREYGCEAKL UniProtKB: Guide RNA associated protein, GAP2 |
-分子 #2: Mitochondrial guide RNA binding complex subunit 2
| 分子 | 名称: Mitochondrial guide RNA binding complex subunit 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 52.210234 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MQSFSAAAPA ASGDFSHITR NTVWGLWNEG NLFSLSVPEL AFFLQEHCRV ANVDPRAKKS ALVRQVEEIL SAEQQASATV PQEDNPHAI VVTDYDRAED ALEEADEYGD WGAEPGFEDR RELDFMELSP GRMGERYDPL SPRAFQLLHS ETATDVGIAS I DPSKLPGQ ...文字列: MQSFSAAAPA ASGDFSHITR NTVWGLWNEG NLFSLSVPEL AFFLQEHCRV ANVDPRAKKS ALVRQVEEIL SAEQQASATV PQEDNPHAI VVTDYDRAED ALEEADEYGD WGAEPGFEDR RELDFMELSP GRMGERYDPL SPRAFQLLHS ETATDVGIAS I DPSKLPGQ SKVKNALAAI HVAPNDANKM RFRMAFEWCL MNIWNMNMPG ELNIGAGKAL YYRSVAKQNR NVMPLWTVQK HL YAQHPYA WFAIASESNV AAMESLAAAL NMSIQQERTT SYKVTIRRMA EFFDCELNGQ LKCTMMNKPW DRFFVSHYIR SKM PDLRYV VRARHPIKKR IADAYLEADI LRSTRDSVQS VLSPELGDVV YCCERVVRKW AKKTATGVTL QLVETKRTPL IITK AGDEG ERLEYEWIVP LPQQAERIDI AALTDELWEY GNKLAAALEE GMEELMVHTM TAVSAY UniProtKB: Uncharacterized protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.07 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 平均電子線量: 63.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)